Arqueas
| Arqueas Rango fósil: Paleoarcaico – Recente | |||
|---|---|---|---|
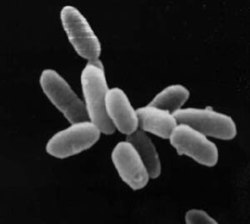 Halobacteria sp. NRC-1; cada célula mide uns 5 μm | |||
| Clasificación científica | |||
| |||
| Reinos e filos | |||
As arqueas (Archaea, do grego ἀρχαῖα 'os antigos') son un grupo de microorganismos unicelulares de morfoloxía procariótica (sen núcleo nin, en xeral, orgánulos membranosos internos), que forman un dos tres grandes dominios dos seres vivos, e que son diferentes das bacterias.
No pasado as arqueas foran clasificadas coas bacterias como procariotas encadradas no antigo reino Monera e recibían o nome de arquebacterias, pero esta clasificación xa non se utiliza.[1] En realidade, as arqueas teñen unha historia evolutiva independente e mostran moitas diferenzas na súa bioquímica coas outras formas de vida, polo que foron clasificadas nun dominio separado dentro do sistema de tres dominios: Archaea, Bacteria e Eukaryota.[2]
As Archaea divídense en cinco filos recoñecidos, pero pénsase que poden haber máis. Destes grupos, os Crenarchaeota e os Euryarchaeota son os máis intensamente estudados. A clasificación das arqueas é aínda difícil, porque a gran maioría nunca foron estudadas no laboratorio e só foron detectadas por análises dos seus ácidos nucleicos en mostras tomadas do ambiente.[3][4]
As arqueas e as bacterias son bastante similares en tamaño e forma, aínda que unhas poucas arqueas teñen formas moi pouco comúns, como as células de forma cadrada aplanadas de Haloquadratum walsbyi[5]. Malia a súa semellanza en aspecto coas bacterias, as arqueas posúen xenes e varias rutas metabólicas que son máis parecidas aos dos eucariotas, como os encimas implicados na transcrición xenética e na tradución de proteínas. Outros aspectos da bioquímica das arqueas son tamén exclusivos, como o feito de presentaren enlaces éter nos seus lípidos da membrana plasmática.[6] As arqueas utilizan unha variedade de fontes de enerxía moito maior cós eucariotas, que van desde compostos orgánicos correntes como azucres, a amoníaco, ións metálicos ou mesmo gas hidróxeno. As arqueas tolerantes ao sal (as haloarqueas) utilizan a luz do sol como fonte de enerxía, e outras especies de arqueas fixan o carbono; pero, a diferenza de plantas e cianobacterias, ningunha especie de arqueas pode facer as dúas cousas. As arqueas reprodúcense asexualmente por fisión binaria, fragmentación ou xemación, pero, a diferenza de bacterias e eucariotas, ningunha forma esporas.[7]
Inicialmente, as arqueas eran consideradas todas extremófilas que vivían en ambientes con duras condicións, como fontes termais e lagos salgados, pero desde entón atopáronse arqueas nos máis diversos hábitats, como o solo, océanos, pantanos e no colon humano.[8] As arqueas son especialmente numerosas nos océanos, e as arqueas do plancto poden ser un dos grupos de seres vivos máis abundantes do planeta. As Archaea son hoxe recoñecidas como unha parte fundamental da vida na Terra, xa que xogan un papel nos ciclos do carbono e do nitróxeno. Non existen exemplos claros de arqueas patóxenas ou parasitas, pero si de mutualistas ou comensais. Un exemplo son os metanóxenos que habitan nos intestinos de humanos e ruminantes; nestes últimos a súa grande abundancia facilita a dixestión. Os metanóxenos utilízanse para a produción de biogás[9] e no tratamento de augas residuais,[10] e certos encimas de arqueas, que poden resistir altas temperaturas e solventes orgánicos, utilízanse en biotecnoloxía.
Historia[editar | editar a fonte]
O grupo de arqueas que primeiro se estudou foi o das metanóxenas. A metanoxénese foi descuberta no lago Maior de Italia en 1776, ao observar nel o burbullar de "aire combustible". En 1882 observouse que a produción de metano no intestino de animais debíase á presenza de microorganismos (Popoff, Tappeiner e Hoppe-Seyler).[11]
En 1936, ano que marcou o principio da era moderna no estudo da metanoxénese, H.A Barker brindou as bases científicas para o estudo da súa fisioloxía e logrou desenvolver un medio de cultivo apropiado para o crecemento dos metanóxenos. Nese ano identificáronse os xéneros Methanococcus e Methanosarcina.[12]
As primeiras arqueas extremófilas encontráronse en ambientes quentes. En 1970, Thomas D. Brock, da Universidade de Wisconsin, descubriu a Thermoplasma, unha arquea termoacidófila e en 1972 a Sulfolobus, unha hipertermófila.[13] Brock iniciárase en 1969 no campo da bioloxía dos hipertermófilos co descubrimento de Thermus aquaticus, que non é unha arquea senón unha bacteria.
En 1977 identifícase ás arqueas como o grupo procariota máis distante ao descubrir que os metanóxenos presentan unha profunda diverxencia con todas as bacterias estudadas. Ese mesmo ano proponse a categoría de superreino para este grupo co nome de Archaebacteria. En 1978, o manual de Bergey dálle a categoría de filo co nome de Mendosicutes e en 1984 divide o reino Procaryotae ou Monera en 4 divisións, agrupando as Archaebacteria na división Mendosicutes.[14]
As arqueas hipertermófilas foron agrupadas en 1984 co nome de Eocyta, identificándoas como un grupo independente das entón chamadas arqueobacterias (en referencia aos metanóxenos) e as eubacterias, descubríndose ademais que Eocyta era o grupo máis achegado aos eucariontes.[15] A relación filoxenética entre metanóxenos e hipertermófilos fai que en 1990 se renomee a Eocyta como Crenarchaeota e ás metanóxenas como Euryarchaeota, formando o novo grupo Archaea como parte do sistema de tres dominios.[2]
Clasificación[editar | editar a fonte]
Novo dominio[editar | editar a fonte]
Durante a maior parte do século XX os procariotas considerábanse como un único grupo de organismos e clasificábanse segundo as súas características bioquímicas, morfoloxía e metabolismo. Por exemplo, os microbiólogos clasificaban os microorganismos segundo a estrutura da súa parede celular, forma e substancias que consumían.[16] Porén, en 1965 propúxose un novo enfoque,[17] usar as secuencias dos xenes destes organismos para coñecer as relacións entre os distintos grupos de procariotas. Este enfoque, coñecido como filoxenética, é o principal método utilizado dende entón.

As Archaea foron ao primeiro clasificadas como un grupo separado de procariotas en 1977 por Carl Woese e George E. Fox en árbores filoxenéticas que estaban baseadas na comparación das secuencias dos xenes dos ARN ribosómicos.[18] Estes dous grupos foron nomeados entón Archaebacteria e Eubacteria e tratados como reinos ou subreinos, que Woese e Fox denominaron Urkingdoms. Woese consideraba que este grupo de procariotas era unha forma de vida fundamentalmente diferente das outras e para salientar esa diferenza, os dous dominios procariotas foron despois redenominados Archaea e Bacteria.[19]
Inicialmente, só se situaron no novo dominio os metanóxenos, e as arqueas víanse como extremófilos que só vivían en fontes termais e lagos salgados. Ao final do século XX, os microbiólogos decatáronse de que as arqueas formaban un grupo amplo e diverso extensamente distribuído na natureza e común en hábitats moito menos extremos, como solos e océanos.[8] Esta nova apreciación da importancia e ubicuidade das arqueas chegou co uso da reacción en cadea da polimerase para detectar a presenza de procariotas nas mostras de auga e solo baseándose só nos seus ácidos nucleicos. Isto permitiu a detección e identificación de organismos que non se cultivaran no laboratorio.[3][4]
Clasificación actual[editar | editar a fonte]
- Véxase tamén: Clasificación biolóxica e Taxonomía bacteriana.
A clasificación das arqueas, e dos procariotas en xeral, é un campo rapidamente cambiante e controvertido. Os sistemas de clasificación actuais pretenden organizar as arqueas en grupos de organismos que comparten características estruturais e devanceiros comúns.[20] Estas clasificacións dependen principalmente da comparación das secuencias dos xenes do ARNr para revelar as relacións entre os distintos grupos (filoxenia molecular).[21] A maioría das especies cultivadas e ben investigadas de arqueas son membros de dous filos principais, Euryarchaeota e Crenarchaeota. Foron propostos ademais outros grupos para especies peculiares como Nanoarchaeum equitans, descuberta en 2003, que se clasificou no seu propio filo, o das Nanoarchaeota.[22] Tamén se propuxo o novo filo Korarchaeota, que contén un pequeno grupo de especies termófilas que comparten características dos dous filos principais de arqueas, pero que está máis relacionado cos Crenarchaeota.[23][24] Outras especies de arqueas están só lonxanamente relacionadas cos outros grupos, como os organismos ARMAN (Archaeal Richmond Mine Acidophilic Nanoorganisms, Nanoorganismos Acidófilos Arqueanos da Mina Richmond), que foron descubertos en 2006[25] e son uns dos organismos máis pequenos coñecidos.[26]
O filo proposto en 2008 dos Thaumarchaeota figuraba tamén na LPSN (List of Prokaryotic names with Standing in Nomenclature[27]). Conforme se codifiquen máis xenomas de arqueas é probable que o número de grupos aumente, e tamén será necesario comprobar os grupos controvertidos. Por exemplo, en 2011 propúxose o novo grupo dos Aigarchaeota.

As características dos filos son:
- Crenarchaeota: Teñen varias características comúns e xeralmente son hipertermófilos, acidófilos, redutores e/ou oxidantes do xofre e quimiolitoheterótrofos.
- Euryarchaeota: É o grupo máis variado, con catro clases que son metanóxenas, tres termoacidófilas e dúas hiperhalófilas. Tamén abundan en ambientes mariños.
- Korarchaeota: Son escasas e encóntranse en fontes termais.
- Nanoarchaeota: Hipertermófilos ou acidófilos moi pequenos. Considérase que as ARMAN, con só 300 nm de diámetro, son os procariotas máis pequenos.
- Thaumarchaeota: Son quimiolitoautótrofos nitrificantes de ambientes mariños e terrestres.
- Aigarchaeota: Proposto en 2011, de características intermedias entre mesófilos e hipertermófilos.[28]
Modificacións recentes[editar | editar a fonte]
En 2011 propúxose que un superfilo chamado TACK, que inclúe os Thaumarchaeota (agora chamados Nitrososphaerota), "Aigarchaeota", Crenarchaeota (agora Thermoproteota), e "Korarchaeota" estaba relacionado coa orixe dos eucariotas.[29] En 2017 propúxose que o superfilo de novo descubrimento e coa nova denominación Asgard estaba máis estreitamente relacionado cos eucariotas orixinais e era un grupo irmán de TACK.[30]
En 2013 propúxose o superfilo DPANN no que se agrupaban os "Nanoarchaeota", "Nanohaloarchaeota", Nanoorganismos Acidófilos Arqueanos da Mina Richmond (ARMAN, que comprendían os "Micrarchaeota" e "Parvarchaeota"), e outras arqueas similares. Este superfilo arqueano abrangue polo menos 10 liñaxes diferentes e inclúe organismos con células e tamaños xenómicos extremadamente pequenos e limitadas capacidades metabólicas. Por tanto, moitos membros de DPANN poden ser obrigatoriamente dependentes de interaccións simbióticas con outros organismos e poden mesmo incluír novos parasitos. Porén, noutras análises filoxenéticas atopouse que DPANN non forma un grupo monofilético e que se orixina pola chamada atracción das ramas longas, o que suxire que todas estas liñaxes pertencen aos "Euryarchaeota".[31][32][33]
Filoxenia[editar | editar a fonte]
Un estudo realizado en 2013 baseado en 38 xenes marcadores, organiza a filoxenia arqueana en supergrupos, incluíndo novos filos candidatos do seguinte xeito:[34]
| Archaea |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||
De acordo con Tom A. Williams et al. 2017,[37] Castelle e Banfield (2018)[38] e a entrega da GTDB 08-RS214 (do 28 de abril de 2023) a filoxenia das arqueas é a representada nos seguintes cladogramas:[39][40][41]
| Tom A. Williams et al. 2017[37] e Castelle e Banfield 2018[38] | 08-RS214 (28 de abril de 2023)[39][40][41] | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
Especies[editar | editar a fonte]
A clasificación das arqueas en especies é tamén controvertida. En Bioloxía defínese especie como un grupo de organismos relacionados. O criterio de que se poden reproducir entre si e non con outros, que se pode aplicar con relativa facilidade a animais ou plantas, non é de ningunha axuda coas arqueas, que se reproducen asexualmente.[7]
Ademais, as Archaea mostran un alto grao de transferencia horizontal de xenes entre distintas liñaxes. Algúns investigadores suxiren que os individuos poden agruparse en poboacións similares a especies cando se observa unha gran similitude dos seus xenomas e unha pouco frecuente transferencia de xenes a/de células que teñan xenomas menos relacionados, como no caso do xénero Ferroplasma.[43] Por outra parte, estudos do xénero Halorubrum si atoparon unha transferencia de xenes significativa a/doutras poboacións menos relacionadas, o que limita a aplicabilidade deste criterio.[44] Unha segunda preocupación é en que medida esas designacións de especies teñen un significado práctico.[45]
Os coñecementos que se teñen da súa diversidade xenética son fragmentarios e o número total de especies de arqueas non se pode estimar con precisión.[21] A estimación dos posibles filos que podería haber (case todos sen describir) é de entre 18 e 23, dos cales só 8 teñen representantes que fosen cultivados e estudados directamente (na LPSN figuran 5 filos). Moitos destes grupos hipotéticos só se coñecen por unha soa secuencia de ARNr, o que indica que a diversidade entre estes organismos permanece pouco clara.[46] Entre as bacterias tamén hai moitos microbios non cultivados con implicacións similares para a súa caracterización.[47]
Orixe e evolución[editar | editar a fonte]

Aínda que se encontraron probables fósiles de procariotas de case 3.500 millóns de anos de antigüidade, a morfoloxía da maioría dos procariotas e dos seus fósiles non permite distinguir entre bacterias e arqueas.[48] Porén, os fósiles químicos de lípidos característicos das arqueas son máis informativos, porque ditos compostos non aparecen noutros organismos.[49] Algunhas publicacións suxiren que se encontran lípidos característicos de arqueas ou eucariotas en lousas de hai 2.700 millóns de anos;[50] pero estes datos foron cuestionados.[51] Tales lípidos foron tamén detectados en fósiles do Precámbrico. A máis antiga destas trazas procede do distrito de Isua, ao oeste de Groenlandia, onde se encontran os sedimentos máis antigos da Terra, formados hai 3.800 millóns de anos.[52] A liñaxe das arqueas pode ser a máis antiga da Terra.[53]
Woese consideraba que as bacterias, arqueas e eucariotas representan liñas separadas de descendencia que diverxeron cedo na evolución a partir de colonias de organismos ancestrais.[54][55] Unha posibilidade[55][56] é que isto ocorrese antes da evolución das células, cando a falta dunha membrana celular típica permitía unha transferencia lateral de xenes non restrinxida, e que o antepasado común dos tres dominios se orixinou por fixación dun subconxunto específico de xenes.[55][56] É posible que o último devanceiro común das bacterias e arqueas fose un termófilo, o que formula a posibilidade de que os ambientes de baixa temperatura fosen un "ambiente extremo" para as arqueas, e que os organismos que vivían en ambientes máis fríos non aparecesen ata máis tarde.[57] Como Archaea e Bacteria non están máis relacionados entre si có que o están cos eucariotas, ao termo procariota só lle queda o significado de 'non eucariota', o que limita a súa utilidade.[58]
Comparación con outros dominios[editar | editar a fonte]

A seguinte táboa describe algunhas das características principais que as arqueas comparten cos outros dominios ou que lles son exclusivas.[60] Moitas destas características serán discutidas máis abaixo.
| Compartidas con Bacteria | Compartidas con Eukarya | Exclusivas de Archaea |
|---|---|---|
| Sen núcleo nin orgánulos membranosos | Sen peptidoglicano | Estrutura da parede celular (por exemplo, algunhas arqueas teñen paredes con pseudomureína) |
| Xenoma circular | ADN asociado con histonas[61][62] | Membrana celular que contén lípidos con enlace éter |
| Xenes agrupados en operóns | Tradución de proteínas que se inicia coa metionina | Proteína flaxelina característica[63] |
| Sen intróns nin procesamento do ARN | ARN polimerase, promotores, e outra maquinaria tanscricional similar[63][64][65] | Estrutura dos ribosomas (característica compartida tanto con Bacteria coma con Eukarya) |
| ARNm policistrónico | Replicación do ADN e reparación similares[66] | Secuencia do ARNt e metabolismo[63][67] |
| Tamaño celular (>100 veces menor cós eucariotas) | ATPase similar (ATPase V ou Tipo V) | Sen o encima ácido graxo sintase[63] |
Relacións con outros procariotas[editar | editar a fonte]
A relación entre os tres dominios é de grande importancia para comprender a orixe da vida. A maioría das vías metabólicas, que implican a maioría dos xenes dun organismo, son comúns entre Archaea e Bacteria, e a maioría dos xenes implicados na expresión do xenoma son comúns entre Archaea e Eukarya.[68] Nos procariotas, a estrutura da célula das arqueas é moi similar á das bacterias grampositivas, principalmente porque ambas as dúas teñen unha bicapa lipídica[69] e xeralmente contén un groso sáculo de variada composición química.[70] Nas árbores filoxenéticas baseadas nas secuencias de diferentes xenes/proteínas de homólogos procariotas, os homólogos de arqueas están máis próximos aos das bacterias grampositivas.[69] As arqueas e as bacterias grampositivas tamén comparten indeis en varias proteínas importantes, como a Hsp70 e a glutamina sintetase I.[69][71][72]
R. S. Gupta propuxo que as arqueas evolucionaron de bacterias grampositivas en resposta a unha presión selectiva exercida por antibióticos liberados por outras bacterias.[69][71][73] Esta idea está apoiada en que as arqueas son resistentes a unha ampla variedade de antibióticos producidos principalmente por bacterias grampositivas,[69][71] e en que estes antibióticos actúan principalmente sobre xenes que distinguen as arqueas das bacterias. A súa proposta é que a presión selectiva cara á resistencia xerada polos antibióticos das grampositivas foi finalmente suficiente para causar grandes cambios en moitos dos xenes que eran o obxectivo dos antibióticos, e que estas cepas de microorganismos representaban o devanceiro común das Archaea actuais.[73] A evolución das Archaea en resposta á selección por antibióticos, ou calquera outra presión selectiva competitiva, podería tamén explicar a súa adaptación a ambientes extremos (como a alta temperatura ou acidez) como resultado dunha procura de nichos ecolóxicos desocupados para escapar dos organismos produtores de antibióticos;[73][74] Cavalier-Smith fixo unha suxestión similar.[75] A proposta de Gupta está tamén apoiada por outros traballos de investigación sobre as relacións entre proteínas estruturais[76] e por estudos que suxiren que as bacterias grampositivas poden constituír unha das liñaxes que primeiro se ramificaron nos procariotas.[77]
Relación cos eucariotas[editar | editar a fonte]
As relacións evolutivas entre arqueas e eucariotas non están claras. Á parte das semellanzas na estrutura e funcións celulares que serán discutidas máis abaixo, moitas árbores xenéticas agrupan as dúas liñaxes.
As complicacións aumentan porque algúns opinan que as relacións entre os eucariotas e o filo arqueano das Crenarchaeota son máis próximas cás relacións entre os filos arqueanos de Euryarchaeota e Crenarchaeota,[78] e pola presenza de xenes de tipo arqueano en certas bacterias, como Thermotoga maritima, debido a transferencia horizontal de xenes.[79] A hipótese principal é que o antepasado dos eucariotas diverxeu moi cedo das Archaea,[80][81] e que os eucariotas se orixinaron pola fusión dunha arquea e unha eubacteria, que deron lugar ao núcleo e ao citoplasma, respectivamente; isto explicaría varias semellanzas xenéticas pero atopa dificultades para explicar a estrutura celular.[82] Finalmente, outra hipótese establece que Eukaryota se relaciona con Archaea pola proximidade de ambos co superfilo TACK (siglas de catro filos arqueanos).[83] O descubrimento do clado TACK é fundamental para a comprensión da orixe das arqueas e da primeira célula eucariota. Nas diferentes árbores filoxenéticas elaboradas Archaea estaría relacionada con Eukaryota de diversas maneiras. Cruzando estes datos obtense un resultado que desbotaría a árbore dos tres dominios, apoiando a hipótese do eocito e á simbioxénese preeucariota e deduciríase a presenza do superfilo que agrupa a varios filos arqueanos con Eukaryota baixo as siglas TACK,[84] o que, segundo as árbores filoxenéticas dos partidarios desta hipótese, pode resumirse do seguinte modo:
| Prokaryota |
| |||||||||||||||||||||||||||
Cuestionamento do sistema de tres dominios[editar | editar a fonte]
En hipóteses anteriores como a de Woese, argumentouse que as bacterias, arqueas e eucariotas representaban tres liñaxes evolutivas distintas que diverxeran hai moitos millóns de anos dun grupo ancestral de organismos.[85][86] Outros argumentaron que as arqueas e eucariotas xurdiron dun grupo de bacterias.[87] Cavalier-Smith propuxo o clado Neomura para representar esta teoría; Neomura significa 'paredes novas' e fai referencia á teoría de que as arqueas e os eucariotas terían derivado de bacterias que (entre outras adaptacións) substituíran as paredes de peptidoglicano por outras de glicoproteínas. Segundo Woese, como arqueas e bacterias non estarían relacionadas máis estreitamente unhas con outras que cos eucariontes, propúxose que o termo "procariota" non tería sentido evolutivo auténtico e que habería que desbotalo por completo.[58] Porén, moitos biólogos evolutivos consideran que no sistema de tres dominios se esaxerou a diferenza entre arqueas e bacterias, e sosteñen que a transición máis drástica se produciu entre Prokaryota e Eukaryota (sistema de dous imperios), este último de orixe máis recente por eucarioxénese e como resultado da fusión endosimbiótica de polo menos dous procariontes: unha arquea e unha bacteria.
Morfoloxía[editar | editar a fonte]

O tamaño das arqueas vai de 0,1 a 15 μm de diámetro, e a forma pode ser moi variada, normalmente esférica, bacilar, espiral ou plana.[88] Outras morfoloxías que aparecen nos Crenarchaeota son células lobuladas irregulares en Sulfolobus, filamentos con forma de agulla de menos dun micrómetro de diámetro en Thermofilum, e bacilos case perfectamente rectangulares en Thermoproteus e Pyrobaculum.[89] Haloquadratum walsbyi é unha arquea plana cadrada que vive en pozas hipersalinas.[90] Estas formas infrecuentes mantéñense probablemente grazas ás súas paredes celulares e ao citoesqueleto procariota. Nas arqueas existen proteínas relacionadas co citoesqueleto doutros organismos,[91] e forman filamentos dentro das células,[92] pero a diferenza deles, estas estruturas celulares das arqueas non son ben coñecidas.[93] En Thermoplasma e Ferroplasma a falta de parede celular implica que a célula ten formas irregulares e pode parecer unha ameba.[94]
Algunhas especies forman agregados ou filamentos de células de ata 200 μm de longo.[88] Estes organismos poden ser abondosos nos biofilmes.[95] Hai que salientar que agregados de células de Thermococcus coalescens se fusionan en cultivo, formando unha soa célula xigante.[96] As arqueas do xénero Pyrodictium producen unha elaborada colonia multicelular na que hai un conxunto de tubos ocos longos e finos chamados cánulas que sobresaen da superficie das células e conéctanse formando unha aglomeración de aspecto arbustivo.[97] A función destas cánulas non está establecida, pero poden permitir a comunicación ou intercambio de nutrientes coas células veciñas.[98] Tamén existen colonias formadas por membros de varias especies distintas, como a comunidade de "colar de perlas" descuberta en 2001 nun pantano alemán. Nestas comunidades, colonias arredondadas branquechas dunha nova especie de Euryarchaeota están mesturadas con filamentos finos de ata 15 cm de longo formados por unha determinada especie de bacteria.[99]
Estrutura e composición[editar | editar a fonte]
As arqueas e as bacterias teñen xeralmente unha estrutura celular similar, pero a composición e organización da célula diferenza ás arqueas. Igual que as bacterias, as arqueas carecen de membranas internas e orgánulos membranosos.[58] Igual que as bacterias, as membranas plasmáticas das arqueas están normalmente rodeadas por unha parede celular e nadan utilizando un ou máis flaxelos.[100] Estruturalmente, as arqueas son máis similares ás bacterias grampositivas. A maioría teñen unha única membrana celular e parede celular, e carecen de espazo periplásmico (ou periplasma); a excepción a esta regra xeral é Ignicoccus, que posúe un periplasma especialmente grande que contén vesículas rodeadas de membrana e está encerrado dentro dunha membrana externa.[101]
Membranas[editar | editar a fonte]

As membrana arqueanas están formadas por moléculas que difiren moito das que se encontran noutras formas de vida, o que indica que as arqueas están só emparentadas moi de lonxe con bacterias e eucariotas.[102] En todos os organismos as membranas celulares están feitas de moléculas chamadas fosfolípidos. Estas moléculas son anfipáticas, xa que posúen unha parte polar ou hidrófila que ten afinidade pola auga (a cabeza fosfato), e unha parte apolar ou hidrófoba "graxa" que non ten esa afinidade (a cola lipídica). Estas dúas partes están conectadas por unha molécula de glicerol. Na auga, os fosfolípidos agrúpanse, colocando as súas cabezas polares en contacto coa auga e as partes apolares en dirección contraria. A principal estrutura das membranas celulares é unha bicapa lipídica formada por estes fosfolípidos.
Estes fosfolípidos son infrecuentes por catro razóns:
- As bacterias e os eucariotas teñen membranas compostas principalmente de lípidos glicerol-éster, pero nas arqueas as membranas están compostas de lípidos glicerol-éter.[6] A diferenza é o tipo de enlace (éter en vez de éster) que une os lípidos á molécula de glicerol; os dous tipos móstranse en amarelo na figura da dereita. Os enlaces éter son quimicamente máis resistentes cós enlaces éster. Esta estabilidade maior podería axudar ás arqueas a sobrevivir mellor nas extremas temperaturas ou condicións moi ácidas ou moi alcalinas dos ambientes nos que viven.[103] As bacterias e os eucariotas conteñen algúns lípidos con enlace éter, pero, a diferenza coas arqueas, estes lípidos non constitúen a maior parte das súas membranas.
- A estereoquímica do residuo de glicerol é nas arqueas a contraria á que se encontra nos outros organismos. O glicerol dos lípidos das arqueas é o enantiómero L e nos demais o D. Isto suxire que as arqueas utilizan encimas completamente diferentes para sintetizar fosfolípidos (adaptados ao L-glicerol) aos que utilizan as bacterias e eucariotas. Eses encimas deberon desenvolverse moi cedo na historia dos seres vivos, o que suxire unha separación temperá das arqueas dos outros dous dominios.[102]
- As colas hidrocarbonadas dos lípidos arqueanos son quimicamente diferentes ás dos outros organismos. Os lípidos das arqueas están formados por cadeas isoprenoides e son longas e con múltiples ramificacións, e ás veces mesmo con aneis de ciclopropano ou ciclohexano.[104] As membranas doutros organismos están formadas por ácidos graxos, e teñen cadeas liñais sen ramificacións e sen aneis. Aínda que os isoprenoides xogan un importante papel na bioquímica de moitos organismos, só as arqueas os utilizan para fabricar fosfolípidos. Estas cadeas ramificadas poden axudar a impermeabilizar as membranas arqueanas a altas temperaturas.[105]
- Nalgunhas arqueas a bicapa lipídica é substituída por unha monocapa. De feito, as arqueas fusionan as colas de dúas moléculas de fosfolípido distintas formando unha soa molécula con dúas cabezas polares nos extremos e unha parte central hidrófoba (un bolaanfífilo); esta fusión pode facer as súas membranas máis ríxidas e mellor adaptadas para resistir os seus ambientes.[106] Por exemplo, os lípidos de Ferroplasma son deste tipo, o que se cre que facilita a supervivencia deste organismo no seu hábitat, que é moi ácido.[107]
Parede celular e flaxelos[editar | editar a fonte]
A maioría das arqueas (agás Thermoplasma e Ferroplasma) posúen parede celular.[94] Na maioría das arqueas a parede está unida a proteínas dunha capa superficial, que forman a chamada capa S.[108] Unha capa S é un armazón proteico ríxido que cobre exteriormente a célula (como unha cota de malla).[109] Esta capa proporciónalles protección química e física, e pode impedir que macromoléculas externas contacten coa membrana celular.[110] A diferenza das bacterias, as arqueas non teñen peptidoglicano na parede celular.[111] As Methanobacteriales teñen parede celular que contén pseudopeptidoglicano, o cal lembra o peptidoglicano das eubacterias en morfoloxía, función e estrutura física, pero é distinto en estrutura química, xa que lle faltan os D-aminoácidos e o ácido N-acetilmurámico.[110]
Os flaxelos arqueanos funcionan como os bacterianos, é dicir, ao seu longo talo móvese por motores rotatorios situados na base. Porén, os flaxelos arqueanos son completamente diferentes en composición e desenvolvemento.[100] Os dous tipos de flaxelos evolucionaron de diferentes antepasados. O flaxelo bacteriano ten homoloxías co sistema de secreción de tipo III,[112][113] mentres que o flaxelo arqueano parece que evolucionou do pili bacteriano tipo IV.[114] A diferenza do flaxelo bacteriano, que é oco e está formado por subunidades que flúen polo oco central ata o extremo, onde se ensamblan, os flaxelos arqueanos sintetízanse engadindo subunidades na base.[115]
Metabolismo[editar | editar a fonte]
- Véxase tamén: Metabolismo microbiano.

As arqueas mostran unha gran variedade de reaccións químicas no seu metabolismo e usan moitas fontes de enerxía. Estas reaccións clasifícanse dependendo das fontes de enerxía e carbono. Algunhas arqueas obteñen enerxía de compostos inorgánicos como o xofre e o amoníaco (son litótrofos). Entre eles están as arqueas nitrificantes, metanóxenas e oxidantes anaeróbicas do metano.[116] Nestas reaccións un composto pasa electróns a outro nunha reacción redox, liberando enerxía, que será consumida nas actividades celulares. Un composto actúa como doante de electróns e o outro como aceptor de electróns. A enerxía liberada xera adenosín trifosfato (ATP) por medio de quimiosmose, polo mesmo proceso básico que ocorre nas mitocondrias das células eucarióticas.[117]
Outros grupos de arqueas usan a luz como fonte de enerxía (son fotótrofos). Porén, nestes organismos non ten lugar a fotosíntese xeradora de osíxeno.[117] Moitas rutas metabólicas básicas son compartidas por todas as formas de vida; por exemplo, as arqueas utilizan unha forma modificada da glicólise e presentan un ciclo do ácido cítrico completo ou parcial.[63] Estas similitudes con outros organismos reflicten probablemente que son organismos que apareceron moi cedo na historia da vida e que teñen un alto grao de eficiencia.[118]
| Tipo nutricional | Fonte de enerxía | Fonte de carbono | Exemplos |
|---|---|---|---|
| Fotótrofos | Luz solar | Compostos orgánicos | Halobacteria |
| Litótrofos | Compostos inorgánicos | Compostos orgánicos ou fixación do carbono | Ferroglobus, Methanobacteria ou Pyrolobus |
| Organótrofos | Compostos orgánicos | Compostos orgánicos ou fixación do carbono | Pyrococcus, Sulfolobus ou Methanosarcinales |
Algunhas Euryarchaeota son metanóxenas e viven en ambientes anaeróbicos como os pantanos. Esta forma de metabolismo evolucionou moi cedo, e é posible que os primeiros organismos vivos de vida libre fosen metanóxenos.[119] Unha reacción común implica o uso de dióxido de carbono como aceptor de electróns para oxidar hidróxeno. A metanoxénese implica o uso dun conxunto de coencimas que é exclusivo destas arqueas, como o coencima M e o metanofurano.[120] Como alternativa os metanóxenos poden usar outros compostos orgánicos como alcohois, ácido acético ou ácido fórmico como aceptores de electróns. Estas reaccións son comúns nas arqueas que viven no tracto dixestivo de animais. O ácido acético tamén o poden degradar directamente a metano e dióxido de carbono as arqueas acetotróficas. Estes acetótrofos son arqueas da orde Methanosarcinales, e constitúen a maior parte das comunidades microbianas que producen biogás.[121]

Outras arqueas utilizan o CO2 atmosférico como fonte de carbono, nun proceso chamado fixación do carbono (son autótrofos). Este proceso pode utilizar unha forma moi modificada do ciclo de Calvin[123] ou ben unha vía metabólica descuberta en 2007 chamada ciclo do 3-hidroxipropionato/4-hidroxibutirato.[124] As Crenarchaeota tamén usan o ciclo de Krebs inverso, e as Euryarchaeota usan a vía do acetil-CoA redutiva.[125] A fixación do carbono está impulsada por fontes de enerxía inorgánicas. Ningunha arquea realiza unha fotosíntese como a das plantas.[126] As fontes de enerxía das arqueas son moi diversas, e van desde a oxidación do amoníaco nas Nitrosopumilales[127][128] á oxidación do sulfuro de hidróxeno ou do xofre elemental por especies do xénero Sulfolobus, utilizando oxíxeno ou ións metálicos como aceptores de electróns.[117]
As arqueas fotótrofas utilizan a luz para producir enerxía química en forma de ATP. As Halobacteria teñen bombas iónicas activadas pola luz, como a bacteriorrodopsina e a halorrodopsina, e xeran gradientes iónicos bombeando ións a través da membrana plasmática. A enerxía almacenada nestes gradientes electroquímicos é despois convertida en ATP pola ATP sintase.[88] Este proceso é unha forma de fotofosforilación. A capacidade destas bombas impulsadas pola luz de moveren ións a través das membranas depende de cambios causados pola luz na estrutura do cofactor retinol situado no centro da proteína.[129]
Xenética[editar | editar a fonte]
As arqueas xeralmente teñen un só cromosoma circular,[130] cun tamaño que vai desde 5.751.492 pares de bases en Methanosarcina acetivorans,[131] que é o meirande, ata só 490.885 pares de bases (10 veces menos) en Nanoarchaeum equitans, que é o menor entre as arqueas e que se estima que contén só 537 xenes codificantes de proteínas.[132] As arqueas tamén teñen pequenos círculos independentes máis pequenos de ADN chamados plásmidos. Os plásmidos poden ser transferidos dunha célula a outra por contacto físico, nun proceso que pode ser similar á conxugación bacteriana.[133][134]

As arqueas poden ser infectadas por virus de ADN bicatenario que non están relacionados con ningún outro tipo de virus e teñen varias formas pouco usuais, como forma de botella, bastóns ganchudos ou bágoa.[136] Estes virus foron estudados en maior detalle en termófilos das ordes Sulfolobales e Thermoproteales.[137] En 2009 identificouse un virus de ADN monocatenario que infectaba arqueas halófilas.[138] As defensas contra estes virus poden ser secuencias de ADN repetitivo do xenoma arqueano, que son homólogas con xenes dos virus, e que funcionan dun modo similar á interferencia de ARN.[139][140]
As arqueas son xeneticamente distintas das bacterias e dos eucariotas. Ata o 15% das proteínas codificadas polo xenoma arqueano son exclusivas dese dominio de seres vivos, pero a maioría deses xenes non teñen función coñecida.[141] Dos poucos dos que se coñece a función, a maioría son de euriarqueotas e están implicados na metanoxénese. As proteínas que comparten as arqueas coas bacterias e eucariotas forman un núcleo común para as funcións celulares básicas, e están relacionadas principalmente coa transcrición xenética, tradución de proteínas e metabolismo de nucleótidos.[142] Outras características tipicamente arqueanas son a organización dos xenes de funcións relacionadas como encimas que catalizan reaccións dunha mesma ruta metabólica agrupados en novos operóns, e grandes diferenzas nos xenes dos ARNt e das aminoacil ARNt sintetases.[142]
A transcrición e tradución nas arqueas é máis parecida á dos eucariotas ca á das bacterias, xa que as ARN polimerase e ribosomas de arqueas están moi próximos aos seus equivalentes en eucariotas.[130] Aínda que as arqueas só teñen un tipo de ARN polimerase, a súa estrutura e función na transcrición parece estar próxima á da ARN polimerase II eucariótica, cunha ensamblaxe similar de proteínas (os factores de transcrición xerais) que dirixen a unión da ARN polimerase ao promotor do xene.[143] Porén, outros factores de transcrición de arqueas son parecidos aos das bacterias.[144] A modificación postranscricional é máis simple ca nos eucariotas, xa que a maior parte dos xenes de arqueas carecen de intróns, pero teñen moitos intróns nos seus xenes dos ARNt e ARNr,[145] e hai tamén intróns nuns poucos xenes codificantes de proteínas.[146][147]
Transferencia de xenes e intercambio xenético[editar | editar a fonte]
Haloferax volcanii (antes chamada Halobacterium volcanii), unha arquea halófila extrema, forma pontes citoplasmáticas entre células que parecen utilizarse para a transferencia de ADN dunha célula a outra en ambas as direccións.[148]
Cando as arqueas hipertermófilas Sulfolobus solfataricus[149] e Sulfolobus acidocaldarius[150] son expostas a axentes que producen danos no ADN como radiación ultravioleta, bleomicina ou mitomicina C, indúcese unha agregación celular específica de especie. A agregación en S. solfataricus non pode ser inducida por outros estresantes físicos, como os cambios de pH e temperatura,[149] o que suxire que a agregación está inducida especificamente polos danos no ADN. Ajon et al.[150] mostraron que a agregación celular inducida pola luz UV vai acompañada do intercambio de marcadores cromosómicos con alta frecuencia en S. acidocaldarius. As proporcións de recombinación exceden as de cultivos non inducidos en ata tres ordes de magnitude. Frols et al.[149][151] e Ajon et al.[150] formularon a hipótese de que a agregación celular potencia a transferencia de ADN específica de especie entre células de Sulfolobus para proporcionar un incremento na reparación dos danos no ADN por medio da recombinación homóloga. Esta resposta pode ser unha forma primitiva de interacción sexual similar aos sistemas de transformación bacterianos mellor estudados que están tamén asociados coa transferencia de ADN específica de especie entre células, que orixina unha reparación recombinacional homóloga dos danos no ADN.[152]
Reprodución[editar | editar a fonte]
As arqueas reprodúcense asexualmente por fisión binaria ou múltiple, fragmentación ou xemación; non hai meiose, de modo que se unha arquea existe en máis dunha forma fenotípica todas elas teñen o mesmo material xenético.[88] A división celular ten lugar despois de que o cromosoma se replica e os dous cromosomas fillos se separan.[153] Só se investigaron os detalles no xénero Sulfolobus, pero viuse que o seu ciclo celular ten características similares tanto ao das bacterias coma ao eucariótico. Os cromosomas replícanse empezando desde múltiples puntos (orixes de replicación) utilizando ADN polimerases parecidas aos equivalentes eucarióticos.[154] Porén, as proteínas que dirixen a división celular, como a proteína FtsZ, que forma un anel de contracción arredor da célula, e os compoñentes do septo que dividirá ás dúas células fillas, son similares aos equivalentes bacterianos.[153]
As bacterias e os eucariotas, pero non as arqueas, forman esporas.[155] Algunhas especies de Haloarchaea sofren cambios fenotípicos e crecen como células de varios tipos diferentes, como estruturas de paredes grosas resistentes aos choques osmóticos, que posibilitan que as arqueas poidan vivir en ambientes con baixas concentracións salinas, pero que non son estruturas reprodutivas senón que poden servir para colonizar novos hábitats.[156]
Ecoloxía[editar | editar a fonte]
Hábitats[editar | editar a fonte]
As arqueas viven nunha gran variedade de hábitats, e son unha parte importante dos ecosistemas globais,[8] e poden supoñer ata o 20% das células no océano.[157] As primeiras arqueas que se descubriron eran extremófilas.[116] Algunhas poden vivir a altas temperaturas, a miúdo por riba dos 100 °C, en géyseres, chemineas negras submarinas e pozos de petróleo. Outros hábitats comúns son ambientes acuáticos moi fríos ou moi salinos, ácidos ou alcalinos. Porén, entre as arqueas tamén hai moitas especies mesófilas que crecen en condicións suaves, en pantanos, auga residual, océanos e solos.[8]

As arqueas extremófilas poden pertencer a catro grupos fisiolóxicos principais. Son os halófilos, termófilos, alcalófilos e acidófilos.[158] Estes grupos non pertencen a filos específicos, e non son mutuamente exclusivos dentro dun filo, xa que algunhas arqueas destes tipos pertencen a varios grupos, pero agrupalas así é un útil punto de comezo para a clasificación.
Os halófilos, como o xénero Halobacterium, viven en ambientes extremadamente salinos, como lagos salgados, e superan ás bacterias cando a salinidade supera o 20-25%.[116] As termófilas crecen mellor a temperaturas superiores a 45 °C, en lugares como fontes termais; as hipertermófilas crecen optimamente a temperaturas superiores a 80 °C.[159] A arquea Methanopyrus kandleri cepa 116 crece a 122 °C, que é a máxima temperatura rexistrada para un organismo vivo.[160]
Outras arqueas viven en condicións moi ácidas ou moi alcalinas.[158] Por exemplo, unha das arqueas acidófilas máis extremas é Picrophilus torridus, que crece a pH 0, o que equivale a vivir nunha concentración de ácido sulfúrico 1,2 molar.[161]
Esta resistencia a ambientes extremos fixo que as arqueas fosen o foco de especulacións sobre as posibles propiedades da vida extraterrestre.[162] Algúns hábitats extremófilos non son moi distintos dos que existen en Marte,[163] o que levou a suxerir que algúns microbios viables poderían ser transferidos dun planeta a outro por meteoritos (panspermia).[164]
No ano 2001, varios estudos encontraron que as arqueas viven non só en ambientes mesófilos e termófilos senón tamén, e ás veces con grande abundancia, a baixas temperaturas. Por exemplo, as arqueas son comúns en ambientes oceánicos fríos como os mares polares.[165] Aínda máis significativa é a gran cantidade de arqueas que viven nos océanos en condicións non extremas formando parte do plancto (como parte do picoplancto).[166] Aínda que estas arqueas poden estar presentes en enorme número (ata o 40% da biomasa microbiana), case ningunha destas especies foi illada e estudada nun cultivo puro.[167] En consecuencia, a nosa comprensión do papel das arqueas na ecoloxía dos océanos é rudimentaria, polo que a súa influencia na bioxeoquímica global está en gran medida inexplorada.[168] Algunhas Crenarchaeota mariñas poden facer a nitrificación, o que suxire que estes organismos poden afectar o ciclo do nitróxeno oceánico,[169] aínda que estas Crenarchaeota oceánicas poden utilizar outras fontes de enerxía.[170] Gran número de arqueas atópanse nos sedimentos que cobren o leito do mar, e estes organismos son a maioría dos que viven a 1 metro de profundidade nos sedimentos oceánicos.[171][172]
Papel nos ciclos bioxeoquímicos[editar | editar a fonte]
- Véxase tamén: Ciclo bioxeoquímico.
As arqueas reciclan elementos como o carbono, o nitróxeno e o xofre nos seus diversos hábitats. Estas actividades son vitais para o funcionamento normal dos ecosistemas, pero as arqueas poden contribuír a acrecentar cambios causados polo ser humano e mesmo poden producir contaminación.
As arqueas poden levar a cabo moitos dos pasos do ciclo do nitróxeno. Isto inclúe tanto reaccións que eliminan o nitróxeno dos ecosistemas, como a respiración baseada no nitrato e a desnitrificación, coma procesos que introducen nitróxeno, como a asimilación do nitrato e a fixación do nitróxeno.[173][174] A implicación das arqueas nas reaccións de oxidación do amoníaco descubriuse no ano 2007. Estas reaccións son especialmente importantes nos océanos.[175][176] As arqueas son tamén importantes na oxidación do amoníaco nos solos. Producen nitrito, que outros microbios oxidan a nitrato. As plantas e outros organismos consomen este último.[177]
No ciclo do xofre, as arqueas que crecen oxidando compostos de xofre liberan este elemento das rochas, facendo que quede dispoñible para outros organismos. Porén, as arqueas que fan isto, como Sulfolobus, producen ácido sulfúrico como produto residual, e o crecemento destes organismos en minas abandonadas pode contribuír á formación dos líquidos da drenaxe ácida de minas e outros danos ambientais.[178]
No ciclo do carbono, as arqueas metanóxenas eliminan hidróxeno e son importantes na descomposición da materia orgánica realizada polas poboacións de microorganismos que actúan como descompoñedores nos sistemas anaeróbicos, como depósitos de sedimentos, pantanos e no tratamento de augas residuais.[10] Porén, o metano é un dos gases de efecto invernadoiro máis abundantes na atmosfera terrestre, e constitúe o 18% do total global.[179] É 25 veces máis potente como gas de efecto invernadoiro có dióxido de carbono.[180] Os microorganismos metanóxenos son a primeira fonte de metano atmosférico, e son responsables da maioría das emisións de metano anuais mundiais.[181] Como consecuencia, estas arqueas contribúen ás emisións de gases de efecto invernadoiro globais e ao quecemento global.
Interaccións con outros organismos[editar | editar a fonte]

As interaccións ben caracterizadas entre as arqueas e outros organismos poden ser do tipo do mutualismo ou do comensalismo. Non hai claros exemplos de arqueas coñecidas como patóxenas ou parasitas.[182][183] Porén, propúxose unha relación entre determinadas arqueas metanóxenas e a periodontite,[184][185] e Nanoarchaeum equitans pode ser parasita doutras especies de arqueas, xa que só sobrevive e se reproduce en asociación coa crenarqueota Ignicoccus hospitalis,[186] e non parece que lle proporcione beneficios ao seu hóspede.[187] A diferenza do caso anterior, os Nanoorganismos Acidófilos Arqueanos da Mina Richmond (ARMAN) ocasionalmente conectan con outras células de arqueas nos biofilmes das drenaxes ácidas das minas.[188] A natureza desta relación é descoñecida, pero é distinta da Nanoarchaeum-Ignicoccus en que as superdiminutas células ARMAN vense normalmente vivindo independentemente das células das Thermoplasmatales coas que viven.
Mutualismo[editar | editar a fonte]
Un exemplo ben coñecido de mutualismo é a interacción que establecen cos ruminantes e as térmites os protozoos e arqueas metanóxenas que viven nos seus tractos dixestivos e lles axudan a dixerir a celulosa.[189] Nestes ambientes anaeróbicos, os protozoos degradan a celulosa da materia vexetal para obter enerxía. Este proceso libera hidróxeno como produto residual, pero os altos niveis de hidróxeno reducen a produción de enerxía. Despois os metanóxenos converten o hidróxeno en metano, e os protozoos benefícianse de máis enerxía.[190]
En protozoos anaeróbicos como Plagiopyla frontata, as arqueas viven dentro dos protozoos e consomen o hidróxeno producido nos seus hidroxenosomas.[191][192] As arqueas tamén se asocian con organismos meirandes. Por exemplo, a arquea mariña Cenarchaeum symbiosum vive dentro (como endosimbionte) dunha esponxa do xénero Axinella.[193]
Comensalismo[editar | editar a fonte]
As arqueas poden ser tamén comensais que se benefician dunha asociación sen danar nin beneficiar o outro organismo. Por exemplo, o metanóxeno Methanobrevibacter smithii é con diferenza a arquea máis común na flora intestinal humana, e supón a décima parte de todos os procariotas do tracto dixestivo humano.[194] En térmites e humanos, estes metanóxenos poden ser mutualistas que interaccionan con outros microbios no tracto dixestivo para axudaren nas funcións do aparato dixestivo.[195] As comunidades arqueanas tamén se asocian con outros organismos, como fan os que viven na superficie do coral,[196] e na rexión do solo que rodea as raíces das plantas (rizosfera).[197][198]
Importancia na tecnoloxía e industria[editar | editar a fonte]
- Véxase tamén: Biotecnoloxía.
As arqueas extremófilas, especialmente as resistentes ás altas temperaturas ou a grande acidez ou alcalinidade, son unha fonte de encimas que poden funcionar nesas duras condicións e que teñen moitas aplicacións.[199][200] Por exemplo, as ADN polimerases termoestables, como a ADN polimerase Pfu de Pyrococcus furiosus, revolucionou a bioloxía molecular ao permitir utilizar a reacción en cadea da polimerase como unha técnica simple para o clonado do ADN. Na industria, as amilases, galactosidases e pululanases doutras especies de Pyrococcus, que funcionan por riba dos 100 °C, permiten o procesamento de alimentos a altas temperaturas, como na produción de leite e soro lácteo baixos en lactosa.[201] Encimas destas arqueas termófilas tamén adoitan a ser moi estables en solventes orgánicos, permitindo o seu uso en procesos inocuos para o ambiente da chamada química verde para sintetizar compostos orgánicos.[200] Esta estabilidade fainos máis fáciles de usar en bioloxía estrutural. En consecuencia os encimas equivalentes aos bacterianos e eucarióticos das arqueas extremófilas son a miúdo utilizados en estudos estruturais.[202]
A diferenza da gran gama de aplicacións que teñen os encimas arqueanos, o uso das propias células das arqueas en biotecnoloxía está menos desenvolvido. As arqueas metanóxenas son unha parte vital dos procesos de tratamento de augas residuais, xa que forman parte da comunidade de microorganismos que levan a cabo a dixestión anaeróbica e producen biogás.[9] No procesamento de minerais, as arqueas acidófilas son moi prometedoras para a extracción de metais de minerais como ouro, cobalto e cobre.[203]
As arqueas producen un novo tipo de antibióticos potencialmente útiles. Algunhas destas arqueocinas xa foron caracterizadas, pero pénsase que deben de existir centos máis, especialmente nas haloarqueas e Sulfolobus.[204] Estes compostos difiren en estrutura dos antibióticos bacterianos, polo que poden ter novas formas de acción. Ademais, poden permitir a creación de novos marcadores seleccionables para o seu uso en bioloxía molecular arqueana.[205]
Notas[editar | editar a fonte]
- ↑ Pace NR (maio de 2006). "Time for a change". Nature 441 (7091): 289. Bibcode:2006Natur.441..289P. PMID 16710401. doi:10.1038/441289a.
- ↑ 2,0 2,1 Woese, C. R.; Kandler, O & Wheelis, M. L. (1990). "Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya". Proc. Natl. Acad. Sci. U.S.A. 87 (12): 4576–9. PMID 2112744. doi:10.1073/pnas.87.12.4576. Arquivado dende o orixinal o 16 de outubro de 2019. Consultado o 08 de xullo de 2012.
- ↑ 3,0 3,1 Theron J, Cloete TE (2000). "Molecular techniques for determining microbial diversity and community structure in natural environments". Crit. Rev. Microbiol. 26 (1): 37–57. PMID 10782339. doi:10.1080/10408410091154174.
- ↑ 4,0 4,1 Schmidt TM (2006). "The maturing of microbial ecology" (PDF). Int. Microbiol. 9 (3): 217–23. PMID 17061212. Arquivado dende o orixinal (PDF) o 16 de decembro de 2008. Consultado o 08 de xullo de 2012.
- ↑ Non confundir con Haloarcula quadrata, que ten forma similar e foi atopada nas mesmas lagoas salinas.
- ↑ 6,0 6,1 De Rosa M, Gambacorta A, Gliozzi A (1986). "Structure, biosynthesis, and physicochemical properties of archaebacterial lipids". Microbiol. Rev. 50 (1): 70–80. PMC 373054. PMID 3083222.
- ↑ 7,0 7,1 de Queiroz K (2005). "Ernst Mayr and the modern concept of species". Proc. Natl. Acad. Sci. U.S.A. 102 (Suppl 1): 6600–7. Bibcode:2005PNAS..102.6600D. PMC 1131873. PMID 15851674. doi:10.1073/pnas.0502030102.
- ↑ 8,0 8,1 8,2 8,3 DeLong EF (1998). "Everything in moderation: archaea as 'non-extremophiles'". Curr. Opin. Genet. Dev. 8 (6): 649–54. PMID 9914204. doi:10.1016/S0959-437X(98)80032-4.
- ↑ 9,0 9,1 Schiraldi C, Giuliano M, De Rosa M (2002). "Perspectives on biotechnological applications of archaea" (PDF). Archaea 1 (2): 75–86. PMC 2685559. PMID 15803645. doi:10.1155/2002/436561. Arquivado dende o orixinal (PDF) o 26 de agosto de 2013. Consultado o 08 de xullo de 2012.
- ↑ 10,0 10,1 Schimel J (2004). "Playing scales in the methane cycle: from microbial ecology to the globe". Proc. Natl. Acad. Sci. U.S.A. 101 (34): 12400–1. Bibcode:2004PNAS..10112400S. PMC 515073. PMID 15314221. doi:10.1073/pnas.0405075101.
- ↑ James G. Ferry (1993). "Methanogenesis: Ecology, Physiology, Biochemistry & Genetics".
- ↑ Kluyver e van Niel 1936
- ↑ Brock TD, Brock KM, Belly RT, Weiss RL (1972). "Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature". Arch. Mikrobiol. (84 (1)): 54–68. PMID 4559703. doi:10.1007/BF00408082.
- ↑ "Bergey's Manual of Systematic Bacteriology". (1ª (4 vols.) ed.). 1984.
- ↑ Lake, James A.; et al. (1984). ""Eocytes: A new ribosome structure indicates a kingdom with a close relationship to eukaryotes"". PNAS (81): 3786–3790.
- ↑ Staley JT (2006). "The bacterial species dilemma and the genomic-phylogenetic species concept". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1475): 1899–909. PMC 1857736. PMID 17062409. doi:10.1098/rstb.2006.1914.
- ↑ Zuckerkandl E, Pauling L (1965). "Molecules as documents of evolutionary history". J. Theor. Biol. 8 (2): 357–66. PMID 5876245. doi:10.1016/0022-5193(65)90083-4.
- ↑ Woese C, Fox G (1977). "Phylogenetic structure of the prokaryotic domain: the primary kingdoms". Proc Natl Acad Sci USA 74 (11): 5088–90. Bibcode:1977PNAS...74.5088W. PMC 432104. PMID 270744. doi:10.1073/pnas.74.11.5088.
- ↑ Woese CR, Kandler O, Wheelis ML (1990). "Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya". Proc. Natl. Acad. Sci. U.S.A. 87 (12): 4576–9. Bibcode:1990PNAS...87.4576W. PMC 54159. PMID 2112744. doi:10.1073/pnas.87.12.4576. Arquivado dende o orixinal o 16 de outubro de 2019. Consultado o 08 de xullo de 2012.
- ↑ Gevers D, Dawyndt P, Vandamme P; et al. (2006). "Stepping stones towards a new prokaryotic taxonomy". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1475): 1911–6. PMC 1764938. PMID 17062410. doi:10.1098/rstb.2006.1915.
- ↑ 21,0 21,1 Robertson CE, Harris JK, Spear JR, Pace NR (2005). "Phylogenetic diversity and ecology of environmental Archaea". Curr. Opin. Microbiol. 8 (6): 638–42. PMID 16236543. doi:10.1016/j.mib.2005.10.003.
- ↑ Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO. (2002). "A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont". Nature 417 (6884): 27–8. PMID 11986665. doi:10.1038/417063a.
- ↑ Barns SM, Delwiche CF, Palmer JD, Pace NR (1996). "Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences". Proc. Natl. Acad. Sci. U.S.A. 93 (17): 9188–93. Bibcode:1996PNAS...93.9188B. PMC 38617. PMID 8799176. doi:10.1073/pnas.93.17.9188.
- ↑ Elkins JG, Podar M, Graham DE, Goltsman E; Barry K; Koonin EV; Hugenholtz P; Kyrpides N; Wanner G; Richardson P; Keller M; Stetter KO (xuño de 2008). "A korarchaeal genome reveals insights into the evolution of the Archaea". Proc. Natl. Acad. Sci. U.S.A. 105 (23): 8102–7. Bibcode:2008PNAS..105.8102E. PMC 2430366. PMID 18535141. doi:10.1073/pnas.0801980105.
- ↑ Baker, B.J., Tyson, G.W., Webb, R.I., Flanagan, J., Hugenholtz, P. and Banfield, J.F. (2006). "Lineages of acidophilic Archaea revealed by community genomic analysis. Science". Science 314 (6884): 1933–1935. Bibcode:2006Sci...314.1933B. PMID 17185602. doi:10.1126/science.1132690.
- ↑ Baker BJ, Comolli LR, Dick GJ; et al. (maio de 2010). "Enigmatic, ultrasmall, uncultivated Archaea". Proc. Natl. Acad. Sci. U.S.A. 107 (19): 8806–11. Bibcode:2010PNAS..107.8806B. PMC 2889320. PMID 20421484. doi:10.1073/pnas.0914470107.
- ↑ "Classification of domains and phyla". LPSN. Consultado o 27 de xuño de 2015.. Na LPSN distínguense 5 filos de arqueas: Euryarchaeota, Crenarchaeota, Korarchaeota, Nanoarchaeota e Thaumarchaeota.
- ↑ Takuro Nunoura; et al. (2011). "Insights into the evolution of Archaea and eukaryotic protein modifier systems revealed by the genome of a novel archaeal group.". Nucleic Acids Research (39(8)): 3204–3223.
- ↑ Guy L, Ettema TJ (decembro de 2011). "The archaeal 'TACK' superphylum and the origin of eukaryotes". Trends in Microbiology 19 (12): 580–87. PMID 22018741. doi:10.1016/j.tim.2011.09.002.
- ↑ Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, Bäckström D, Juzokaite L, Vancaester E, et al. (xaneiro de 2017). "Asgard archaea illuminate the origin of eukaryotic cellular complexity" (PDF). Nature 541 (7637): 353–58. Bibcode:2017Natur.541..353Z. OSTI 1580084. PMID 28077874. doi:10.1038/nature21031.
- ↑ Nina Dombrowski, Jun-Hoe Lee, Tom A Williams, Pierre Offre, Anja Spang (2019). Genomic diversity, lifestyles and evolutionary origins of DPANN archaea. Nature.
- ↑ Petitjean C, Deschamps P, López-García P, Moreira D (decembro de 2014). "Rooting the domain archaea by phylogenomic analysis supports the foundation of the new kingdom Proteoarchaeota". Genome Biology and Evolution 7 (1): 191–204. PMC 4316627. PMID 25527841. doi:10.1093/gbe/evu274.
- ↑ "NCBI taxonomy page on Archaea".
- ↑ Christian Rinke; et al. (2013). "Figure 2: Maximum-likelihood phylogenetic inference of Archaea and Bacteria". Nature (499): 431–437. doi:10.1038/nature12352.
- ↑ TACK: acrónimo de Thaumarchaeota, Aigarchaeota, Crenarchaeota, Korarchaeota.
- ↑ DPANN: acrónimo de Diapherotrites, Parvarchaeota, Aenigmaarchaeota, Nanoarchaeota, Nanohaloarchaeota.
- ↑ 37,0 37,1 Williams TA, Szöllősi GJ, Spang A, Foster PG, Heaps SE, Boussau B, et al. (xuño de 2017). "Integrative modeling of gene and genome evolution roots the archaeal tree of life". Proceedings of the National Academy of Sciences of the United States of America 114 (23): E4602–E4611. Bibcode:2017PNAS..114E4602W. PMC 5468678. PMID 28533395. doi:10.1073/pnas.1618463114.
- ↑ 38,0 38,1 Castelle CJ, Banfield JF (2018). "Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life". Cell 172 (6): 1181–1197. PMID 29522741. doi:10.1016/j.cell.2018.02.016.
- ↑ 39,0 39,1 "GTDB release 08-RS214". Genome Taxonomy Database. Consultado o 6 de decembro de 2021.
- ↑ 40,0 40,1 "ar53_r214.sp_label". Genome Taxonomy Database. Consultado o 10 de maio de 2023.
- ↑ 41,0 41,1 "Taxon History". Genome Taxonomy Database. Consultado o 6 de decembro de 2021.
- ↑ Seitz KW, Dombrowski N, Eme L, Spang A, Lombard J, Sieber JR, et al. (abril de 2019). "Asgard archaea capable of anaerobic hydrocarbon cycling". Nature Communications 10 (1): 1822. Bibcode:2019NatCo..10.1822S. PMC 6478937. PMID 31015394. doi:10.1038/s41467-019-09364-x.
- ↑ Eppley JM, Tyson GW, Getz WM, Banfield JF (2007). "Genetic exchange across a species boundary in the archaeal genus ferroplasma". Genetics 177 (1): 407–16. PMC 2013692. PMID 17603112. doi:10.1534/genetics.107.072892.
- ↑ Papke RT, Zhaxybayeva O, Feil EJ, Sommerfeld K, Muise D, Doolittle WF (2007). "Searching for species in haloarchaea". Proc. Natl. Acad. Sci. U.S.A. 104 (35): 14092–7. Bibcode:2007PNAS..10414092P. PMC 1955782. PMID 17715057. doi:10.1073/pnas.0706358104.
- ↑ Kunin V, Goldovsky L, Darzentas N, Ouzounis CA (2005). "The net of life: reconstructing the microbial phylogenetic network". Genome Res. 15 (7): 954–9. PMC 1172039. PMID 15965028. doi:10.1101/gr.3666505.
- ↑ Hugenholtz P (2002). "Exploring prokaryotic diversity in the genomic era". Genome Biol. 3 (2): REVIEWS0003. PMC 139013. PMID 11864374. doi:10.1186/gb-2002-3-2-reviews0003. Arquivado dende o orixinal o 15 de marzo de 2020. Consultado o 08 de xullo de 2012.
- ↑ Rappé MS, Giovannoni SJ (2003). "The uncultured microbial majority". Annu. Rev. Microbiol. 57: 369–94. PMID 14527284. doi:10.1146/annurev.micro.57.030502.090759.
- ↑ Schopf J (2006). "Fossil evidence of Archaean life". Philos Trans R Soc Lond B Biol Sci 361 (1470): 869–85. PMC 1578735. PMID 16754604. doi:10.1098/rstb.2006.1834.
- ↑ Chappe B, Albrecht P, Michaelis W (xullo de 1982). "Polar Lipids of Archaebacteria in Sediments and Petroleums". Science 217 (4554): 65–66. Bibcode:1982Sci...217...65C. PMID 17739984. doi:10.1126/science.217.4554.65.
- ↑ Brocks JJ, Logan GA, Buick R, Summons RE (1999). "Archean molecular fossils and the early rise of eukaryotes". Science 285 (5430): 1033–6. PMID 10446042. doi:10.1126/science.285.5430.1033.
- ↑ Rasmussen B, Fletcher IR, Brocks JJ, Kilburn MR (outubro de 2008). "Reassessing the first appearance of eukaryotes and cyanobacteria". Nature 455 (7216): 1101–4. Bibcode:2008Natur.455.1101R. PMID 18948954. doi:10.1038/nature07381.
- ↑ Hahn Jürgen, Pat Haug (1986). "Traces of Archaebacteria in ancient sediments". System Applied Microbiology 7 (Archaebacteria '85 Proceedings): 178–83.
- ↑ Wang M, Yafremava LS, Caetano-Anollés D, Mittenthal JE, Caetano-Anollés G (2007). "Reductive evolution of architectural repertoires in proteomes and the birth of the tripartite world". Genome Res. 17 (11): 1572–85. PMC 2045140. PMID 17908824. doi:10.1101/gr.6454307.
- ↑ Woese CR, Gupta R (1981). "Are archaebacteria merely derived 'prokaryotes'?". Nature 289 (5793): 95–6. Bibcode:1981Natur.289...95W. PMID 6161309. doi:10.1038/289095a0.
- ↑ 55,0 55,1 55,2 Woese C (1998). "The universal ancestor". Proc. Natl. Acad. Sci. U.S.A. 95 (12): 6854–9. Bibcode:1998PNAS...95.6854W. PMC 22660. PMID 9618502. doi:10.1073/pnas.95.12.6854.
- ↑ 56,0 56,1 Kandler O. The early diversification of life and the origin of the three domains: A proposal. In: Wiegel J, Adams WW, editors. Thermophiles: The keys to molecular evolution and the origin of life? Athens: Taylor and Francis, 1998: 19-31.
- ↑ Gribaldo S, Brochier-Armanet C (2006). "The origin and evolution of Archaea: a state of the art". Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1470): 1007–22. PMC 1578729. PMID 16754611. doi:10.1098/rstb.2006.1841. Arquivado dende o orixinal o 04 de xuño de 2012. Consultado o 08 de xullo de 2012.
- ↑ 58,0 58,1 58,2 Woese CR (marzo de 1994). "There must be a prokaryote somewhere: microbiology's search for itself". Microbiol. Rev. 58 (1): 1–9. PMC 372949. PMID 8177167.
- ↑ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P (2006). "Toward automatic reconstruction of a highly resolved tree of life". Science 311 (5765): 1283–7. Bibcode:2006Sci...311.1283C. PMID 16513982. doi:10.1126/science.1123061.
- ↑ A información procede de Willey, JM; Sherwood, LM; Woolverton, CJ (2008). "19". "Microbiology" (7ª ed.). pp. 474–475., excepto as partes que teñen notas.
- ↑ Talbert PB, Henikoff S (2010). "Histone variants – ancient wrap artists of the epigenome". Nature Reviews Molecular Cell Biology 11: 264–275. doi:10.1038/nrm2861.
- ↑ Sandman K, Reeve JN (2006). "Archaeal histones and the origin of the histone fold". Curr. Opin. Microbiol 9: 520–525. doi:10.1016/j.mib.2006.08.003.
- ↑ 63,0 63,1 63,2 63,3 63,4 Zillig W (1991). "Comparative biochemistry of Archaea and Bacteria". Curr. Opin. Genet. Dev. 1 (4): 544–51. PMID 1822288. doi:10.1016/S0959-437X(05)80206-0.
- ↑ Bell SD, Jackson SP (abril de 2001). "Mechanism and regulation of transcription in archaea". Curr. Opin. Microbiol. 4 (2): 208–13. PMID 11282478. doi:10.1016/S1369-5274(00)00190-9.
- ↑ Reeve JN (maio de 2003). "Archaeal chromatin and transcription". Mol. Microbiol. 48 (3): 587–98. PMID 12694606.
- ↑ Kelman LM, Kelman Z (maio de 2003). "Archaea: an archetype for replication initiation studies?". Mol. Microbiol. 48 (3): 605–15. PMID 12694608.
- ↑ Phillips G, Chikwana VM, Maxwell A; et al. (abril de 2010). "Discovery and characterization of an amidinotransferase involved in the modification of archaeal tRNA". J. Biol. Chem. 285 (17): 12706–13. PMC 2857094. PMID 20129918. doi:10.1074/jbc.M110.102236.
- ↑ Koonin EV, Mushegian AR, Galperin MY, Walker DR. (1997). ""Comparison of archaeal and bacterial genomes: computer analysis of protein sequences predicts novel functions and suggests a chimeric origin for the archaea".". Mol Microbiol (25): 619–637.
- ↑ 69,0 69,1 69,2 69,3 69,4 Gupta R. S. (1998). "Protein phylogenies and signature sequences: A reappraisal of evolutionary relationships among archaebacteria, eubacteria, and eukaryotes". Microbiol. Mol. Biol. Rev 62: 1435–1491.
- ↑ Koch AL (2003). "Were Gram-positive rods the first bacteria?". Trends Microbiol (11(4)): 166–170.
- ↑ 71,0 71,1 71,2 Gupta R.S. (1998). "What are archaebacteria: life's third domain or monoderm prokaryotes related to gram-positive bacteria? A new proposal for the classification of prokaryotic organisms". Mol. Microbiol 29: 695–708.
- ↑ Brown JR, Masuchi Y, Robb FT, Doolittle WF. (1994). "Evolutionary relationships of bacterial and archaeal glutamine synthetase genes". J Mol Evol. (38(6)): 566–576.
- ↑ 73,0 73,1 73,2 Gupta, R. S.(2000) The natural evolutionary relationships among prokaryotes. Crit. Rev. Microbiol. 26: 111-131.
- ↑ Gupta, RS (2005). Oxford University Press, ed. Molecular Sequences and the Early History of Life. En: Sapp J, editor. "Microbial Phylogeny and Evolution: Concepts and Controversies". Nova York. pp. 160–183.
- ↑ Cavalier-Smith T. (2002). "The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification". Int J Syst Evol Microbiol (52(Pt 1)): 7–76.
- ↑ Valas RE, Bourne PE (2011). "The origin of a derived superkingdom: how a Gram-positive bacterium crossed the desert to become an archaeon". Biol Direct (6): 16.
- ↑ Skophammer RG, Herbold CW, Rivera MC, Servin JA, Lake JA (2006). "Evidence that the root of the tree of life is not within the Archaea". Mol Biol Evol. (23(9)): 1648–1651.
- ↑ Lake JA (xaneiro de 1988). "Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences". Nature 331 (6152): 184–6. Bibcode:1988Natur.331..184L. PMID 3340165. doi:10.1038/331184a0.
- ↑ Nelson KE, Clayton RA, Gill SR, Utterback TR; Malek JA; Linher KD; Garrett MM; Stewart AM; Cotton MD; Pratt MS; Phillips CA; Richardson D; Heidelberg J; Sutton GG; Fleischmann RD; Eisen JA; White O; Salzberg SL; Smith HO; Venter JC; Fraser CM (1999). "Evidence for lateral gene transfer between Archaea and bacteria from genome sequence of Thermotoga maritima". Nature 399 (6734): 323–9. Bibcode:1999Natur.399..323N. PMID 10360571. doi:10.1038/20601.
- ↑ Gouy M, Li WH (1989). "Phylogenetic analysis based on rRNA sequences supports the archaebacterial rather than the eocyte tree". Nature 339 (6220): 145–7. Bibcode:1989Natur.339..145G. PMID 2497353. doi:10.1038/339145a0.
- ↑ Yutin N, Makarova KS, Mekhedov SL, Wolf YI, Koonin EV (2008). "The deep archaeal roots of eukaryotes". Mol. Biol. Evol. 25 (8): 1619–30. PMC 2464739. PMID 18463089. doi:10.1093/molbev/msn108. Arquivado dende o orixinal o 03 de maio de 2009. Consultado o 08 de xullo de 2012.
- ↑ Lake JA. (1988). "Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences". Nature 331 (6152): 184–6. Bibcode:1988Natur.331..184L. PMID 3340165. doi:10.1038/331184a0.
- ↑ Tom A. Williams; et al. (2012.). "A congruent phylogenomic signal places eukaryotes within the Archaea". Proc. R. Soc. (B rspb20121795).
- ↑ L. Guy & T. Ettema (2011). "The archaeal ‘TACK’ superphylum and the origin of eukaryotes". Sciencedirect 19 (12): 580–587.
- ↑ Woese C. R. & Gupta, R. (1981). "Are archaebacteria merely derived 'prokaryotes'?". Nature 289 (5793): 95–6. PMID 6161309. doi:10.1038/289095a0.
- ↑ Woese C (1998). "The universal ancestor". Proc. Natl. Acad. Sci. U.S.A. 95 (12): 6854–9. PMID 9618502. doi:10.1073/pnas.95.12.6854.
- ↑ Gupta RS (2000). "The natural evolutionary relationships among prokaryotes". Crit. Rev. Microbiol. 26 (2): 111–31. PMID 10890353. doi:10.1080/10408410091154219.
- ↑ 88,0 88,1 88,2 88,3 Krieg, Noel (2005). Springer, ed. Bergey's Manual of Systematic Bacteriology. US. pp. 21–6. ISBN 978-0-387-24143-2.
- ↑ Barns, Sue and Burggraf, Siegfried. (1997) "Crenarchaeota" Arquivado 02 de maio de 2012 en Wayback Machine.. Versión do 01 de xaneiro de 1997. en "The Tree of Life Web Project"
- ↑ Walsby, A.E. (1980). "A square bacterium". Nature 283 (5742): 69–71. Bibcode:1980Natur.283...69W. doi:10.1038/283069a0.
- ↑ Hara F, Yamashiro K, Nemoto N; et al. (2007). "An actin homolog of the archaeon Thermoplasma acidophilum that retains the ancient characteristics of eukaryotic actin". J. Bacteriol. 189 (5): 2039–45. PMC 1855749. PMID 17189356. doi:10.1128/JB.01454-06. Arquivado dende o orixinal o 27 de maio de 2020. Consultado o 08 de xullo de 2012.
- ↑ Trent JD, Kagawa HK, Yaoi T, Olle E, Zaluzec NJ (1997). "Chaperonin filaments: the archaeal cytoskeleton?". Proc. Natl. Acad. Sci. U.S.A. 94 (10): 5383–8. Bibcode:1997PNAS...94.5383T. PMC 24687. PMID 9144246. doi:10.1073/pnas.94.10.5383. Arquivado dende o orixinal o 27 de maio de 2020. Consultado o 08 de xullo de 2012.
- ↑ Hixon WG, Searcy DG (1993). "Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts". BioSystems 29 (2–3): 151–60. PMID 8374067. doi:10.1016/0303-2647(93)90091-P.
- ↑ 94,0 94,1 Golyshina OV, Pivovarova TA, Karavaiko GI; et al. (2000). "Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea". Int. J. Syst. Evol. Microbiol. 50 (3): 997–1006. PMID 10843038.
- ↑ Hall-Stoodley L, Costerton JW, Stoodley P (2004). "Bacterial biofilms: from the natural environment to infectious diseases". Nat. Rev. Microbiol. 2 (2): 95–108. PMID 15040259. doi:10.1038/nrmicro821.
- ↑ Kuwabara T, Minaba M, Iwayama Y, Kamekura M. (novembro de 2005). "Thermococcus coalescens sp. nov., a cell-fusing hyperthermophilic archaeon from Suiyo Seamount". Int. J. Syst. Evol. Microbiol. 55 (Pt 6): 2507–14. PMID 16280518. doi:10.1099/ijs.0.63432-0.
- ↑ Nickell S, Hegerl R, Baumeister W, Rachel R (2003). "Pyrodictium cannulae enter the periplasmic space but do not enter the cytoplasm, as revealed by cryo-electron tomography". J. Struct. Biol. 141 (1): 34–42. PMID 12576018. doi:10.1016/S1047-8477(02)00581-6.
- ↑ Horn C, Paulmann B, Kerlen G, Junker N, Huber H (agosto de 1999). "In vivo observation of cell division of anaerobic hyperthermophiles by using a high-intensity dark-field microscope". J. Bacteriol. 181 (16): 5114–8. PMC 94007. PMID 10438790.
- ↑ Rudolph C, Wanner G, Huber R (2001). "Natural communities of novel archaea and bacteria growing in cold sulfurous springs with a string-of-pearls-like morphology". Appl. Environ. Microbiol. 67 (5): 2336–44. PMC 92875. PMID 11319120. doi:10.1128/AEM.67.5.2336-2344.2001.
- ↑ 100,0 100,1 Thomas NA, Bardy SL, Jarrell KF (2001). "The archaeal flagellum: a different kind of prokaryotic motility structure". FEMS Microbiol. Rev. 25 (2): 147–74. PMID 11250034. doi:10.1111/j.1574-6976.2001.tb00575.x.
- ↑ Rachel R, Wyschkony I, Riehl S, Huber H (2002). "The ultrastructure of Ignicoccus: evidence for a novel outer membrane and for intracellular vesicle budding in an archaeon" (PDF). Archaea 1 (1): 9–18. PMC 2685547. PMID 15803654. doi:10.1155/2002/307480. Arquivado dende o orixinal (PDF) o 24 de febreiro de 2009. Consultado o 08 de xullo de 2012.
- ↑ 102,0 102,1 Koga Y, Morii H (2007). "Biosynthesis of ether-type polar lipids in archaea and evolutionary considerations". Microbiol. Mol. Biol. Rev. 71 (1): 97–120. PMC 1847378. PMID 17347520. doi:10.1128/MMBR.00033-06.
- ↑ Albers SV, van de Vossenberg JL, Driessen AJ, Konings WN (2000). "Adaptations of the archaeal cell membrane to heat stress". Front. Biosci. 5: D813–20. PMID 10966867. doi:10.2741/albers.
- ↑ Damsté JS, Schouten S, Hopmans EC, van Duin AC, Geenevasen JA (2002). "Crenarchaeol: the characteristic core glycerol dibiphytanyl glycerol tetraether membrane lipid of cosmopolitan pelagic crenarchaeota". J. Lipid Res. 43 (10): 1641–51. PMID 12364548. doi:10.1194/jlr.M200148-JLR200.
- ↑ Koga Y, Morii H (2005). "Recent advances in structural research on ether lipids from archaea including comparative and physiological aspects". Biosci. Biotechnol. Biochem. 69 (11): 2019–34. PMID 16306681. doi:10.1271/bbb.69.2019. Arquivado dende o orixinal o 31 de decembro de 2008. Consultado o 08 de xullo de 2012.
- ↑ Hanford MJ, Peeples TL (2002). "Archaeal tetraether lipids: unique structures and applications". Appl. Biochem. Biotechnol. 97 (1): 45–62. PMID 11900115. doi:10.1385/ABAB:97:1:45.
- ↑ Macalady JL, Vestling MM, Baumler D, Boekelheide N, Kaspar CW, Banfield JF (2004). "Tetraether-linked membrane monolayers in Ferroplasma spp: a key to survival in acid". Extremophiles 8 (5): 411–9. PMID 15258835. doi:10.1007/s00792-004-0404-5.
- ↑ Sára M, Sleytr UB (2000). "S-Layer proteins". J. Bacteriol. 182 (4): 859–68. PMC 94357. PMID 10648507. doi:10.1128/JB.182.4.859-868.2000.
- ↑ Engelhardt H, Peters J (1998). "Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions". J Struct Biol 124 (2–3): 276–302. PMID 10049812. doi:10.1006/jsbi.1998.4070.
- ↑ 110,0 110,1 Kandler, O.; König, H. (1998-04). "Cell wall polymers in Archaea (Archaebacteria)". Cellular and Molecular Life Sciences CMLS (en inglés) 54 (4): 305–308. ISSN 1420-682X. doi:10.1007/s000180050156.
- ↑ Howland, John L. (2000). Oxford University Press, ed. The Surprising Archaea: Discovering Another Domain of Life. Oxford. pp. 32. ISBN 0-19-511183-4.
- ↑ Gophna U, Ron EZ, Graur D (2003). "Bacterial type III secretion systems are ancient and evolved by multiple horizontal-transfer events". Gene 312: 151–63. PMID 12909351. doi:10.1016/S0378-1119(03)00612-7.
- ↑ Nguyen L, Paulsen IT, Tchieu J, Hueck CJ, Saier MH (2000). "Phylogenetic analyses of the constituents of Type III protein secretion systems". J. Mol. Microbiol. Biotechnol. 2 (2): 125–44. PMID 10939240.
- ↑ Ng SY, Chaban B, Jarrell KF (2006). "Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications". J. Mol. Microbiol. Biotechnol. 11 (3–5): 167–91. PMID 16983194. doi:10.1159/000094053.
- ↑ Bardy SL, Ng SY, Jarrell KF (2003). "Prokaryotic motility structures". Microbiology (Reading, Engl.) 149 (Pt 2): 295–304. PMID 12624192. doi:10.1099/mic.0.25948-0.
- ↑ 116,0 116,1 116,2 Valentine DL (2007). "Adaptations to energy stress dictate the ecology and evolution of the Archaea". Nat. Rev. Microbiol. 5 (4): 316–23. PMID 17334387. doi:10.1038/nrmicro1619.
- ↑ 117,0 117,1 117,2 Schäfer G, Engelhard M, Müller V (1999). "Bioenergetics of the Archaea". Microbiol. Mol. Biol. Rev. 63 (3): 570–620. PMC 103747. PMID 10477309.
- ↑ Romano A, Conway T (1996). "Evolution of carbohydrate metabolic pathways". Res Microbiol 147 (6–7): 448–55. PMID 9084754. doi:10.1016/0923-2508(96)83998-2.
- ↑ Koch A (1998). "How did bacteria come to be?". Adv Microb Physiol 40: 353–99. PMID 9889982. doi:10.1016/S0065-2911(08)60135-6.
- ↑ DiMarco AA, Bobik TA, Wolfe RS (1990). "Unusual coenzymes of methanogenesis". Annu. Rev. Biochem. 59: 355–94. PMID 2115763. doi:10.1146/annurev.bi.59.070190.002035.
- ↑ Klocke M, Nettmann E, Bergmann I; et al. (2008). "Characterization of the methanogenic Archaea within two-phase biogas reactor systems operated with plant biomass". Syst. Appl. Microbiol. 31 (3): 190–205. PMID 18501543. doi:10.1016/j.syapm.2008.02.003.
- ↑ Baseado en PDB 1FBB Arquivado 03 de marzo de 2016 en Wayback Machine.. Información publicada en Subramaniam S, Henderson R (2000). "Molecular mechanism of vectorial proton translocation by bacteriorhodopsin". Nature 406 (6796): 653–7. PMID 10949309. doi:10.1038/35020614.
- ↑ Mueller-Cajar O, Badger MR (2007). "New roads lead to Rubisco in archaebacteria". BioEssays 29 (8): 722–4. PMID 17621634. doi:10.1002/bies.20616.
- ↑ Berg IA, Kockelkorn D, Buckel W, Fuchs G (2007). "A 3-hydroxypropionate/4-hydroxybutyrate autotrophic carbon dioxide assimilation pathway in Archaea". Science 318 (5857): 1782–6. Bibcode:2007Sci...318.1782B. PMID 18079405. doi:10.1126/science.1149976.
- ↑ Thauer RK (2007). "Microbiology. A fifth pathway of carbon fixation". Science 318 (5857): 1732–3. PMID 18079388. doi:10.1126/science.1152209.
- ↑ Bryant DA, Frigaard NU (2006). "Prokaryotic photosynthesis and phototrophy illuminated". Trends Microbiol. 14 (11): 488–96. PMID 16997562. doi:10.1016/j.tim.2006.09.001.
- ↑ Könneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA (2005). "Isolation of an autotrophic ammonia-oxidizing marine archaeon". Nature 437 (7058): 543–6. Bibcode:2005Natur.437..543K. PMID 16177789. doi:10.1038/nature03911.
- ↑ Francis CA, Beman JM, Kuypers MM (2007). "New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation". ISME J 1 (1): 19–27. PMID 18043610. doi:10.1038/ismej.2007.8.
- ↑ Lanyi JK (2004). "Bacteriorhodopsin". Annu. Rev. Physiol. 66: 665–88. PMID 14977418. doi:10.1146/annurev.physiol.66.032102.150049.
- ↑ 130,0 130,1 Allers T, Mevarech M (2005). "Archaeal genetics — the third way". Nat. Rev. Genet. 6 (1): 58–73. PMID 15630422. doi:10.1038/nrg1504.
- ↑ Galagan JE, Nusbaum C, Roy A, Allen N; Naylor J; Stange-Thomann N; Dearellano K; Johnson R; Linton L; Mcewan P; Mckernan K; Talamas J; Tirrell A; Ye W; Zimmer A; Barber RD; Cann I; Graham DE; Grahame DA; Guss AM; Hedderich R; Ingram-Smith C; Kuettner HC; Krzycki JA; Leigh JA; Li W; Liu J; Mukhopadhyay B; Reeve JN; Smith K; Springer TA; Umayam LA; White O; White RH; Conway De Macario E; Ferry JG; Jarrell KF; Jing H; Macario AJ; Paulsen I; Pritchett M; Sowers KR; Swanson RV; Zinder SH; Lander E; Metcalf WW; Birren B (2002). "The genome of M. acetivorans reveals extensive metabolic and physiological diversity". Genome Res. 12 (4): 532–42. PMC 187521. PMID 11932238. doi:10.1101/gr.223902.
- ↑ Waters E; Lin X; Mathur E; Ni J; Podar M; Richardson T; Sutton GG; Simon M; Soll D; Stetter KO; Short JM; Noordewier M (2003). "The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism". Proc. Natl. Acad. Sci. U.S.A. 100 (22): 12984–8. Bibcode:2003PNAS..10012984W. PMC 240731. PMID 14566062. doi:10.1073/pnas.1735403100.
- ↑ Schleper C, Holz I, Janekovic D, Murphy J, Zillig W (1995). "A multicopy plasmid of the extremely thermophilic archaeon Sulfolobus effects its transfer to recipients by mating". J. Bacteriol. 177 (15): 4417–26. PMC 177192. PMID 7635827. Arquivado dende o orixinal o 29 de maio de 2012. Consultado o 08 de xullo de 2012.
- ↑ Sota M; Top EM (2008). "Horizontal Gene Transfer Mediated by Plasmids". En Caister Academic Press. Plasmids: Current Research and Future Trends. ISBN 978-1-904455-35-6.
- ↑ Xiang X, Chen L, Huang X, Luo Y, She Q, Huang L (2005). "Sulfolobus tengchongensis spindle-shaped virus STSV1: virus-host interactions and genomic features". J. Virol. 79 (14): 8677–86. PMC 1168784. PMID 15994761. doi:10.1128/JVI.79.14.8677-8686.2005. Arquivado dende o orixinal o 16 de outubro de 2019. Consultado o 08 de xullo de 2012.
- ↑ Prangishvili D, Forterre P, Garrett RA (2006). "Viruses of the Archaea: a unifying view". Nat. Rev. Microbiol. 4 (11): 837–48. PMID 17041631. doi:10.1038/nrmicro1527.
- ↑ Prangishvili D, Garrett RA (2004). "Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses". Biochem. Soc. Trans. 32 (Pt 2): 204–8. PMID 15046572. doi:10.1042/BST0320204.
- ↑ Pietilä MK, Roine E, Paulin L, Kalkkinen N, Bamford DH (2009). "An ssDNA virus infecting archaea; A new lineage of viruses with a membrane envelope". Mol. Microbiol. 72 (2): 307–19. PMID 19298373. doi:10.1111/j.1365-2958.2009.06642.x.
- ↑ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (2005). "Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements". J. Mol. Evol. 60 (2): 174–82. PMID 15791728. doi:10.1007/s00239-004-0046-3.
- ↑ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (2006). "A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action". Biol. Direct 1: 7. PMC 1462988. PMID 16545108. doi:10.1186/1745-6150-1-7.
- ↑ Graham DE, Overbeek R, Olsen GJ, Woese CR (2000). "An archaeal genomic signature". Proc. Natl. Acad. Sci. U.S.A. 97 (7): 3304–8. Bibcode:2000PNAS...97.3304G. PMC 16234. PMID 10716711. doi:10.1073/pnas.050564797.
- ↑ 142,0 142,1 Gaasterland T (1999). "Archaeal genomics". Curr. Opin. Microbiol. 2 (5): 542–7. PMID 10508726. doi:10.1016/S1369-5274(99)00014-4.
- ↑ Werner F (2007). "Structure and function of archaeal RNA polymerases". Mol. Microbiol. 65 (6): 1395–404. PMID 17697097. doi:10.1111/j.1365-2958.2007.05876.x.
- ↑ Aravind L, Koonin EV (1999). "DNA-binding proteins and evolution of transcription regulation in the archaea". Nucleic Acids Res. 27 (23): 4658–70. PMC 148756. PMID 10556324. doi:10.1093/nar/27.23.4658.
- ↑ Lykke-Andersen J, Aagaard C, Semionenkov M, Garrett RA (1997). "Archaeal introns: splicing, intercellular mobility and evolution". Trends Biochem. Sci. 22 (9): 326–31. PMID 9301331. doi:10.1016/S0968-0004(97)01113-4.
- ↑ Watanabe Y, Yokobori S, Inaba T; et al. (2002). "Introns in protein-coding genes in Archaea". FEBS Lett. 510 (1–2): 27–30. PMID 11755525. doi:10.1016/S0014-5793(01)03219-7.
- ↑ Yoshinari S, Itoh T, Hallam SJ; et al. (2006). "Archaeal pre-mRNA splicing: a connection to hetero-oligomeric splicing endonuclease". Biochem. Biophys. Res. Commun. 346 (3): 1024–32. PMID 16781672. doi:10.1016/j.bbrc.2006.06.011.
- ↑ Rosenshine I, Tchelet R, Mevarech M. (1989). "The mechanism of DNA transfer in the mating system of an archaebacterium". Science (245(4924)): 1387–1389. PMID 2818746.
- ↑ 149,0 149,1 149,2 Fröls S, Ajon M, Wagner M, Teichmann D, Zolghadr B, Folea M, Boekema EJ, Driessen AJ, Schleper C, Albers SV. (2008). "UV-inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus is mediated by pili formation". Mol Microbiol (70(4)): 938–52. PMID 18990182. doi:10.1111/j.1365-2958.2008.06459.x.
- ↑ 150,0 150,1 150,2 Ajon M, Fröls S, van Wolferen M, Stoecker K, Teichmann D, Driessen AJ, Grogan DW, Albers SV, Schleper C (2011). "UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili". Mol Microbiol (82(4)): 807–17. PMID 21999488. doi:10.1111/j.1365-2958.2011.07861.x.
- ↑ Fröls S, White MF, Schleper C (2009). "Reactions to UV damage in the model archaeon Sulfolobus solfataricus". Biochem Soc Trans (37(1)): 36–41. PMID 19143598. doi:10.1042/BST0370036.
- ↑ Bernstein, H; Bernstein, C (2013). Dr. Carol Bernstein, In Tech., ed. Evolutionary Origin and Adaptive Function of Meiosis. ISBN 978-953-51-1197-9.
- ↑ 153,0 153,1 Bernander R (1998). "Archaea and the cell cycle". Mol. Microbiol. 29 (4): 955–61. PMID 9767564. doi:10.1046/j.1365-2958.1998.00956.x.
- ↑ Kelman LM, Kelman Z (2004). "Multiple origins of replication in archaea". Trends Microbiol. 12 (9): 399–401. PMID 15337158. doi:10.1016/j.tim.2004.07.001.
- ↑ Onyenwoke RU, Brill JA, Farahi K, Wiegel J (2004). "Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch ( Firmicutes)". Arch. Microbiol. 182 (2–3): 182–92. PMID 15340788. doi:10.1007/s00203-004-0696-y.
- ↑ Kostrikina NA, Zvyagintseva IS, Duda VI. (1991). "Cytological peculiarities of some extremely halophilic soil archaeobacteria". Arch. Microbiol. 156 (5): 344–49. doi:10.1007/BF00248708.
- ↑ DeLong EF, Pace NR (2001). "Environmental diversity of bacteria and archaea". Syst. Biol. 50 (4): 470–8. PMID 12116647. doi:10.1080/106351501750435040.
- ↑ 158,0 158,1 Pikuta EV, Hoover RB, Tang J (2007). "Microbial extremophiles at the limits of life". Crit. Rev. Microbiol. 33 (3): 183–209. PMID 17653987. doi:10.1080/10408410701451948.
- ↑ Madigan MT, Martino JM (2006). Pearson, ed. Brock Biology of Microorganisms (11th ed.). p. 136. ISBN 0-13-196893-9.
- ↑ Takai K, Nakamura K, Toki T, Tsunogai U, Miyazaki M, Miyazaki J, Hirayama H, Nakagawa S, Nunoura T, Horikoshi K (2008). "Cell proliferation at 122 °C and isotopically heavy CH4 production by a hyperthermophilic methanogen under high-pressure cultivation". Proc Natl Acad Sci USA 105 (31): 10949–54. Bibcode:2008PNAS..10510949T. PMC 2490668. PMID 18664583. doi:10.1073/pnas.0712334105.
- ↑ Ciaramella M, Napoli A, Rossi M (2005). "Another extreme genome: how to live at pH 0". Trends Microbiol. 13 (2): 49–51. PMID 15680761. doi:10.1016/j.tim.2004.12.001.
- ↑ Javaux EJ (2006). "Extreme life on Earth—past, present and possibly beyond". Res. Microbiol. 157 (1): 37–48. PMID 16376523. doi:10.1016/j.resmic.2005.07.008.
- ↑ Nealson KH (1999). "Post-Viking microbiology: new approaches, new data, new insights" (PDF). Orig Life Evol Biosph 29 (1): 73–93. PMID 11536899. doi:10.1023/A:1006515817767. Arquivado dende o orixinal (PDF) o 16 de outubro de 2019. Consultado o 08 de xullo de 2012.
- ↑ Davies PC (1996). "The transfer of viable microorganisms between planets". Ciba Found. Symp. 202: 304–14; discussion 314–7. PMID 9243022.
- ↑ López-García P, López-López A, Moreira D, Rodríguez-Valera F (2001). "Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front". FEMS Microbiol. Ecol. 36 (2–3): 193–202. PMID 11451524.
- ↑ Karner MB, DeLong EF, Karl DM (2001). "Archaeal dominance in the mesopelagic zone of the Pacific Ocean". Nature 409 (6819): 507–10. PMID 11206545. doi:10.1038/35054051.
- ↑ Giovannoni SJ, Stingl U. (2005). "Molecular diversity and ecology of microbial plankton". Nature 427 (7057): 343–8. Bibcode:2005Natur.437..343G. PMID 16163344. doi:10.1038/nature04158.
- ↑ DeLong EF, Karl DM (2005). "Genomic perspectives in microbial oceanography". Nature 437 (7057): 336–42. Bibcode:2005Natur.437..336D. PMID 16163343. doi:10.1038/nature04157.
- ↑ Konneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA. (2005). "Isolation of an autotrophic ammonia-oxidizing marine archaeon". Nature 437 (7057): 543–6. Bibcode:2005Natur.437..543K. PMID 16177789. doi:10.1038/nature03911.
- ↑ Agogué H; Brink M; Maaike B; Dinasquet J; Herndl GJ (2008). "Major gradients in putatively nitrifying and non-nitrifying Archaea in the deep North Atlantic". Nature 456 (7223): 788–791. Bibcode:2008Natur.456..788A. PMID 19037244. doi:10.1038/nature07535.
- ↑ Teske A, Sørensen KB (2008). "Uncultured archaea in deep marine subsurface sediments: have we caught them all?". ISME J 2 (1): 3–18. PMID 18180743. doi:10.1038/ismej.2007.90.
- ↑ Lipp JS, Morono Y, Inagaki F, Hinrichs KU (2008). "Significant contribution of Archaea to extant biomass in marine subsurface sediments". Nature 454 (7207): 991–4. Bibcode:2008Natur.454..991L. PMID 18641632. doi:10.1038/nature07174.
- ↑ Cabello P, Roldán MD, Moreno-Vivián C (2004). "Nitrate reduction and the nitrogen cycle in archaea". Microbiology (Reading, Engl.) 150 (Pt 11): 3527–46. PMID 15528644. doi:10.1099/mic.0.27303-0. Arquivado dende o orixinal o 07 de xuño de 2011. Consultado o 08 de xullo de 2012.
- ↑ Mehta MP, Baross JA (2006). "Nitrogen fixation at 92 degrees C by a hydrothermal vent archaeon". Science 314 (5806): 1783–6. Bibcode:2006Sci...314.1783M. PMID 17170307. doi:10.1126/science.1134772.
- ↑ Francis CA, Beman JM, Kuypers MM (2007). "New processes and players in the nitrogen cycle: the microbial ecology of anaerobic and archaeal ammonia oxidation". ISME J 1 (1): 19–27. PMID 18043610. doi:10.1038/ismej.2007.8.
- ↑ Coolen MJ, Abbas B, van Bleijswijk J; et al. (2007). "Putative ammonia-oxidizing Crenarchaeota in suboxic waters of the Black Sea: a basin-wide ecological study using 16S ribosomal and functional genes and membrane lipids". Environ. Microbiol. 9 (4): 1001–16. PMID 17359272. doi:10.1111/j.1462-2920.2006.01227.x.
- ↑ Leininger S, Urich T, Schloter M; et al. (2006). "Archaea predominate among ammonia-oxidizing prokaryotes in soils". Nature 442 (7104): 806–9. Bibcode:2006Natur.442..806L. PMID 16915287. doi:10.1038/nature04983.
- ↑ Baker, B. J ; Banfield, J. F (2003). "Microbial communities in acid mine drainage". FEMS Microbiology Ecology 44 (2): 139–152. PMID 19719632. doi:10.1016/S0168-6496(03)00028-X.
- ↑ "EDGAR 3.2 Fast Track 2000". Arquivado dende o orixinal o 21 de maio de 2008. Consultado o 2008-06-26.
- ↑ "Annual Greenhouse Gas Index (AGGI) Indicates Sharp Rise in Carbon Dioxide and Methane in 2007". 2008-04-23. Arquivado dende o orixinal o 14 de maio de 2008. Consultado o 2008-06-26.
- ↑ United Nations Environment Programme (ed.). "Trace Gases: Current Observations, Trends, and Budgets". Climate Change 2001. Arquivado dende o orixinal o 05 de agosto de 2012. Consultado o 08 de xullo de 2012.
- ↑ Eckburg P, Lepp P, Relman D (2003). "Archaea and their potential role in human disease". Infect Immun 71 (2): 591–6. PMC 145348. PMID 12540534. doi:10.1128/IAI.71.2.591-596.2003.
- ↑ Cavicchioli R, Curmi P, Saunders N, Thomas T (2003). "Pathogenic archaea: do they exist?". BioEssays 25 (11): 1119–28. PMID 14579252. doi:10.1002/bies.10354.
- ↑ Lepp P, Brinig M, Ouverney C, Palm K, Armitage G, Relman D (2004). "Methanogenic Archaea and human periodontal disease". Proc Natl Acad Sci USA 101 (16): 6176–81. Bibcode:2004PNAS..101.6176L. PMC 395942. PMID 15067114. doi:10.1073/pnas.0308766101.
- ↑ Vianna ME, Conrads G, Gomes BP, Horz HP (2006). "Identification and quantification of archaea involved in primary endodontic infections". J. Clin. Microbiol. 44 (4): 1274–82. PMC 1448633. PMID 16597851. doi:10.1128/JCM.44.4.1274-1282.2006. Arquivado dende o orixinal o 16 de outubro de 2019. Consultado o 08 de xullo de 2012.
- ↑ Waters E; Hohn MJ; Ahel I; Lin X; Mathur E; Ni J; Podar M; Richardson T; Sutton GG; Simon M; Soll D; Stetter KO; Short JM; Noordewier M (outubro de 2003). "The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism". Proc. Natl. Acad. Sci. U.S.A. 100 (22): 12984–8. Bibcode:2003PNAS..10012984W. PMC 240731. PMID 14566062. doi:10.1073/pnas.1735403100.
- ↑ Jahn U, Gallenberger M, Paper W; et al. (2008). "Nanoarchaeum equitans and Ignicoccus hospitalis: new insights into a unique, intimate association of two archaea". J. Bacteriol. 190 (5): 1743–50. PMC 2258681. PMID 18165302. doi:10.1128/JB.01731-07. Arquivado dende o orixinal o 27 de maio de 2020. Consultado o 08 de xullo de 2012.
- ↑ Baker BJ, Comolli LR, Dick GJ, Hauser LJ, Hyatt D, Dill BD, Land ML, VerBerkmoes NC, Hettich RL, Banfield JF (2010). "Enigmatic, ultrasmall, uncultivated Archaeaa". Proc. Natl. Acad. Sci. U.S.A. 107 (19): 8806–8811. PMC 2889320. PMID 20421484. doi:10.1073/pnas.0914470107. Arquivado dende o orixinal o 10 de febreiro de 2021. Consultado o 08 de xullo de 2012.
- ↑ Chaban B, Ng SY, Jarrell KF (2006). "Archaeal habitats—from the extreme to the ordinary". Can. J. Microbiol. 52 (2): 73–116. PMID 16541146. doi:10.1139/w05-147.
- ↑ Schink B (1997). "Energetics of syntrophic cooperation in methanogenic degradation". Microbiol. Mol. Biol. Rev. 61 (2): 262–80. PMC 232610. PMID 9184013.
- ↑ Lange M; Westermann P; Ahring BK; Lange, M; Westermann, P; Ahring B.K. (2005). "Archaea in protozoa and metazoa". Applied Microbiology and Biotechnology 66 (5): 465–474. PMID 15630514. doi:10.1007/s00253-004-1790-4.
- ↑ van Hoek AH, van Alen TA, Sprakel VS; et al. (2000). "Multiple acquisition of methanogenic archaeal symbionts by anaerobic ciliates". Mol. Biol. Evol. 17 (2): 251–8. PMID 10677847.
- ↑ Preston, C.M.; Wu, K.Y.; Molinski, T.F.; Delong, E.F. (1996). "A psychrophilic crenarchaeon inhabits a marine sponge: Cenarchaeum symbiosum gen. nov., sp. nov". Proc Natl Acad Sci USA 93 (13): 6241–6. Bibcode:1996PNAS...93.6241P. PMC 39006. PMID 8692799. doi:10.1073/pnas.93.13.6241.
- ↑ Eckburg PB, Bik EM, Bernstein CN; et al. (2005). "Diversity of the human intestinal microbial flora". Science 308 (5728): 1635–8. Bibcode:2005Sci...308.1635E. PMC 1395357. PMID 15831718. doi:10.1126/science.1110591.
- ↑ Samuel BS, Gordon JI (2006). "A humanized gnotobiotic mouse model of host-archaeal-bacterial mutualism". Proc. Natl. Acad. Sci. U.S.A. 103 (26): 10011–6. Bibcode:2006PNAS..10310011S. PMC 1479766. PMID 16782812. doi:10.1073/pnas.0602187103.
- ↑ Wegley, L.; Yu, Y.; Breitbart, M.; Casas, V,; Kline, D.I.; Rohwer, F.; Wegley, L. (2004). "Coral-associated Archaea" (PDF). Marine Ecology Progress Series 273: 89–96. doi:10.3354/meps273089. Arquivado dende o orixinal (PDF) o 11 de setembro de 2008. Consultado o 08 de xullo de 2012.
- ↑ Chelius MK, Triplett EW (2001). "The Diversity of Archaea and Bacteria in Association with the Roots of Zea mays L". Microb. Ecol. 41 (3): 252–63. PMID 11391463. doi:10.1007/s002480000087.
- ↑ Simon HM, Dodsworth JA, Goodman RM (2000). "Crenarchaeota colonize terrestrial plant roots". Environ. Microbiol. 2 (5): 495–505. PMID 11233158. doi:10.1046/j.1462-2920.2000.00131.x.
- ↑ Breithaupt H (2001). "The hunt for living gold. The search for organisms in extreme environments yields useful enzymes for industry". EMBO Rep. 2 (11): 968–71. PMC 1084137. PMID 11713183. doi:10.1093/embo-reports/kve238.
- ↑ 200,0 200,1 Egorova K, Antranikian G (2005). "Industrial relevance of thermophilic Archaea". Curr. Opin. Microbiol. 8 (6): 649–55. PMID 16257257. doi:10.1016/j.mib.2005.10.015.
- ↑ Synowiecki J, Grzybowska B, Zdziebło A (2006). "Sources, properties and suitability of new thermostable enzymes in food processing". Crit Rev Food Sci Nutr 46 (3): 197–205. PMID 16527752. doi:10.1080/10408690590957296.
- ↑ Jenney FE, Adams MW (2008). "The impact of extremophiles on structural genomics (and vice versa)". Extremophiles 12 (1): 39–50. PMID 17563834. doi:10.1007/s00792-007-0087-9.
- ↑ Norris PR, Burton NP, Foulis NA (2000). "Acidophiles in bioreactor mineral processing". Extremophiles 4 (2): 71–6. PMID 10805560. doi:10.1007/s007920050139.
- ↑ O'Connor EM, Shand RF (2002). "Halocins and sulfolobicins: the emerging story of archaeal protein and peptide antibiotics". J. Ind. Microbiol. Biotechnol. 28 (1): 23–31. PMID 11938468. doi:10.1038/sj/jim/7000190.
- ↑ Shand, RF; Leyva KJ (2008). "Archaeal Antimicrobials: An Undiscovered Country". En Blum P (ed.). Caister Academic Press. Archaea: New Models for Prokaryotic Biology. ISBN 978-1-904455-27-1.
Véxase tamén[editar | editar a fonte]
| Wikimedia Commons ten máis contidos multimedia na categoría: Arqueas |
| Vexa a entrada do Galizionario acerca de Arquea |
| Wikispecies posúe unha páxina sobre: Arqueas |

Bibliografía[editar | editar a fonte]
- Howland, John L. (2000). The Surprising Archaea: Discovering Another Domain of Life (en inglés). Oxford University Press. ISBN 0-19-511183-4.
- Martinko, JM; Madigan, MT (2005). Brock Biology of Microorganisms (en inglés) (11th ed.). Englewood Cliffs, N.J: Prentice Hall. ISBN 0-13-144329-1.
- Garrett, RA; Klenk, H (2005). Archaea: Evolution, Physiology and Molecular Biology (en inglés). WileyBlackwell. ISBN 1-4051-4404-1.
- Cavicchioli, R (2007). Archaea: Molecular and Cellular Biology (en inglés). American Society for Microbiology. ISBN 1-55581-391-7.
- Blum, P, ed. (2008). Archaea: New Models for Prokaryotic Biology (en inglés). Caister Academic Press. ISBN 978-1-904455-27-1.
- Lipps, G (2008). "Archaeal Plasmids". Plasmids: Current Research and Future Trends (en inglés). Caister Academic Press. ISBN 978-1-904455-35-6.
- Sapp, Jan (2009). Oxford University Press, ed. The New Foundations of Evolution. On the Tree of Life (en inglés). Nova York. ISBN 0-19-538850-X.
- Schaechter, M (2009). Archaea (Overview) in The Desk Encyclopedia of Microbiology (en inglés) (2nd ed.). San Diego/ Londres: Elsevier Academic Press. ISBN 978-0-12-374980-2.
Outros artigos[editar | editar a fonte]
- Célula procariótica
- Bacterias
- Ligamenvirales, virus que infectan arqueas
Ligazóns externas[editar | editar a fonte]
- "Introduction to the Archaea". UCMP (en inglés).
- "Archaea". NCBI (en inglés).
- "List of Prokaryotic names with Standing in Nomenclature" (en inglés).
- "Tree of Life". Tellapallet (en inglés). Arquivado dende o orixinal o 10 de febreiro de 2015. Consultado o 08 de xullo de 2012.
- Robert Sanders. "Shotgun sequencing finds nanoorganisms". UCBerkely News (en inglés). Descubrimento do grupo ARMAN de arqueas.
- "UCSC Archaeal Genome Browser" (en inglés).
