Expresión xénica

A expresión xénica é o proceso por medio do cal a información dun xene se utiliza para sintetizar un produto xénico funcional. Estes produtos son moitas veces proteínas, pero tamén existen xenes que non codifican proteínas, como os xenes dos ARN ribosómicos, ARN transferentes ou ARN nucleares pequenos, nos que o produto sintetizado é un ARN funcional. O proceso de expresión xénica utilízano todas as formas de vida coñecidas: eucariotas (tanto os unicelulares coma os pluricelulares), procariotas (bacterias e arqueas), para xerar as macromoléculas necesarias para a vida. Poden modularse varias fases do proceso da expresión xénica, como a transcrición, o splicing de ARN, a tradución, e a modificación postraducional dunha proteína. A regulación da expresión xénica dálle á célula o control sobre a súa estrutura e funcións, e é a base para a diferenciación celular, morfoxénese e a versatilidade e adaptabilidade dun organismo. A regulación xénica pode tamén servir como substrato para o cambio evolutivo, xa que o control do momento, localización, e cantidade de expresión dun xene pode ter un profundo efecto nas funcións (accións) do xene na célula ou nun organismo pluricelular.
En xenética, a expresión xénica é o nivel máis fundamental no que o xenotipo orixina o fenotipo. O código xenético do ADN é "interpretado" por medio da expresión xénica, e as propiedades da expresión dan lugar ao fenotipo do organismo. Os fenotipos a miúdo exprésanse pola síntese de proteínas que controlan a forma do organismo, ou que actúan como encimas que catalizan vías metabólicas específicas características de cada organismo.
Mecanismo
[editar | editar a fonte]Transcrición
[editar | editar a fonte]
- Artigo principal: Transcrición xenética.
Un xene é un tramo de ADN que codifica información. O ADN xenómico consta de dúas cadeas antiparalelas e complementarias en bases nitroxenadas, cada unha cos seus extremos 5' e 3'. Con respecto a un xene, as dúas cadeas poden denominarse "cadea molde," que serve como molde para a produción do transcrito de ARN, e a "cadea codificante," que inclúe a versión do ADN igual á do transcrito de ARN (pero con T en vez de U). A produción de copìas de ARN a partir do ADN chámase transcrición xenética, e realízaa a ARN polimerase, que engade un por un nucleótidos de ARN á cadea de ARN en crecemento. Este ARN é complementario da cadea molde 3' → 5' de ADN,[1] a cal é complementaria da cadea codificante 5' → 3' do ADN. Por tanto, a cadea de ARN 5' → 3' resultante é idéntica á cadea codificante coa excepción de que a timina (T) está substituída por uracilo (U) no ARN. Unha cadea codificante de ADN coa secuencia "ATG" pasará indirectamente a súa información ao ARN ao facerse a transcrición da cadea non codificante (ou molde) como "AUG" no ARN.
A transcrición en procariotas lévaa a cabo un só tipo de ARN polimerase, que necesita unha secuencia de ADN chamada caixa de Pribnow e o factor sigma (σ) para empezar a transcrición. Nos eucariotas, a transcrición realízana tres tipos de ARN polimerases, cada unha das cales necesita unha secuencia especial no ADN chamada promotor e un conxunto de proteínas de unión ao ADN (factores de transcrición) para iniciar o proceso. A ARN polimerase I é a responsable da transcrición dos xenes do ARN ribosómico (ARNr). A ARN polimerase II (Pol II) transcribe todos os xenes codificantes de proteínas e tamén algúns non codificantes (como os dos ARN nucleares pequenos, ARN nucleolares pequenos ou ARN non codificantes longos). A Pol II inclúe un dominio C-terminal (CTD) que é rico en residuos de serina. Cando estes residuos son fosforilados, o dominio C-terminal únese a varios factores proteicos que promoven a maduración e modificación do transcrito. A ARN polimerase III transcribe os xenes dos ARNr 5S, os ARN transferentes (ARNt), e algúns pequenos ARNs non codificantes (como o 7SK). A transcrición remata cando a polimerase encontra unha secuencia chamada terminador.
Procesamento do ARN
[editar | editar a fonte]- Artigos principais: Modificación postranscricional e Splicing.
A transcrición en procariotas de xenes que codifican proteínas orixina ARNm que está listo para a súa tradución a proteínas, pero a transcrición nos eucariotas dá lugar a un transcrito primario de ARN (o ARNm precursor ou pre-ARNm), o cal primeiro sofre unha serie de modificacións para transformarse no ARNm que será traducido.
Estas modificacións inclúen a modificación do extremo 5' (proceso chamado capping), que consiste nun conxunto de reaccións encimáticas que engaden 7-metilguanosina (m7G) no extremo 5' do pre-ARNm, o que o protexe da degradación por exonucleases. Esta "carapucha" m7G (ou 5'-cap) únese despois a un complexo de unión á carapucha heterodímero (CBC20/CBC80), que axuda á exportación do ARNm ao citoplasma e ademais protexe ao ARN de perder a carapucha.
Outra modificación afecta ao extremo 3', que é clivado e poliadenilado. Ten lugar se está presente no pre-ARNm a secuencia sinal de poliadenilación (5'- AAUAAA-3'), que se encontra xeralmente entre a secuencia codificadora da proteína e o terminador do xene. Primeiro clívase (córtase) o pre-ARNm e despois engádense unha serie de ~200 bases adeninas (A) para formar a cola poli(A), que protexe o ARN da degradación. Á cola poli(A) únense moitas proteínas de unión á poli(A) (PABP) necesarias para a exportación do ARNm e a reiniciación da tradución.

Unha modificación moi importante do pre-ARNm eucariótico é o splicing do ARN. A maioría dos pre-ARNm eucarióticos consisten en segmentos alternantes codificantes e non codificantes chamados exóns e intróns. Durante o proceso de splicing, un complexo ARN-proteína catalítico chamado espliceosoma, cataliza dúas reaccións de transesterificación, as cales eliminan un intrón e libérano en forma dunha estrutura con forma de lazo ou bucle chamada lariat, e despois empalman xuntos os exóns veciños que limitaban co intrón eliminado. En certos casos, algúns intróns ou exóns poden ser eliminados ou retidos no ARNm maduro. Isto chámase splicing alternativo, e dá lugar a unha serie de transcritos diferentes orixinados todos a partir dun só xene. Como estes transcritos poden ser traducidos potencialmente a diferentes proteínas, o splicing aumenta a complexidade da expresión xénica eucariótica.
O procesamento do ARN pode ser unha vantaxe evolutiva feita posible grazas á existencia do núcleo nos eucariotas. Nos procariotas a transcrición e tradución ocorren á vez mentres que nos eucariotas a membrana nuclear separa os dous procesos dando tempo a que se produza o procesamento do ARN.
Maduración do ARN non codificante
[editar | editar a fonte]Na maioría dos organismos os xenes de ARN non codificante (ARNnc) transcríbense como precursores que sofren un procesamento ulterior. No caso do ARN ribosómico (ARNr), transcríbense a miúdo como un pre-ARNr que contén un ou máis ARNr; o pre-ARNr é clivado e modificado (por 2′-O-metilación e formación de pseudouridina) en sitios específicos por aproximadamente 150 especies de ARN nucleolares pequenos diferentes ou snoRNAs. Os snoRNAs asócianse con proteínas, formando ribonucleoproteínas (snoRNPs). Dese complexo, a parte formada polo snoRNA únese por complementariedade de bases co ARN diana e así sitúa a modificación no sitio preciso, e a parte formada pola proteína realiza a reacción catalítica. En eucariotas, certa snoRNP, chamada RNase MRP cliva o pre-ARNr de 45S nos ARNr de 28S, 5,8S, e 18S. O ARNr e os factores de procesamento do ARN forman grandes agregados que constitúen o nucléolo.[2]
No caso do ARN transferente (ARNt), por exemplo, a secuencia 5' é eliminada pola RNase P,[3] e o extremo 3' é eliminado polo encima tRNase Z [4] e unha nucleotidil transferase engade unha cola 3' CCA (non codificada no ADN molde).[5] No caso do micro ARN (miRNA), estes son primeiro transcritos como pri-miRNA cunha carapucha (cap) e unha cola poli-A e procesados para formar no núcleo celular estruturas de talo-lazo curtas de 70 nucleótidos chamadas pre-miRNA polos encimas Drosha e Pasha. Despois da súa exportación, son despois procesadas a miRNAs maduros no citoplasma ao interaccionaren coa enconuclease Dicer, a cal tamén inicia a formación do complexo RISC, composto pola proteína Argonauta.
Os propios ARN nucleares pequenos e ARN nucleolares pequenos sofren tamén unha serie de modificacións antes de que formen parte de complexos ribonucleoproteicos funcionais. Isto faise no nucleoplasma ou en estruturas especializadas chamadas corpos de Cajal. As súas bases son metiladas ou pseudouridiniladas por un grupo de ARNs pequenos específicos dos corpos de Cajal (scaRNAs), que son estruturalmente similares aos ARN nucleolares pequenos.
Exportación do ARN
[editar | editar a fonte]Nos eucariotas a maioría dos ARN maduros deben ser exportados ao citoplasma desde o núcleo da célula. Aínda que algúns ARNs funcionan no núcleo, moitos ARNs son transportados a través dos poros nucleares ao citosol. Entre os exportados están todos os tipos de ARN que están implicados na síntese de proteínas.[6] Nalgúns casos os ARNs son transportados adicionalmente a partes específicas do citoplasma, como as sinapses; son remorcados ata alí por proteínas motoras que se unen por medio de proteínas de enlace a secuencias específicas (chamadas "códigos cremalleira" ou zipcodes) do ARN.[7]
Tradución
[editar | editar a fonte]
- Artigo principal: Tradución (proteínas).
Para algúns ARN (o ARN non codificante) o ARN maduro é o produto xénico final.[8] No caso do ARN mensaxeiro o ARN é simplemente un transportador de información que codifica para a síntese dunha ou máis proteínas. O ARNm que leva a secuencia dunha soa proteína (común en eucariotas) é monocistrónico, e o que leva as secuencias de varias proteínas (común en procariotas) denomínase policistrónico.
Cada ARNm consta de tres partes: rexión non traducida 5' (5'UTR), rexión codificante da proteína (ou marco de lectura aberto, ORF) e rexión non traducida 3' (3'UTR). A rexión codificante leva a información para a síntese de proteínas codificada no código xenético en forma de tripletes de nucleótidos. Cada triplete de nucleótidos dunha rexión codificante chámase codón e corresponde cun sitio de unión complementario dun triplete anticodón situado no ARN transferente. Os ARNs transferentes coa mesma secuencia anticodón sempre levan o mesmo aminoácido. Os aminoácidos son despois unidos en cadea no ribosoma de acordo coa orde dos tripletes da rexión codificante. O ribosoma axuda ao ARN transferente a unirse ao ARN mensaxeiro, toma o aminoácido de cada ARN transferente e produce unha proteína aínda sen estrutura, que sae del.[9][10] Cada molécula de ARNm tradúcese moitas veces orixinando moitas copias da mesma proteína, como media unhas 900 nos mamíferos.[11]
Nos procariotas a tradución xeralmente ocorre no momento da transcrición (cotranscricionalmente), xa que normalmente se utiliza un ARNm que aínda está no proceso de transcrición (mentres non acabou aínda a transcrición por un dos extremos, xa se está a traducir polo outro extremo). Nos eucariotas a tradución pode ocorrer en varias rexións da célula dependendo de a onde está destinada a proteína que se debe traducir. As principais localizacións son o citoplasma para as proteínas citoplásmicas solubles e a membrana do retículo endoplasmático para as proteínas que van ser exportadas da célula ou inseridas na membrana plasmática. As proteínas que deben expresarse no retículo endoplasmático son recoñecidas cando a proteína en proceso de tradución está traducida no ribosoma só en parte. Isto está determinado pola partícula de recoñecemento do sinal, que é unha ribonucleoproteína que se une ao ribosoma e o dirixe ao retículo endoplasmático. Esta partícula recoñece unha secuencia sinal na cadea proteica en crecemento. Unha vez o ribosoma (coa proteína en crecemento) chega ao retículo endoplasmático, continúa alí ata o final o resto da síntese proteica.[12]
Pregamento
[editar | editar a fonte]- Artigo principal: Pregamento das proteínas.

O polipéptido prégase nunha estrutura tridimensional característica e funcional a partir da súa forma inicial con enrolamento aleatorio.[13] Cada proteína orixínase como un polipéptido non pregado cando é traducida dunha secuencia de ARNm. Este polipéptido carece de calquera estrutura tridimensional desenvolvida, mais os seus aminoácidos interaccionarán uns con outros e producirán unha estrutura tridimensional ben definida, que é a proteína pregada, e que se coñece como estado nativo. A estrutura tridimensional resultante está determinada pola secuencia de aminoácidos (dogma de Anfinsen).[14]
Que unha proteína teña unha correcta estrutura tridimensional é esencial para a súa función, aínda que algunhas partes de proteínas funcionais poden permanecer sen pregar.[15] Os fallos á hora de pregarse na forma axeitada producen xeralmente proteínas inactivas con diferentes propiedades, entre as que se inclúen os prións tóxicos. Pénsase que varias doenzas neurodexenerativas e doutros tipos se producen pola acumulación de proteínas incorrectamente pregadas.[16] Moitas alerxias son causadas polo pregamento das proteínas, e o sistema inmunitario non produce anticorpos para certas estruturas das proteínas.[17]
Os encimas denominados chaperonas axudan a que unha proteína recentemente formada se pregue na estrutura tridimensional necesaria para a súa función.[18] De xeito similar, as chaperonas do ARN axudan a que o ARN adopte a súa forma funcional.[19] A colaboración no pregamento das proteínas é unha das principais funcións do retículo endoplasmático nos eucariotas.
Transporte de proteínas
[editar | editar a fonte]Moitas proteínas non están destinadas a actuar no citosol, senón noutras partes da célula, e utilízase unha ampla variedade de secuencias de sinalización para dirixir ás proteínas ao seu destino. Nos procariotas este é normalmente un proceso simple debido á limitada compartimentalización das súas células. Porén, nos eucariotas hai unha gran variedade de procesos de selección (xeralmente por medio de secuencias diana e proteínas receptoras) que aseguran que a proteína chegue ao orgánulo correcto.
Non todas as proteínas permanecen dentro da célula, xa que moitas son exportadas fóra dela, como por exemplo os encimas dixestivos, hormonas e proteínas da matriz extracelular. Nos eucariotas a vía exportadora ou secretora está ben desenvolvida, e o mecanismo principal para a exportación destas proteínas é a translocación ao retículo endoplasmático, seguida do seu transporte desde o aparato de Golgi.[20][21]
Regulación da expresión xénica
[editar | editar a fonte]
- Artigo principal: Regulación da expresión xénica.
A regulación da expresión xénica é o control da cantidade e o momento en que se debe producir o produto funcional dun xene. O control da expresión é vital para que a célula poida formar os produtos que necesita nos momentos en que se necesitan, e dálle á célula flexibilidade para adaptarse a un ambiente variable, a sinais externos, a danos na célula etc. Algúns exemplos simples nos que é importante a expresión xénica son:
- Control da expresión da insulina para regular a glicosa sanguínea.
- Inactivación do cromosoma X nas femias de mamíferos para previr unha "sobredose" dos xenes que contén.
- Control da variación dos niveis de expresión de ciclinas ao longo do ciclo celular eucariótico.
A regulación xénica dálle á célula o control sobre todas as estruturas e funcións, e é a base da diferenciación celular, morfoxénese e a versatilidade e adaptabilidade dun organismo.
Poden modularse todos os pasos da expresión xenética, desde a transcrición ADN-ARN ata as modificacións postraducionais dunha proteína. A estabilidade do produto xénico final, xa sexa ARN ou proteína, tamén contribúe ao nivel de expresión dun xene, e un produto inestable dá lugar a baixos niveis de expresión. En xeral, a expresión xénica está regulada por medio de cambios [22] no número e tipo de interaccións entre moléculas[23] que en conxunto inflúen na transcrición do ADN [24] e na tradución do ARN.[25]
Utilízanse numerosos termos para describir os tipos de xenes dependendo de como estean regulados, entre os que están:
- Xene constitutivo. É un xene que se transcribe continuamente, a diferenza dun xene facultativo que só se transcribe cando se necesita nun momento dado.
- Xene de mantemento (housekeeping gene). É un xene constitutivo que se transcribe a un nivel relativamente constante. Os produtos dos xenes de mantemento necesítanse para o mantemento básico da célula. Asúmese xeralmente que a súa expresión non está afectada polas condicións experimentais. Exemplos son: actina, GAPDH e ubiquitina.
- Xene facultativo. É un xene que só se transcribe no momento que se necesita.
- Xene inducible. É un xene cuxa expresión responde aos cambios ambientais ou depende do ciclo celular.
Regulación transcricional
[editar | editar a fonte]A regulación da transcrición pode dividirse en tres rutas de influencia principais: xenética (interacción directa dun factor de control cun xene), modulación (interacción dun factor de control coa maquuinaria de transcrición) e epixenética (cambios na estrutura do ADN que non afectan á súa secuencia e teñen influencia na transcrición).

A interacción directa co ADN é o máis simple deles e o método máis directo polo cal unha proteína pode cambiar o seu nivel de transcrición. Os xenes a miúdo teñen varios sitios de unión arredor das rexións codificantes que teñen a función específica de regular a transcrición. Hai moitas clases de sitios de unión ao ADN regulatorios coñecidos como amplificadores (enhancers), illadores (insulators) e silenciadores. Os mecanismos para regular a transcrición son moi variados, e van desde bloquear os sitios de unión chaves no ADN para a ARN polimerase ata actuar como un activador e promover a transcrición axudando á unión da ARN polimerase.
A actividade dos factores de transcrición é ademais modulada por sinais intracelulares que causan modificacións postraducionais como a fosforilación, acetilación, ou glicosilación. Estes cambios inflúen na capacidade dos factores de transcrición de unirse, directa ou indirectamente, ao promotor do ADN, para recrutar a ARN polimerase, ou para favorecer a elongación dunha molécula de ARN en síntese.
A existencia da membrana nuclear nos eucariotas permite tamén a regulación dos factores de transcrición por medio da duración da súa presenza no núcleo, a cal é regulada por cambios reversibles na súa estrutura e pola unión doutras proteínas.[26] Os estímulos ambientais ou os sinais endócrinos [27] poden causar modificacións nas proteínas regulatorias [28] producindo cascadas de sinais intracelulares,[29] que dan lugar a unha regulación da expresión xénica.
Máis recentemente fíxose evidente que existe unha grande influencia sobre a tradución de efectos que non son específicos da secuencia do ADN. Estes efectos denomínanse epixenéticos e implican unha estrutura do ADN de maior orde, proteínas de unión ao ADN non específicas de secuencia e modificacións químicas do ADN. En xeral os efectos epixenéticos alteran a accesibilidade do ADN ás proteínas e así modulan a transcrición.

A metilación do ADN é un mecanismo epixenético moi estendido que inflúe na expresión xénica en bacterias e eucariotas e ten papeis no silenciamento da transcrición herdable e na regulación da transcrición. Nos eucariotas a estrutura da cromatina, controlada polo código de histonas, regula o acceso ao ADN con impactos significativos na expresión de xenes nas zonas de eucromatina e heterocromatina.
Regulación postranscricional
[editar | editar a fonte]- Artigo principal: Regulación postranscricional.
Nos eucariotas, nos que se require a exportación do ARN para que a tradución sexa posible, pénsase que a exportación desde o núcleo do ARN proporciona un control adicional sobre a expresión xénica. Todo o transporte cara a dentro e a fóra do núcleo faise a través dos poros nucleares e o transporte está controlado por unha gran variedade de proteínas importinas e exportinas.
A expresión dun xene que codifica unha proteína é só posible se o ARN mensaxeiro que que leva a información sobrevive o suficiente como para ser traducido. Nunha célula típica a molécula de ARN é só estable se está especificamente protexida contra a degradación. A degradación do ARN ten especial importancia na regulación da expresión nas células eucarióticas nas que o ARNm ten que viaxar distancias significativas antes de ser traducido. O ARN eucariótico é estabilizado por medio de certas modificacións postranscricionais, que afectan aos seus extremos, que son a colocación da "gorra" ou "carapucha" (5' cap) no extremo 5' e a cola poliA no extremo 3'.
A degradación deliberada do ARNm utilízase non só como un mecanismo de defensa contra a entrada na célula de ARN alleo (normalmente vírico), senón tamén como unha ruta de desestabilización do ARNm. Se unha molécula de ARNm ten unha secuencia complementaria á dun ARN interferente pequeno entón é destinado á destrución pola vía da interferencia de ARN.
Regulación traducional
[editar | editar a fonte]
- Artigo principal: Tradución (xenética).
A regulación directa da tradución é menos frecuente ca o control da transcrición ou da estabilidade do ARNm pero utilízase ocasionalmente. A inhibición da tradución de proteínas é un obxectivo principal para as toxinas e antibióticos para matar a unha célula ao anular o seu control da expresión xénica normal. Entre os inhibidores da síntese de proteínas están o antibiótico neomicina e a toxina ricina.
Degradación de proteínas
[editar | editar a fonte]- Artigo principal: Proteasoma.
Unha vez que a síntese proteica está completa o nivel de expresión desa proteína pode reducirse pola degradación da proteína. Hai vías de degradación das proteínas en todas as células procariotas e eucariotas, nas cales o proteasoma é un compoñente común. Unha proteína non necesaria ou danada é a miúdo etiquetada para a súa degradación ao engadirlle ubiquitina (ubiquitinilación).
Medida
[editar | editar a fonte]A medida da expresión xénica é unha parte importante de moitas ciencias da vida. A capacidade de cuantificar o nivel ao cal se expresa un determinado xene nunha célula, tecido ou organismo pode dar unha gran cantidade de información. Por exemplo, a medición da expresión dun xene pode:
- Identificar unha infección viral dunha célula (expresión de proteínas virais).
- Determinar a susceptibilidade dunha persoa ao cancro (expresión de oncoxenes).
- Comprobar se unha bacteria é resistente á penicilina (expresión da beta-lactamase).
De xeito similar, a análise da localización da proteína expresada é unha feramenta poderosa e pode realizarse a escala celular ou de organismo. A investigación da localización é particularmente importante para o estudo do desenvolvemento en organismos pluricelulares e como un indicador da función da proteína nunha célula. Idealmente a medida da expresión faise detectando o produto xénico final (para moitos xenes este é a proteína), aínda que con frecuencia é máis doado detectar algún dos precursores, xeralmente o ARNm, e inferir despois o nivel de expresión xénica.
Cuantificación do ARNm
[editar | editar a fonte]Os niveis do ARNm poden ser medidos cuantitativamente pola técnica northern blot, que indica o tamaño e información da secuencia de moléculas de ARNm. Unha mostra de ARN sepárase nun xel de agarosa e hibrídase cunha sonda de ARN marcado radioactivamente, que é complementaria da secuencia diana. O ARN marcado radioactivamente detéctase despois por unha autorradiografía. Como o uso de reactivos radioactivos fai que o procedemento leve moito tempo e sexa potencialmente perigoso, existen métodos alternativos de marcaxe e detección, como o uso de digoxixenina e biotina. As desvantaxes da northern blot son que se requiren grandes cantidades de ARN e que a cuantificación pode non ser moi precisa, xa que implica a medida da intensidade da banda nunha imaxe dun xel. Por outra parte, a información adicional do tamaño do ARNm que dá a northern blot permite a discriminación entre transcritos de splicing alternativo.
Outra aproximación para medir a abundancia dun ARNm é a RT-qPCR. Nesta técnica, á aplicación dunha transcrición inversa (RT) séguelle unha PCR cuantitativa en tempo real (qPCR). A transcrición inversa xera primeiro un molde de ADN a partir do ARNm; este molde monocatenario chámase ADNc. O molde de ADNc amplifícase despois na fase cuantitativa da técnica, durante a cal a fluorescencia emitida pola sonda de hibridación marcada ou colorantes intercalados cambia a medida que o proceso da amplificación do ADN progresa. Cunha curva estándar elaborada con coidado, a qPCR pode realizar unha medida absoluta do número de copias do ARNm orixinal, tipicamente en unidades de copias por nanolitro de tecido homoxenizado ou copias por célula. A qPCR é moi sensible (mesmo é teoricamente posible a detección dunha soa molécula de ARNm), pero pode ser cara dependendo do tipo de reporter usado; as sondas de oligonucleótidos marcados con fluorescencia son máis caras ca os colorantes fluorescentes intercalados non específicos.
Para realizar un perfil de expresión, ou análises de alto rendemento de moitos xenes nunha mostra, pode realizarse a RT-qPCR para centos de xenes simultaneamente no caso de matrices de baixa densidade. Unha segunda aproximación son as matrices de hibridación. Unha soa matriz ou "chip" pode conter mostras para determinar o nivel dos transcritos de case todos os xenes coñecidos dun xenoma dun ou máis organismos. Alternativamente, poden usarse as tecnoloxías baseadas en "etiquetados" como a análise en serie da expresión xénica (SAGE) e a RNA-Seq, as cales poden proporcionar unha medida relativa da concentración celular de diferentes ARNm. Unha vantaxe destes métodos é a "arquitectura aberta", que permite a medición exacta de calquera transcrito, sexa de secuencia coñecida ou descoñecida. As secuenciacións de nova xeración (Next-generation sequencing ou NGS) como a RNA-Seq son outra posible aproximación, que produce unha enorme cantidade de datos de secuencias que poden ser emparelladas co xenoma de referencia. Aínda que a realización dunha NGS leva comparativamente moito máis tempo, é máis cara e require o uso intensico de recursos, permite identificar polimorfismos dun só nucleótido, variantes de splicing, e novos xenes, e pode tamén utilizarse para obter o perfil de expresión dun organismo do cal se dispón de pouca un ningunha información de secuencias.
Cuantificación das proteínas
[editar | editar a fonte]O nivel de expresión xénica dos xenes que codifican proteínas pode estimarse directamente de varias formas, que teñen claras analoxías coas técnicas para a cuantificación do ARNm.
O método usado máis frecuentemente é aplicar a Western blot ás proteínas que interesan, o que dá información do tamaño da proteína ademais da súa identidade. Unha mostra (a miúdo un lisado celular) sepárase en xel de poliacrilamida, transfírese a unha membrana e despois sondéase cun anticorpo para a proteína procurada. O anticorpo pode ser conxugado cun fluoróforo ou coa peroxidase do ravo picante (Armoracia rusticana) para obter imaxes e cuantificación. A natureza deste ensaio baseada no xel fai que a cuantificación sexa menos precisa pero ten a vantaxe de que pode identificar posteriores modificacións da proteína, por exemplo proteólise ou ubiquitinación, a partir dos cambios no tamaño.
Localización
[editar | editar a fonte]- Artigos principais: Hibridación in situ e Inmunofluorescencia.

A análise da expresión non está limitado só á cuantificación; tamén se pode determinar a localización. O ARNm pode detectarse cunha cadea de ARNm complementario coa marcaxe axeitada e as proteínas poden detectarse por medio de anticorpos marcados. A mostra sondeada óllase despois con microscopio para identificar onde está a proteína ou o ARNm.

Ao substituír un xene por unha nova versión fusionada coa proteína fluorescente verde (ou outro marcador similar), a expresión pode ser cuantificada directamente nas células vivas. Isto faise obtendo imaxes cun microscopio de fluorescencia. É moi difícil clonar unha proteína fusionada coa proteína fluorescente verde na súa localización nativa no xenoma sen afectar aos niveis de expresión, polo que este método a miúdo non pode utilizarse para medir a expresión de xenes endóxenos. Porén, é amplamente usado para medir a expresión dun xene introducido artificialmente na célula, por exemplo por medio dun vector de expresión. É importante salientar que ao fusionar unha proteína diana cun compoñente fluorescente, o comportamento da proteína, incluíndo a súa localización celular e nivel de expresión, pode cambiar significativamente.
O ensaio de inmunoabsorción ligado a encima (ELISA) funciona utilizando antibióticos inmobilizados nunha placa microtituladora para capturar proteínas de interese de mostras engadidas ao pozo da placa. Utilizando para a detección un anticorpo conxugado a un encima ou fluoróforo, a cantidade de proteína que se une pode medirse con precisión por detección fluorimétrica ou colorimétrica. O proceso de detección é moi similar ao da Western blot, pero ao evitar os pasos nos que se usa o xel, pode atinguirse unha cuantificación máis precisa.
Sistema de expresión
[editar | editar a fonte]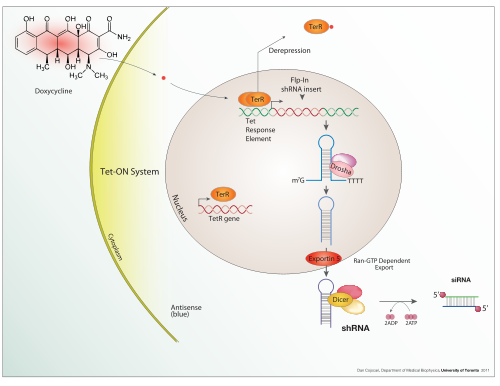
Un sistema de expresión é un sistema especificamente deseñado para a produción dun determinado produto xénico. Este é normalmente unha proteína aínda que pode ser tamén ARN, como o ARNt ou un ribozima. Un sistema de expresión consta dun xene, normalmente codificado por ADN, e a maquinaria molecular requirida para a transcrición do ADN a ARNm e a tradución do ARNm a proteína utilizando os reactivos proporcionados. En sentido amplo isto inclúe a todas as células vivas, pero o termo é normalmente utilizado para referirse á expresión como unha ferramenta de laboratorio. Un sistema de expresión é, pois, a miúdo artificial dalgunha maneira. Porén, os sistemas de expresión baséanse en procesos fundamentalmente naturais. Os virus son un excelente exemplo, xa que se replican utilizando a célula hóspede como un sistema de expresión para as proteínas do xenoma viral.
Expresión inducible
[editar | editar a fonte]A doxiciclina tamén se usa na activación transcricional controlada por tetraciclina "Tet-on" e "Tet-off" para regular a expresión de transxenes en organismos e cultivos celulares.
Na natureza
[editar | editar a fonte]Ademais destas ferramentas biolóxicas, na natureza obsérvanse certas configuracións do ADN (xenes, promotores, amplificadores, represores) e a propia maquinaria asociada, que se denominan sistema de expresión. Este termo utilízase normalmente no caso no que un xene ou conxunto de xenes é activado ou desactivado en condicións ben definidas. Por exemplo o sistema de expresión que funciona como un interruptor de represor simple do fago lambda e o sistema bacteriano do operador lac. Varios sistemas naturais de expresión son usados directamente ou modificados e usados en sistemas de expresión artificial, como o sistema de expresión Tet-on e Tet-off.
Redes de xenes
[editar | editar a fonte]Os xenes son considerados ás veces como os nós dunha rede, nos que as entradas (inputs) son as proteínas (como os factores de transcrición), e as saídas (outputs) son o nivel de expresión xénica. O propio nó realiza unha función, e a posta en operación destas funcións foi interpretada como a realización dun tipo de procesamento da información na célula e determina o comportamento celular.
As redes de xenes poden tamén construírse sen formular un modelo causal explícito. Este é normalmente o caso no que as redes se constrúen a partir de grandes conxuntos de datos de expresión. A covariación e correlación da expresión compútase a través dunha gran mostra de casos e medidas (a miúdo datos de transcriptoma ou proteoma). A fonte da variación pode ser experimental ou natural (observacional). Hai varios modos de construír redes de expresión xénica, pero unha aproximación común é computar unha matriz de todos os pares de correlacións da expresión a través de condicións, puntos de tempo, ou individuos e converter a matriz (despois de establecer un limiar en determinado valor de corte) nunha representación gráfica na cal os nodos representan xenes, transcritos, ou proteínas e os bordos que conectan estes nodos representan a forza da asociación (ver [1]).[30]
Técnicas e ferramentas
[editar | editar a fonte]As seguintes técnicas experimentais utilízanse para medir a expresión xénica e están listadas en orde aproximadamente cronolóxica, empezando polas tecnoloxías máis vellas e máis estabilizadas. Están divididas en dous grupos baseados no seu grao de multiplicidade das análises (ensaio múltiplex).
- Técnicas de grao baixo ou medio:
- Técnicas de grao alto:
- SAGE[32]
- micromatrices de ADN (microarrays)[33]
- Tiling array (un subtipo de microarrays)[34]
- RNA-Seq[35]
Notas
[editar | editar a fonte]- ↑ Brueckner F, Armache KJ, Cheung A; et al. (2009). "Structure–function studies of the RNA polymerase II elongation complex". Acta Crystallogr. D Biol. Crystallogr. 65 (Pt 2): 112–20. PMC 2631633. PMID 19171965. doi:10.1107/S0907444908039875.
- ↑ Sirri V, Urcuqui-Inchima S, Roussel P, Hernandez-Verdun D (2008). "Nucleolus: the fascinating nuclear body". Histochem. Cell Biol. 129 (1): 13–31. PMC 2137947. PMID 18046571. doi:10.1007/s00418-007-0359-6.
- ↑ Frank DN, Pace NR (1998). "Ribonuclease P: unity and diversity in a tRNA processing ribozyme". Annu. Rev. Biochem. 67: 153–80. PMID 9759486. doi:10.1146/annurev.biochem.67.1.153.
- ↑ Ceballos M, Vioque A (2007). "tRNase Z". Protein Pept. Lett. 14 (2): 137–45. PMID 17305600. doi:10.2174/092986607779816050.
- ↑ Weiner AM (2004). "tRNA maturation: RNA polymerization without a nucleic acid template". Curr. Biol. 14 (20): R883–5. PMID 15498478. doi:10.1016/j.cub.2004.09.069.
- ↑ Köhler A, Hurt E (2007). "Exporting RNA from the nucleus to the cytoplasm". Nat. Rev. Mol. Cell Biol. 8 (10): 761–73. PMID 17786152. doi:10.1038/nrm2255.
- ↑ Jambhekar A, Derisi JL (2007). "Cis-acting determinants of asymmetric, cytoplasmic RNA transport". RNA 13 (5): 625–42. PMC 1852811. PMID 17449729. doi:10.1261/rna.262607.
- ↑ Amaral PP, Dinger ME, Mercer TR, Mattick JS (2008). "The eukaryotic genome as an RNA machine". Science 319 (5871): 1787–9. PMID 18369136. doi:10.1126/science.1155472.
- ↑ Hansen TM, Baranov PV, Ivanov IP, Gesteland RF, Atkins JF (2003). "Maintenance of the correct open reading frame by the ribosome". EMBO Rep. 4 (5): 499–504. PMC 1319180. PMID 12717454. doi:10.1038/sj.embor.embor825.
- ↑ Berk V, Cate JH (2007). "Insights into protein biosynthesis from structures of bacterial ribosomes". Curr. Opin. Struct. Biol. 17 (3): 302–9. PMID 17574829. doi:10.1016/j.sbi.2007.05.009.
- ↑ Schwanhäusser B, Busse D, Dittmar G, Schuchhardt J, Wolf J, Chen W, Selbach M (2011). "Global quantification of mammalian gene expression control". Nature 473 (7347): 337–42. PMID 21593866. doi:10.1038/nature10098.
- ↑ Hegde RS, Kang SW (2008). "The concept of translocational regulation". J. Cell Biol. 182 (2): 225–32. PMC 2483521. PMID 18644895. doi:10.1083/jcb.200804157.
- ↑ Alberts, Bruce; Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters (2002). "The Shape and Structure of Proteins". Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 0-8153-3218-1.
- ↑ Anfinsen, C. (1972). "The formation and stabilization of protein structure". Biochem. J. 128 (4): 737–49. PMC 1173893. PMID 4565129.
- ↑ Jeremy M. Berg, John L. Tymoczko, Lubert Stryer; Web content by Neil D. Clarke (2002). "3. Protein Structure and Function". Biochemistry. San Francisco: W. H. Freeman. ISBN 0-7167-4684-0.
- ↑ Dennis J. Selkoe (2003). "Folding proteins in fatal ways". Nature 426 (6968): 900–904. PMID 14685251. doi:10.1038/nature02264.
- ↑ Alberts, Bruce, Dennis Bray, Karen Hopkin, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walter. "Protein Structure and Function." Essential Cell Biology. Edition 3. New York: Garland Science, Taylor and Francis Group, LLC, 2010. Pg 120-170.
- ↑ Hebert DN, Molinari M (2007). "In and out of the ER: protein folding, quality control, degradation, and related human diseases". Physiol. Rev. 87 (4): 1377–408. PMID 17928587. doi:10.1152/physrev.00050.2006.
- ↑ Russell R (2008). "RNA misfolding and the action of chaperones". Front. Biosci. 13 (13): 1–20. PMC 2610265. PMID 17981525. doi:10.2741/2557.
- ↑ Moreau P, Brandizzi F, Hanton S; et al. (2007). "The plant ER-Golgi interface: a highly structured and dynamic membrane complex". J. Exp. Bot. 58 (1): 49–64. PMID 16990376. doi:10.1093/jxb/erl135.
- ↑ Prudovsky I, Tarantini F, Landriscina M; et al. (2008). "Secretion Without Golgi". J. Cell. Biochem. 103 (5): 1327–43. PMC 2613191. PMID 17786931. doi:10.1002/jcb.21513.
- ↑ Zaidi SK, Young DW, Choi JY, Pratap J, Javed A, Montecino M, Stein JL, Lian JB, van Wijnen AJ, Stein GS (2004). "Intranuclear trafficking: organization and assembly of regulatory machinery for combinatorial biological control". J. Biol. Chem. 279 (42): 43363–6. PMID 15277516. doi:10.1074/jbc.R400020200.
- ↑ Mattick JS, Amaral PP, Dinger ME, Mercer TR, Mehler MF (2009). "RNA regulation of epigenetic processes". BioEssays 31 (1): 51–9. PMID 19154003. doi:10.1002/bies.080099.
- ↑ Martinez NJ, Walhout AJ (2009). "The interplay between transcription factors and microRNAs in genome-scale regulatory networks". BioEssays 31 (4): 435–45. PMC 3118512. PMID 19274664. doi:10.1002/bies.200800212.
- ↑ Tomilin NV (2008). "Regulation of mammalian gene expression by retroelements and non-coding tandem repeats". BioEssays 30 (4): 338–48. PMID 18348251. doi:10.1002/bies.20741.
- ↑ Veitia RA (2008). "One thousand and one ways of making functionally similar transcriptional enhancers". BioEssays 30 (11–12): 1052–7. PMID 18937349. doi:10.1002/bies.20849.
- ↑ Nguyen T, Nioi P, Pickett CB (2009). "The Nrf2-Antioxidant Response Element Signaling Pathway and Its Activation by Oxidative Stress". J. Biol. Chem. 284 (20): 13291–5. PMC 2679427. PMID 19182219. doi:10.1074/jbc.R900010200.
- ↑ Paul S (2008). "Dysfunction of the ubiquitin-proteasome system in multiple disease conditions: therapeutic approaches". BioEssays 30 (11–12): 1172–84. PMID 18937370. doi:10.1002/bies.20852.
- ↑ Los M, Maddika S, Erb B, Schulze-Osthoff K (2009). "Switching Akt: from survival signaling to deadly response". BioEssays 31 (5): 492–5. PMC 2954189. PMID 19319914. doi:10.1002/bies.200900005.
- ↑ Chesler EJ, Lu L, Wang J, Williams RW, Manly KF (2004). "WebQTL: rapid exploratory analysis of gene expression and genetic networks for brain and behavior". Nat Neurosci 7 (5): 485–86. PMID 15114364. doi:10.1038/nn0504-485.
- ↑ Song Y, Wang W, Qu X, Sun S (2009). "Effects of hypoxia inducible factor-1alpha (HIF-1alpha) on the growth & adhesion in tongue squamous cell carcinoma cells". Indian J. Med. Res. 129 (2): 154–63. PMID 19293442.
- ↑ Hanriot L, Keime C, Gay N; et al. (2008). "A combination of LongSAGE with Solexa sequencing is well suited to explore the depth and the complexity of transcriptome". BMC Genomics 9: 418. PMC 2562395. PMID 18796152. doi:10.1186/1471-2164-9-418.
- ↑ Wheelan SJ, Martínez Murillo F, Boeke JD (2008). "The incredible shrinking world of DNA microarrays". Mol Biosyst 4 (7): 726–32. PMC 2535915. PMID 18563246. doi:10.1039/b706237k.
- ↑ Miyakoshi M, Nishida H, Shintani M, Yamane H, Nojiri H (2009). "High-resolution mapping of plasmid transcriptomes in different host bacteria". BMC Genomics 10: 12. PMC 2642839. PMID 19134166. doi:10.1186/1471-2164-10-12.
- ↑ Denoeud F, Aury JM, Da Silva C, F; Artiguenave; et al. (2008). "Annotating genomes with massive-scale RNA sequencing". Genome Biol. 9 (12): R175. PMC 2646279. PMID 19087247. doi:10.1186/gb-2008-9-12-r175.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]Ligazóns externas
[editar | editar a fonte]- "Genes & Gene Expression". The Virtual Library of Biochemistry and Cell Biology. BioChemWeb.org. 2005-12-04. Arquivado dende o orixinal o 23 de novembro de 2012. Consultado o 2008-06-10.
- John Kryk (2008-05-28). "DNA makes RNA". Arquivado dende o orixinal o 14 de maio de 2019. Consultado o 2008-06-10.
- "Advancing Gene Expression Studies". Genetic Engineering & Biotechnology News. Mary Ann Liebert, Inc. 2008-08-01. Arquivado dende o orixinal o 21 de decembro de 2008.
- "Optimizing Transient Gene Expression". Genetic Engineering & Biotechnology News. Mary Ann Liebert, Inc. 2008-03-01. Arquivado dende o orixinal o 21 de decembro de 2008.
