Proteína

As proteínas (do grego πρωτεϊνη, primeiro) son unhas macromoléculas formadas por α-aminoácidos. Poden estar formadas por unha ou varias cadeas de aminoácidos. As proteínas son moléculas orgánicas esenciais na estrutura e función dos seres vivos (por exemplo, forman parte integral dos tecidos biolóxicos e moitas delas funcionan como encimas). Levan a cabo unha ampla gama de funcións nos organismos, como a catálise metabólica, replicación do ADN, resposta aos estímulos, transporte de moléculas etc. A súa función nutritiva fundamental é plástica, formadora de estruturas orgánicas. Tamén poden servir en determinados casos como fonte de enerxía, pero nunha alimentación equilibrada esta función é asumida polos carbohidratos e os lípidos.
As proteínas son substancias sólidas, incoloras, coloidais, insolubles en solventes orgánicos, unhas con algunha solubilidade en auga, e outras con algunha solubilidade en solucións acuosas diluídas de ácidos, bases ou sales. Quimicamente, son compostos cuaternarios de carbono (C), hidróxeno (H), osíxeno (O) e nitróxeno (N).
Unha cadea longa de aminoácidos denomínase polipéptido. O termo proteína dáse xeralmente cando o polipéptido ou un conxunto de varios ten unha función e conformación determinada. Porén, os termos péptido, polipéptido e proteína úsanse con frecuencia dun modo pouco específico, xa que ás veces non hai un límite claro entre eles. Nas proteínas normalmente hai 20 aminoácidos (ver aminoácido proteinoxénico), pero poden incorporar algúns máis en casos especiais. Os aminoácidos teñen grupos amino e carboxilo por medio dos que se enlazan por enlace peptídico formando a cadea polipeptídica. As proteínas difiren unhas das outras principalmente na súa secuencia de aminoácidos, que está determinada pola secuencia de nucleótidos dos seus xenes, e que xeralmente dá lugar a un pregamento tridimensional específico da proteína, que lle dá unha estrutura necesaria para a súa actividade. Cada posible combinación de tres nucleótidos (tripletes) que se suceden na secuencia dun xene codifica un determinado aminoácido, segundo as correspondencias do código xenético. En xeral, o código xenético só codifica os 20 aminoácidos proteinoxénicos normais, pero en certos organismos dito código pode codificar por un mecanismo especial a selenocisteína e, en certas arqueas a pirrolisina. Despois de rematada a síntese de proteínas ou mesmo durante a mesma, poden modificarse encimaticamente residuos de aminoácidos da proteína por modificación postraducional, o que altera as súas propiedades físicas e químicas, o pregamento, estabilidade, actividade e, finalmente, a función das proteínas. Ás veces as proteínas levan unidos grupos químicos que non son peptídicos, que se denominan grupos prostéticos ou cofactores. Distintas proteínas poden tamén funcionar en conxunto para realizaren unha determinada función, e a miúdo poden estar asociadas formando complexos proteicos estables.
As proteínas poden purificarse a partir doutros compoñentes celulares utilizando varias técnicas como a ultracentrifugación, precipitación, electroforese e cromatografía; a aparición das técnicas de enxeñaría xenética fixo posible aplicar diversos métodos que facilitan a purificación. Entre os métodos que se utilizan comunmente para estudar a estrutura e función das proteínas están a inmunohistoquímica, mutaxénese dirixida, resonancia magnética nuclear e espectrometría de masas.
Bioquímica
[editar | editar a fonte]- Artigo principal: Enlace peptídico.




O enlace peptídico que enlaza os aminoácidos da proteína establécese entre o grupo amino dun aminoácido e o grupo carboxilo do seguinte aminoácido. Nos extremos os grupos amino e carboxilo quedan libres, polo que se di que a proteína ten un extremo amino terminal ou N-terminal (por onde por convención se considera que empeza a proteína) e un extremo carboxilo terminal ou C-terminal (por onde acaba). O enlace peptídico ten dúas formas de resonancia (o dobre enlace pode estar no C=O ou no C=N), que lle dan certo carácter de dobre enlace e inhiben a rotación arredor do seu eixe, de modo que os carbonos alfa son coplanares. Os outros dous ángulos diedros no enlace peptídico determinan a forma local que adopta o esqueleto da proteína.[1]
Síntese
[editar | editar a fonte]Biosíntese
[editar | editar a fonte]- Artigos principais: Biosíntese de proteínas e Tradución de proteínas.
As proteínas son compostos orgánicos de estrutura complexa e masa molecular elevada (entre 15.000 e 20.000.000 Da) e son sintetizadas polos organismos vivos nos ribosomas por medio da combinación dun número maior ou menor de moléculas de aminoácidos, que se ligan por enlace peptídico. Son, pois polímeros formados pola unión de aminoácidos, que son os seus monómeros. Poden ter centos de aminoácidos.
A secuencia de aminoácidos da proteína está determinada pola secuencia de nucleótidos do xene que a codifica. O xene do ADN é transcrito a un ARNm, que madura (en eucariotas) e se dirixe aos ribosomas, únese a eles e é traducido. A secuencia de nucleótidos do ARNm, tomados de 3 en 3, formando codóns, é lida no ribosoma segundo as correspondencias do código xenético. Neste código cada codón determina un aminoácido.[2]. Os aminoácidos chegan ao ribosoma traídos por ARNts, que están entrando e saíndo do ribosoma continuamente. Os ARNts teñen unha secuencia anticodón, complementaria dalgún dos codóns. Hai ARNt específicos para cada aminoácido, e uns encimas chamados aminoacil ARNt sintetases, enlazan cada aminoácido co ARNt correcto. No ribosoma os aminoácidos únense por enlace peptídico formando unha cadea de aminoácidos cada vez máis longa, que permanece unida ao último ARNt que entra. O polipéptido en crecemento denomínase a miúdo cadea nacente. As proteínas biosintetízanse sempre empezando no extremo N-terminal e acabando no C-terminal.[2] O ARNm vai expoñendo sucesivamente todos os seus codóns no ribosoma. Cando se chega a unha secuencia de finalización do ARNm, a cadea polipeptídica libérase. A proteína prégase e pode despois sufrir modificacións postraducionais.
A taxa de síntese proteica en procariotas é máis alta que en eucariotas e pode chegar a 20 aminoácidos por segundo.[3] O tamaño dunha proteína sintetizada pode medirse poo número de aminoácidos que contén e pola súa masa molecular total en daltons, Da, (sinónimo de unidade de masa atómica), ou en kDa. Unha proteína de lévedo media ten uns 446 aminoácidos e uns 53 kDa.[4] A proteína máis longa coñecida é a titina, que se encontra no sarcómero muscular, cunha masa de 3.000 kDa e case 27.000 aminoácidos.[5]
Síntese química
[editar | editar a fonte]No laboratorio poden sintetizarse pequenas proteínas por un conxunto de métodos chamados síntese de péptidos, que dependen de técnicas de síntese orgánica para producir péptidos con alto rendemento.[6] A síntese química permite a introdución de aminoácidos non naturais nas cadeas polipeptídicas, como a unión de sondas flourescentes ás cadeas laterais de aminoácidos.[7] Estes métodos son útiles en bioquímica e bioloxía celular de investigación, aínda que xeralmente non para aplicacións comerciais. A síntese química é ineficiente para producir polipéptidos de máis duns 300 aminoácidos, e as proteínas sintetizadas poden non adoptar doadamente a súa estrutura terciaria nativa. Os métodos en síntese química avanzan dende o extremo C-terminal ao N-terminal, ao revés ca a síntese biolóxica nos ribosomas[8], se son en fase sólida, mais os feitos en disolución avanzan no mesmo sentido que o biolóxico.
Clasificación
[editar | editar a fonte]Composición
[editar | editar a fonte]Canto á estrutura molecular, as proteínas clasifícanse en:
- Proteínas simples:[9]
- Proteínas constituídas soamente por aminoácidos como, por exemplo, a queratina (cabelo).
- Hainas fibrosas, como o coláxeno, elastina, queratinas e fibroína e globulares, como as albuminas, globulinas e histonas e protaminas.
- Proteínas complexas, conxugadas ou heteroproteínas:
- Proteínas que presentan a cadea de aminoácidos ligada a un radical diferente (grupo prostético).
- Dependendo do grupo prostético, as proteínas poden ser clasificadas en:
- Glicoproteínas: o grupo é un glícido. Exemplos: mucina, inmunoglobulinas (anticorpos).
- Cromoproteínas: o grupo é un pigmento. Exemplos: hemoglobina (sangue), que leva un grupo hemo porfirínico con ferro, ou a hemocianina, que non é porfirínica e leva cobre.
- Lipoproteínas: asociadas a lípidos, como as lipoproteínas das membranas celulares ou as que circulan polo plasma sanguíneo.
- Fosfoproteínas: o grupo é o ácido fosfórico. Exemplos: vitelina (xema de ovo) e caseína (leite).
- Nucleoproteínas: o grupo é un ácido nucleico. Por exemplo certas proteínas víricas.
Proteínas fibrosas e globulares
[editar | editar a fonte]- Proteínas fibrosas:
Son aquelas que presentan moléculas distendidas e filamentosas, semellantes a longos fíos. coláxeno e fibrina son exemplos de proteínas fibrosas. Son raras.
- Proteínas globulares:
Presentan as moléculas enroladas como novelos, e son solúbeis na auga formando micelas. A maioría das proteínas presentan estrutura globular, como, por exemplo, os encimas, anticorpos, hemoglobina, proteínas asociadas á clorofila e proteínas estruturais.
Estrutura tridimensional
[editar | editar a fonte]- Artigo principal: Estrutura das proteínas.


As proteínas presentan catro niveis de estudo ou de estrutura que dependen da configuración espacial da cadea polipeptídica, do tamaño da cadea, e do tipo de aminoácidos que posúe. As estruturas básicas son:
- Estrutura primaria:
A estrutura primaria é a secuencia linear de aminoácidos unidos por enlaces peptídicos. Dita estrutura vén predeterminada polo secuencia do ácido nucleico que a codifica.
- Estrutura secundaria:
Constituída por unha cadea de aminoácidos na que se estabelecen ligazóns por pontes de hidróxeno entre os aminoácidos distantes da cadea. Estas ligazóns confírenlle a forma en hélice alfa, folla pregada beta. Esta estrutura depende, entre outros factores, do medio onde a proteína se prega.
- Estrutura terciaria:
Resulta do enrolamento da hélice ou da folla pregada, e é mantido por pontes de hidróxeno e disulfuro entre aminoácidos da mesma cadea. Esta estrutura confire a actividade biolóxica ás proteínas. Pode ser globular ou filamentosa.
- Estrutura cuaternaria:
Resulta da asociación de varias subunidades peptídicas con estrutura terciaria; estas permanecen unidas xeralmente a través de ligazóns covalentes coma pontes disulfuro entre cisteínas.
As proteínas non son moléculas totalmente ríxidas. Ademais de teren os seus niveis estruturais característicos, as proteínas poden cambiar entre varias estruturas relacionadas mentres realizan as súas funcións. No contexto desde rearranxos funcionais, estas estruturas terciarias e cuatrernarias son normalmente denominadas "conformacións", e as transicións entre elas chámanse cambios conformacionais. Tales cambios están a miúdo inducidos pola unión de moléculas substrato ao sitio activo dun encima, ou á rexión física da proteína que participa na catálise química. En proteínas en disolución tamén sofren a variación na estrutura por vibración térmica e a colisión con outras moléculas.[10]
As proteínas poden dividirse informalmente en tres clases principais, que se correlacionan con estruturas terciarias típicas: proteínas globulares, fibrosas e de membrana.[11]
Determinación da estrutura
[editar | editar a fonte]O descubrimento de cal é a estrutura terciaria dunha proteína ou a cuaternaria dun complexo proteico pode proporcionar importantes indicios sobre como realiza a proteína a súa función. Os métodos experimentais máis comúns para a determinación da estrutura son a cristalografía de raios X e a espectroscopía de resonancia magnética nuclear (NMR), as cales poden fornecer información a resolución atómica. Porén, os experimentos de NMR poden proporcionar información a partir da cal pode estimarse un conxunto de distancias entre pares de átomos, e as posibles conformacións finais dunha proteína determínanse resolvendo a distancia xeométrica problema. A interferometría de polarización dual é un método analítico cuantitativo para medir a conformación da proteína total e os cambios conformacionais debidos a interaccións ou outros estímulos. O dicroísmo circular é outra técnica de laboratorio para determinar a composición en folla beta ou helicoidal interna das proteínas. A microscopia crioelectrónica utilízase para producir información estrutural de baixa resolución sobre complexos proteicos moi grandes, incluídos virus ensamblados;[12] unha variante coñecida como cristalografía electrónica pode tamén producir información de alta resolución nalgúns casos, especialmente para cristais bidimensionais de proteínas de membrana.[13]
As estruturas resoltas almacénanse xeralmente en Protein Data Bank (PDB), unha fonte de acceso libre da que se poden obter datos de miles de proteínas en forma de coordenadas cartesianas para cada átomo da proteína.[14] Coñécense moitas máis secuencias que estruturas de proteínas. Ademais, o conxunto de estruturas xa resoltas é predominantemente o de proteínas que poden ser sometidas facilmente ás condicións requiridas na cristalografía de raios X, que é un dos principais métodos de determinación estrutural. En especial, as proteínas globulares son comparativamente máis doadas de cristalizar en preparacións de cristalografía de raios X. As proteínas de membrana, polo contrario, son difíciles de cristalizar e están subrepresentadas na PDB.[15] As iniciativas baseadas na xenómica estrutural intentaron remediar estas deficiencias resolvendo sistematicamente estruturas representativas dos principais tipos de pregamento. Os métodos de predición de estruturas das proteínas tratan de proporcionar métodos de xerar unha estrutura plausible de proteínas cuxas estruturas non foron aínda determinadas experimentalmente.[16]
Desnaturalización e renaturalización
[editar | editar a fonte]As proteínas poden desnaturalizar. Isto acontece cando, por acción de substancias químicas ou da calor as proteínas sofren alteración da estrutura terciaria ou a quebra das ligazóns non covalentes da estrutura cuaternaria.
As proteínas perden a súa conformación e, consecuentemente, a súa funcionalidade. A desnaturalización pode ser reversíbel ou irreversíbel.
Dependendo da forma pola cal a proteína foi desnaturalizada, a súa conformación nativa pódese recuperar (renaturalización) retirándose lentamente o axente desnaturalizante, como por exemplo facer unha diálise contra auga para retirar o axente desnaturalizante urea.
Función biolóxica
[editar | editar a fonte]As proteínas son as principais moléculas da célula, que levan a cabo as instrucións especificadas na información xenética.[4] Coa excepción de certos tipos de ARN, a maioría das demais moléculas biolóxicas son elementos relativamente inertes sobre as cales actúan as proteínas. As proteínas supoñen a metade do peso seco dunha célula de Escherichia coli, mentres que outras macromoléculas como o ADN e ARN supoñen só o 3% e 20%, respectivamente.[17] O conxunto de proteínas expresadas nunha determinada célula ou tipo celular denomínase proteoma.
Igual que outras macromoléculas biolóxicas como os polisacáridos e ácidos nucleicos, as proteínas son partes esenciais do organismo e participan virtualmente en todos os procesos celulares. Moitas proteínas son encimas que catalizan reaccións bioquímicas e son esenciais no metabolismo. As proteínas tamén teñen funcións estruturais e mecánicas, como a actina e miosina do músculo e as proteínas do citoesqueleto, o cal forma un armazón que mantén a forma celular. Outras proteínas son importantes na sinalización celular, respostas inmunes, adhesión celular e o ciclo celular. As proteínas son tamén necesarias nas dietas dos animais, xa que os animais non poden sintetizar todos os aminoácidos que necesitan e deben obter os aminoácidos esenciais da dieta. Por medio do proceso da dixestión, os animais degradan as proteínas a aminoácidos libres que son despois absorbidos no intestino e utilizados no metabolismo.
As proteínas teñen as seguintes funcións:
Estrutural e motil
[editar | editar a fonte]Son aquelas que participan dos tecidos dándolles rixidez, consistencia e elasticidade. Son proteínas estruturais: coláxeno ( cartilaxes), elastina, fibroína (arañeiras), queratina (cabelo), resilina (moi elástica, nas ás de insectos) e outras.
Algunhas proteínas estruturais interveñen na motilidade celular ou de todo o organismo. Algunhas delas teñen propiedades contráctiles e interveñen nos movementos das células musculares e outras, como é o caso da actinae miosina. Outras proteínas que interveñen na motilidade intracelular son a cinesina e dineína. A proteína tubulina non é contráctil, pero forma os flaxelos e cilios.
Regulatoria
[editar | editar a fonte]Algunhas hormonas (substancias que exercen algunha función específica sobre algún órgano ou estrutura dun organismo) son de natureza proteica, como por exemplo a insulina, hormona do crecemento ou hormona paratiroide. Outras interveñen na sinalización celular como receptores de moléculas sinalizadoras eucariotas, ou son represores de xenes bacterianos. Moitos receptores teñen un sitio de unión exposto na superficie da célula e un dominio efector dentro da célula, o cal pode ter unha actividade encimática ou poden sufrir un cambio conformacional detectado por outras proteínas do interior da célula.[18]
Defensa
[editar | editar a fonte]Os anticorpos son proteínas (inmunoglobulinas) que participan na defensa do organismo contra microorganismos e todo tipo de substancias estrañas. A afinidade de unión dun anticorpo pola súa molécula diana é extraordinariamente alta.[19] Tamén teñen unha función defensiva impedindo a perda de sangue nas hemorraxias, como o fibrinóxeno ou a trombina. Outras son tóxicas, como a ricina, que é unha forma de defensa das plantas. No plasma de peixes antárticos hai proteínas anticonxelantes que os defenden da conxelación.
Enerxética (nutrición e reserva)
[editar | editar a fonte]Obtención de enerxía a partir dos aminoácidos que compoñen as proteínas. Esta é unha función de urxencia en organismos superiores, cando a indispoñibilidade de ácidos graxos ou monosacáridos así o dita. Porén, hai algunhas proteínas que están especializadas na nutrición e reserva enerxética, como a caseína do leite, a ovoalbumina do ovo ou a gliadina das sementes de trigo.
Encimática
[editar | editar a fonte]- Artigo principal: Encima.
Posibilitan as reaccións bioquímicas, permitindo reducir a enerxía libre de Gibbs de activación e acelerando as reaccións. Coñécense máis de 4000 reaccións catalizadas por encimas. Os encimas son xeralmente moi específicos e catalizan só unha ou unhas poucas reaccións químicas. Os encimas levan a cabo a maior parte das reaccións do metabolismo, interveñen na replicación do ADN, reparación do ADN e transcrición xenética. Algúns encimas actúan sobre outras proteínas par engadir ou quitar grupos químicos nun proceso chamado modificación postraducional.[20] O grao de aceleración proporcionado pola catálise encimática é xeralmente enorme, por exemplo, incrementando a velocidade de reacción arredor de 1017 veces con respecto a unha reacción non catalizada no caso da orotato descarboxilase.[21]
As moléculas que se unen e sobre as que actúa o encima denomínanse substratos. O substrato entra nunha rexión do encima denominada centro activo, onde ten lugar a reacción. Aínda que o encima pode estar formado por centos de aminoácidos, só unha pequena fracción dos aminoácidos interaccionan co substrato, e na catálise adoitan estar implicados só tres ou catro aminoácidos.[22]
Transporte
[editar | editar a fonte]Algunhas proteínas transportan substancias. Por exemplo, os gases respiratorios (osíxeno e dióxido de carbono) son transportados por proteínas como a hemoglobina e hemocianina. A hemoglobina é un exemplo de proteína que se une con alta afinidade a unha pequena molécula ligando cando o ligando está presente en altas concentracións (como ocorre nos pulmóns) e que liberan o ligando cando chegan a tecidos onde hai baixas concentracións deste.[23]
As lipoproteínas transportan lípidos polo sangue. Algunhas proteínas son transportadores de membrana, que levan moléculas dun lado a outro da membrana.
Unión a outras moléculas
[editar | editar a fonte]
A principal característica das proteínas que lles permiten realizar o seu diverso conxunto de funcións é a súa capacidade de unirse a outras moléculas de forma específica e firme. A rexión das moléculas proteicas responsable da unión a outras moléculas denomínase sitio de unión e é xeralmente unha depresión na superficie da molécula ou "peto". Esta capacidade de unión está mediada pola súa estrutura terciaria, a cal define o peto do sitio de unión, e polas propiedades químicas das cadeas laterais dos aminoácidos que o rodean. A unión das proteínas a outras moléculas pode ser extraordinariamente forte e específica; por exemplo, a proteína inhibidora da ribonuclease únese á anxioxenina humana cunha constante de disociación subfemtomolar (<10−15 M) pero non se pode unir a ningún dos seus homólogos de anfibios, as onconases (>1 M). Cambios químicos extremadamente pequenos como a adición dun só grupo metilo a unha molécula á que se debe unir a proteína pode ás veces ser suficiente para case eliminar a unión; por exemplo, a aminoacil ARNt sintetase específica do aminoácido valina distingue esta do aminoácido leucina de cadea lateral moi similar.[24]
As proteínas poden unirse a outras proteínas ou a pequenas moléculas substrato. Cando as proteínas se unen especificamente a outras copias da mesma molécula, poden oligomerizarse para formar fibrilas; este proceso ocorre a miúdo en proteínas estruturais que constan de monómeros globulares que se autoensamblan para formar fibras ríxidas. As interaccións proteína-proteína tamén regulan a actividade encimática, o control do progreso do ciclo celular e permiten a ensamblaxe de grandes complexos proteicos que levan a cabo moitas reaccións moi relacionadas cunha función biolóxica común. As proteínas poden tamén unirse ou mesmo integrarse nas membranas cellares. A capacidade de unirse a outras moléculas para inducir cambios conformacionais nas proteínas permite a formación de redes de sinalización celular de enorme complexidade.[25] As interaccións entre proteínas son reversibles, e dependen fortemente da dispoñibilidade de diferentes grupos de proteínas ás que se asocian para formar agregados que son capaces de realizar un determinado conxunto de funcións. O estudo da interacción entre proteínas específicas é fundamental para comprender aspectos importantes das funcións celulares, e finalmente as propiedades que distinguen os distintos tipos celulares.[26][27]

Métodos de estudo
[editar | editar a fonte]As estruturas e actividades das proteínas foron estudadas in vitro e in vivo. Os estudos in vitro de proteínas purificadas en condicións controladas son útiles para descubrir como unha proteína leva a cabo a súa función: por exemplo, os estudos de cinética encimática exploran os mecanismos químicos da actividade catalítica dun encima e a súa afinidade relativa por varios posibles substratos. Os experimentos in vivo estudan as actividades das proteínas dentro das células ou dentro do organismo no seu conxunto, e poden proporcionar información complementaria sobre o lugar onde actúa a proteína e como se regula.
Purificación de proteínas
[editar | editar a fonte]Para realizar unha análise in vitro dunha proteína, esta debe ser purificada e separada doutros compoñentes celulares. Este proceso xeralmente empeza coa lise da célula, na cal se destrúe a membrana celular e o contido da célula libérase formando unha solución chamada lisado cru. A mestura resultante pode ser purificada utilizando ultracentrifugación, a cal fracciona os diversos compoñentes celulares en fraccións que conteñen proteínas solubles; lípidos de membrana e proteínas; orgánulos celulares e ácidos nucleicos. A precipitación por un método baseado en utilizar altas concentracións salinas pode concentrar as proteínas do lisado. Despois úsanse varios tipos de cromatografía para illar a proteína ou proteínas que interesan baseándose en propiedades como a masa molecular, carga neta e afinidade de unión.[28] O nivel de purificación pode ser monitorizado usando varios tipos de electroforese en xel se se coñecen a masa molecular da proteína desexada e o seu punto isoeléctrico, ou por espectroscopia se a proteína ten características espectroscópicas distinguibles, ou por ensaios de encimas se a proteína ten unha actividade encimática. Ademais, as proteínas poden ser illadas segundo a súa carga por medio do electroenfoque.[29]
Para as proteínas naturais, pode ser necesario seguir unha serie de pasos de purificación para obter proteínas puras dabondo para as aplicacións de laboratorio. Para simplificar este proceso, a míúdo utilízase a enxeñaría xenética para engadir características químicas á proteína que a fagan máis fácil de purificar sen afectar á súa estrutura ou actividade. Por exemplo, únese unha "etiqueta" consistente nunha secuencia de aminoácidos específica, como unha serie de residuos de histidina (unha "etiqueta His"), que se enlaza a un extremo da proteína. Como resultado, cando o lisado se fai pasar por unha columna de cromatografía que conteña níquel, os residuos de histidina líganse ao níquel e quedan unidos á columna (xunto con toda a proteína) mentres que os compoñentes non etiquetados do lisado escoan sen dificultade. Ideáronse varias etiquetas diferentes para axudar aos investigadores a purificar proteínas específicas a partir de mesturas complexas.[30]
Localización celular
[editar | editar a fonte]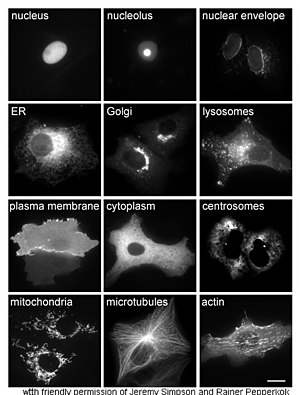
O estudo das proteínas in vivo trata moitas veces de coñecer cal é a localización e lugar de síntese da proteína na célula. Aínda que moitas proteínas intracelulares se sintetizan no citoplasma e as proteínas unidas a membrana ou segregadas polo retículo endoplasmático, moitas veces non están tan claros os detalles de como as proteínas se destinan a orgánulos específicos ou estruturas celulares. Unha técnica útil para estimar a localización celular utiliza a enxeñaría xenética para expresar na célula unha proteína de fusión ou quimera formada pola proteína natural de interese unida a un "reporteiro" como a proteína fluorescente verde (GFP).[31] A localización na célula da proteína fusionada pode ser visualizada de forma clara e eficiente por microscopía,[32] como se ve no exemplo da figura.
Outros métodos para dilucidar a localización celular de proteínas require o uso de marcadores de compartimento coñecidos para rexións como o retículo endoplasmático, o aparato de Golgi, lisosomas ou vacúolos, mitocondria, cloroplastos, membrana plasmática etc. Co uso de versións etiquetadas fluorescentemente destes marcadores ou de anticorpos para marcadores coñecidos, fíxose máis sinxelo identificar a localización dunha proteína de interese. Por exemplo, a inmunofluorescencia indirecta permite a colocalizaciónn fluorescente e a demostración da localización. As tinguiduras fluorescentes utilízanse para etiquetar compartimentos celulares para un propósito similar.[33]
Existen outras posibilidades. Por exemplo, a inmunohistoquímica xeralmente utiliza un anticorpo para unha ou máis proteínas de interese que están conxugadas a encimas producindo sinais luminescentes ou cromoxénicos que poden compararse entre mostras, o que permite a localización da información. Outra técnica aplicable é a cofraccionación en gradientes de sacarosa (ou outro material) utilizando centrifugación isopícnica.[34] Aínda que esta técnica non proba a colocalización dun compartimento de densidade coñecida e a proteína de interese, incrementa a probabilidade, e é máis axeitado para estudos a grande escala.
Finalmente, o método estándar de localización celular é a microscopía inmunoelectrónica. Esta técnica tamén usa un anticorpo para a proteína de interese, xunto coas técnicas de microscopía electrónica clásicas. A mostra prepárase para o exame por microscopía electrónica normal, e despois é tratada cun anticorpo para a proteína de interese que é conxugada a un material extremadamente electrodenso, xeralmente ouro. Isto permite a localización tanto dos detalles estruturais coma da proteína procurada.[35]
Con outra aplicación de enxeñaría xenética chamada mutaxénese dirixida a sitio, pódese alterar a secuencia da proteína e, polo tanto, a súa estrutura, localización celular e susceptibilidade á regulación. Esta técnica mesmo permite a incorporación ás proteínas de aminoácidos, utilizando ARNts modificados,[36] e pode permitir o deseño de novas proteínas con novas propiedades.[37]
Proteómica e bioinformática
[editar | editar a fonte]- Artigos principais: Proteómica e Bioinformática.
O complemento proteico completo presente nun determinado momento nunha célula ou tipo celular denomínase proteoma, e o estudo de conxuntos de datos a grande escala define o campo da proteómica, denominado así por analoxía co campo relacionado da xenómica. Técnicas experimentais fundamentais en proteómica son a electroforese en xel bidimensional,[38] que posibilita a separación dun gran número de proteínas, a espectrometría de masas,[39] que permite unha rápida identificación de alto rendemento de proteínas e a secuenciación de péptidos (xeralmente despois dunha dixestión en xel), as micromatrices de proteínas,[40] coas que se poden detectar os niveis relativos de gran número de proteínas presentes na célula, e sistema de dobre híbrido, que permite s exploración sistemática das interaccións proteína-proteína.[41] O conxunto total de todas estas interaccións bioloxicamente posibles denomínase interactoma.[42] Un intento sistemático de determinar as estruturas de proteínas que inclúa todos os posibles pregamentos coñécese como xenómica estrutural.[43]
Disponse dunha gran cantidade de datos xenómicos e proteómicos de varios organismos, entre eles o do xenoma humano, que facilitan que os investigadores identifiquen eficientemente proteínas homologas mesmo en organismos escasamente relacionados por medio de aliñamento de secuencias. As ferramentas de perfilado de secuencias poden realizar manipulacións máis específicas de secuencia como os mapas de encimas de restrición, análises de marcos abertos de lectura de secuencias de nucleótidos e predición da estrutura secundaria. A partir destes datos poden construírse árbores filoxenéticas e desenvolverse hipóteses sobre a evolución utilizando un software especial como ClustalW en relación cos antepasados dos organismos modernos e os xenes que expresan. O eido da bioinformática trata de ensamblar, anotar e analizar datos proteómicos e xenómicos, aplicando técnicas computacionais a problemas biolóxicos como a procura de xenes e a cladística.
Predición da estrutura e simulación
[editar | editar a fonte]A predición da estrutura das proteínas é unha área complementaria da xenómica estrutural, que trata de predicir as estruturas proteicas para elaborar modelos axeitados das proteínas cuxas estruturas non foron aínda determinadas experimentalmente.[44] O tipo máis exitoso de predición de estruturas, chamado modelado de homoloxía, baséase na existencia dunha estrutura "patrón" ou "molde" con semellanza de secuencia coa proteína que está a ser modelada; o obxectivo da xenómica estrutural é proporcionar unha representación suficiente das estruturas xa resoltas para modelar a maioría das que quedan.[45] Aínda que elaborar modelos axeitados segue a ser un reto cando só se dispón de estruturas "patrón" pouco relacionadas, suxeriuse que o aliñamento de secuencia é o punto crítico neste proceso, xa que poden producirse modelos bastante axeitados se se coñece un aliñamento de secuencia "perfecto".[46] Moitos métodos de predición de estruturas serviron para informar o campo emerxente da enxeñaría de proteínas, no cal foron xa deseñados os novos pregamentos de proteínas.[47] Un problema computacional máis complexo é a predición de interaccións intermoleculares, como no acoplamento molecular e a predición da interacción proteína–proteína.[48]
O proceso de pregamento de proteínas e a súa unión a outras moléculas poden ser simulados utilizando técnicas como a mecánica molecular, en especial, a dinámica molecular e o método Monte Carlo, que cada vez máis aproveitan a computación distribuída e en paralelo (proxecto Folding@home;[49] modelado molecular en GPU). O pregamento de pequenos dominios proteicos en hélice alfa como as "cabezas" da proteína vilina[50] e a proteína accesoria do VIH[51] simulouse con éxito en silicio (no PC), e os métodos híbridos que combinan a dinámica molecular estándar con cálculos de mecánica cuántica permitiron a exploración dos estados electrónicos das rodopsinas.[52]
Historia
[editar | editar a fonte]As proteínas foron recoñecidas como unha clase distinta de moléculas biolóxicas no século XVIII por Antoine Fourcroy e outros, pola súa capacidade de coagular ou flocular cando se trataban con calor ou ácidos. Notables exemplos de proteínas recoñecidas naquel momento foron a albumina da clara dos ovos, a albumina sérica do sangue, a fibrina e o glute do trigo.
As proteínas foron descritas por primeira vez polo químico holandés Gerardus Johannes Mulder e nomeadas polo químico sueco Jöns Jacob Berzelius en 1838. Mulder levou a cabo a análise elemental de proteínas comúns e encontrou que case todas as proteínas tiñan a mesma fórmula empírica, C400H620N100O120P1S1.[53] Chegou á errada conclusión de que poderían estar compostas por un só tipo de molécula (moi grande). O termo "proteína" proposto por Berzelius deriva do grego πρωτεῖος (proteios), que significa "primario",[54] "á cabeza", ou "situado diante".[55] Mulder seguiu os seus traballos identificando os produtos da degradación das proteínas como o aminoácido leucina para a cal calculou un peso molecular (case correcto) de 131 Da.[53]
Os primeiros científicos da nutrición como o alemán Carl von Voit crían que as proteínas eran os nutrientes máis importantes para manter a estrutura do corpo, porque críase xeralmente que a "carne fai carne".[56] O papel fundamental das proteínas como encimas nos organismos vivos non foi totalmente apreciado ata 1926, cando James Batcheller Sumner mostrou que o encima urease era, de feito, unha proteína.[57]
A dificultade que ten purificar proteínas en grandes cantidades fixo que fose moi difícil o seu estudo polos primeiros bioquímicos. Por iso, os primeiros estudos centráronse en proteínas que podían ser purificadas en grandes cantidades, como as do sangue, clara de ovo, varias toxinas e encimas dixestivos ou metabólicos obtidos en matadeiros. Na década de 1950, a compañía Armour Hot Dog Co. purificou 1 kg de ribonuclease A pancreática bovina pura e púxoa a disposición dos científicos gratuitamente, o que contribuíu a que a ribonuclease A se convertese nun dos obxectivos principais dos estudos bioquímicos nos anos seguintes.[53]

Linus Pauling predixo con éxito a estrutura secundaria dunha proteína regular baseándose na formación de enlaces de hidróxeno, unha idea que fora adiantada en 1933 por William Astbury.[58] Os traballos posteriores de Walter Kauzmann sobre a desnaturalización,[59][60] baseados en parte en estudos previos de Kaj Linderstrøm-Lang,[61] contribuíron ao coñecemento do pregamento das proteínas e das estruturas mediadas por interaccións hidrofóbicas.
A primeira proteína que foi secuenciada foi a insulina, por Frederick Sanger, en 1949. Sanger determinou correctamente a secuencia de aminoácidos da insulina, demostrando así concluíntemente que as proteínas consisten en polímeros liñais de aminoácidos en vez de cadeas ramificadas, coloides, ou ciclois.[62] Este descubrimento valeulle o premio Nobel en 1958.
As primeiras estruturas proteicas resoltas foron as da hemoglobina e mioglobina, por Max Perutz e Sir John Cowdery Kendrew, respectivamente, en 1958.[63][64] A primeira estrutura proteica resolta con resolución atómica por análise de difracción de raios X fíxose na década de 1960 (Perutz e Kendrew compartiron o premio Nobel de Química de 1962 por estes descubrimentos) e posteriormente por espectroscopía de resonancia magnética nuclear de proteínas (NMR) na década de 1980. En 2009,o Protein Data Bank contiña unhas 55.000 estruturas de proteínas con resolución atómica.[65] Recentemente, a microscopía crioelectrónica de grandes ensamblaxes macromoleculares[66] e a predición da estrutura de proteínas computacional de dominios de pequenas proteínas[67] son dous métodos con resolución atómica.
Notas
[editar | editar a fonte]- ↑ Murray et al., p. 31.
- ↑ 2,0 2,1 van Holde and Mathews, pp. 1002–42.
- ↑ Dobson CM (2000). "The nature and significance of protein folding". En Pain RH (ed.). Mechanisms of Protein Folding. Oxford, Oxfordshire: Oxford University Press. pp. 1–28. ISBN 0-19-963789-X.
- ↑ 4,0 4,1 Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipurksy SL, Darnell J (2004). Molecular Cell Biology (5th ed.). New York, New York: WH Freeman and Company.
- ↑ Fulton A, Isaacs W (1991). "Titin, a huge, elastic sarcomeric protein with a probable role in morphogenesis". BioEssays 13 (4): 157–61. PMID 1859393. doi:10.1002/bies.950130403.
- ↑ Bruckdorfer T, Marder O, Albericio F (2004). "From production of peptides in milligram amounts for research to multi-tons quantities for drugs of the future". Current Pharmaceutical Biotechnology 5 (1): 29–43. PMID 14965208. doi:10.2174/1389201043489620.
- ↑ Schwarzer D, Cole P (2005). "Protein semisynthesis and expressed protein ligation: chasing a protein's tail". Current Opinions in Chemical Biology 9 (6): 561–69. PMID 16226484. doi:10.1016/j.cbpa.2005.09.018.
- ↑ Kent SB (2009). "Total chemical synthesis of proteins". Chemical Society Reviews 38 (2): 338–51. PMID 19169452. doi:10.1039/b700141j.
- ↑ En España, na literatura en castelán, adoita utilizarse o termo holoproteína para denominar as proteínas simples. Pero, ao contrario, na literatura inglesa holoprotein significa heteroproteína (de modo análogo ao termo holoencima), e este significado está moi estendido polo mundo. Neste artigo evitouse usar o termo holoproteína debido á súa ambigüidade.
- ↑ van Holde and Mathews, pp. 368–75.
- ↑ van Holde and Mathews, pp. 165–85.
- ↑ Branden and Tooze, pp. 340–41.
- ↑ Gonen T, Cheng Y, Sliz P, Hiroaki Y, Fujiyoshi Y, Harrison SC, Walz T (2005). "Lipid-protein interactions in double-layered two-dimensional AQP0 crystals". Nature 438 (7068): 633–38. Bibcode:2005Natur.438..633G. PMC 1350984. PMID 16319884. doi:10.1038/nature04321.
- ↑ Standley DM, Kinjo AR, Kinoshita K, Nakamura H (2008). "Protein structure databases with new web services for structural biology and biomedical research". Briefings in Bioinformatics 9 (4): 276–85. PMID 18430752. doi:10.1093/bib/bbn015.
- ↑ Walian P, Cross TA, Jap BK (2004). "Structural genomics of membrane proteins". Genome Biology 5 (4): 215. PMC 395774. PMID 15059248. doi:10.1186/gb-2004-5-4-215. Arquivado dende o orixinal o 02 de xaneiro de 2016. Consultado o 27 de maio de 2013.
- ↑ Sleator RD. (2012). "Prediction of protein functions". Methods in Molecular Biology 815: 15–24. doi:10.1007/978-1-61779-424-7_2.
- ↑ Voet D, Voet JG. (2004). Biochemistry Vol 1 3rd ed. Wiley: Hoboken, NJ.
- ↑ Branden and Tooze, pp. 251–81.
- ↑ van Holde and Mathews, pp. 247–50.
- ↑ Bairoch A (2000). "The ENZYME database in 2000". Nucleic Acids Research 28 (1): 304–305. PMC 102465. PMID 10592255. doi:10.1093/nar/28.1.304.
- ↑ Radzicka A, Wolfenden R (1995). "A proficient enzyme". Science 267 (5194): 90–93. Bibcode:1995Sci...267...90R. PMID 7809611. doi:10.1126/science.7809611.
- ↑ EBI External Services (2010-01-20). "The Catalytic Site Atlas at The European Bioinformatics Institute". Ebi.ac.uk. Arquivado dende o orixinal o 03 de agosto de 2013. Consultado o 2011-01-16.
- ↑ van Holde and Mathews, pp. 220–29.
- ↑ Sankaranarayanan R, Moras D (2001). "The fidelity of the translation of the genetic code". Acta Biochimica Polonica 48 (2): 323–35. PMID 11732604.
- ↑ van Holde and Mathews, pp. 830–49.
- ↑ Copland JA, Sheffield-Moore M, Koldzic-Zivanovic N, Gentry S, Lamprou G, Tzortzatou-Stathopoulou F, Zoumpourlis V, Urban RJ, Vlahopoulos SA (2009). "Sex steroid receptors in skeletal differentiation and epithelial neoplasia: is tissue-specific intervention possible?". BioEssays: news and reviews in molecular, cellular and developmental biology 31 (6): 629–41. PMID 19382224. doi:10.1002/bies.200800138.
- ↑ Samarin S, Nusrat A (2009). "Regulation of epithelial apical junctional complex by Rho family GTPases". Frontiers in bioscience: a journal and virtual library 14 (14): 1129–42. PMID 19273120. doi:10.2741/3298.
- ↑ Murray et al., pp. 21–24.
- ↑ Hey J, Posch A, Cohen A, Liu N, Harbers A (2008). "Fractionation of complex protein mixtures by liquid-phase isoelectric focusing". Methods in Molecular Biology. Methods in Molecular Biology™ 424: 225–39. ISBN 978-1-58829-722-8. PMID 18369866. doi:10.1007/978-1-60327-064-9_19.
- ↑ Terpe K (2003). "Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems". Applied Microbiology and Biotechnology 60 (5): 523–33. PMID 12536251. doi:10.1007/s00253-002-1158-6.
- ↑ Stepanenko OV, Verkhusha VV, Kuznetsova IM, Uversky VN, Turoverov KK (2008). "Fluorescent proteins as biomarkers and biosensors: throwing color lights on molecular and cellular processes". Current Protein & Peptide Science 9 (4): 338–69. PMC 2904242. PMID 18691124. doi:10.2174/138920308785132668.
- ↑ Yuste R (2005). "Fluorescence microscopy today". Nature Methods 2 (12): 902–904. PMID 16299474. doi:10.1038/nmeth1205-902.
- ↑ Margolin W (2000). "Green fluorescent protein as a reporter for macromolecular localization in bacterial cells". Methods (San Diego, Calif.) 20 (1): 62–72. PMID 10610805. doi:10.1006/meth.1999.0906.
- ↑ Walker JH, Wilson K (2000). Principles and Techniques of Practical Biochemistry. Cambridge, UK: Cambridge University Press. pp. 287–89. ISBN 0-521-65873-X.
- ↑ Mayhew TM, Lucocq JM (2008). "Developments in cell biology for quantitative immunoelectron microscopy based on thin sections: a review". Histochemistry and Cell Biology 130 (2): 299–313. PMC 2491712. PMID 18553098. doi:10.1007/s00418-008-0451-6.
- ↑ Hohsaka T, Sisido M (2002). "Incorporation of non-natural amino acids into proteins". Current Opinion in Chemical Biology 6 (6): 809–15. PMID 12470735. doi:10.1016/S1367-5931(02)00376-9.
- ↑ Cedrone F, Ménez A, Quéméneur E (2000). "Tailoring new enzyme functions by rational redesign". Current Opinion in Structural Biology 10 (4): 405–10. PMID 10981626. doi:10.1016/S0959-440X(00)00106-8.
- ↑ Görg A, Weiss W, Dunn MJ (2004). "Current two-dimensional electrophoresis technology for proteomics". Proteomics 4 (12): 3665–85. PMID 15543535. doi:10.1002/pmic.200401031.
- ↑ Conrotto P, Souchelnytskyi S (2008). "Proteomic approaches in biological and medical sciences: principles and applications". Experimental Oncology 30 (3): 171–80. PMID 18806738.
- ↑ Joos T, Bachmann J (2009). "Protein microarrays: potentials and limitations". Frontiers in Bioscience 14 (14): 4376–85. PMID 19273356. doi:10.2741/3534.
- ↑ Koegl M, Uetz P (2007). "Improving yeast two-hybrid screening systems". Briefings in Functional Genomics & Proteomics 6 (4): 302–12. PMID 18218650. doi:10.1093/bfgp/elm035. Arquivado dende o orixinal o 15 de abril de 2013. Consultado o 27 de maio de 2013.
- ↑ Plewczyński D, Ginalski K (2009). "The interactome: predicting the protein–protein interactions in cells". Cellular & Molecular Biology Letters 14 (1): 1–22. PMID 18839074. doi:10.2478/s11658-008-0024-7.
- ↑ Zhang C, Kim SH (2003). "Overview of structural genomics: from structure to function". Current Opinion in Chemical Biology 7 (1): 28–32. PMID 12547423. doi:10.1016/S1367-5931(02)00015-7.
- ↑ Zhang Y (2008). "Progress and challenges in protein structure prediction". Current Opinions in Structural Biology 18 (3): 342–48. PMC 2680823. PMID 18436442. doi:10.1016/j.sbi.2008.02.004.
- ↑ Xiang Z (2006). "Advances in homology protein structure modeling". Current Protein and Peptide Science 7 (3): 217–27. PMC 1839925. PMID 16787261. doi:10.2174/138920306777452312.
- ↑ Zhang Y, Skolnick J (2005). "The protein structure prediction problem could be solved using the current PDB library". Proceedings of the National Academy of Sciences U.S.A. 102 (4): 1029–34. Bibcode:2005PNAS..102.1029Z. PMC 545829. PMID 15653774. doi:10.1073/pnas.0407152101.
- ↑ Kuhlman B, Dantas G, Ireton GC, Varani G, Stoddard BL, Baker D (2003). "Design of a novel globular protein fold with atomic-level accuracy". Science 302 (5649): 1364–68. Bibcode:2003Sci...302.1364K. PMID 14631033. doi:10.1126/science.1089427.
- ↑ Ritchie DW (2008). "Recent progress and future directions in protein–protein docking". Current Protein and Peptide Science 9 (1): 1–15. PMID 18336319. doi:10.2174/138920308783565741.
- ↑ Scheraga HA, Khalili M, Liwo A (2007). "Protein-folding dynamics: overview of molecular simulation techniques". Annual Review of Physical Chemistry 58: 57–83. Bibcode:2007ARPC...58...57S. PMID 17034338. doi:10.1146/annurev.physchem.58.032806.104614.
- ↑ Zagrovic B, Snow CD, Shirts MR, Pande VS (2002). "Simulation of folding of a small alpha-helical protein in atomistic detail using worldwide-distributed computing". Journal of Molecular Biology 323 (5): 927–37. PMID 12417204. doi:10.1016/S0022-2836(02)00997-X.
- ↑ Herges T, Wenzel W (2005). "In silico folding of a three helix protein and characterization of its free-energy landscape in an all-atom force field". Physical Review Letters 94 (1): 018101. Bibcode:2005PhRvL..94a8101H. PMID 15698135. doi:10.1103/PhysRevLett.94.018101.
- ↑ Hoffmann M, Wanko M, Strodel P, König PH, Frauenheim T, Schulten K, Thiel W, Tajkhorshid E, Elstner M (2006). "Color tuning in rhodopsins: the mechanism for the spectral shift between bacteriorhodopsin and sensory rhodopsin II". Journal of the American Chemical Society 128 (33): 10808–18. PMID 16910676. doi:10.1021/ja062082i.
- ↑ 53,0 53,1 53,2 Perrett D (2007). "From 'protein' to the beginnings of clinical proteomics". Proteomics – Clinical Applications 1 (8): 720–38. PMID 21136729. doi:10.1002/prca.200700525.
- ↑ New Oxford Dictionary of English
- ↑ Reynolds JA, Tanford C (2003). Nature's Robots: A History of Proteins (Oxford Paperbacks). New York, New York: Oxford University Press. p. 15. ISBN 0-19-860694-X.
- ↑ Bischoff TLW, Voit, C (1860). Die Gesetze der Ernaehrung des Pflanzenfressers durch neue Untersuchungen festgestellt (en German). Leipzig, Heidelberg.
- ↑ Sumner JB (1926). "The isolation and crystallization of the enzyme urease. Preliminary paper" (PDF). Journal of Biological Chemistry 69 (2): 435–41.
- ↑ Pauling L, Corey RB, Branson HR (1951). "The structure of proteins: two hydrogen-bonded helical configurations of the polypeptide chain" (PDF). Proceedings of the National Academy of Sciences U.S.A. 37 (5): 235–40. Bibcode:1951PNAS...37..235P. PMC 1063348. PMID 14834145. doi:10.1073/pnas.37.5.235.
- ↑ Kauzmann W (1956). "Structural factors in protein denaturation". Journal of Cellular Physiology. Supplement 47 (Suppl 1): 113–31. PMID 13332017. doi:10.1002/jcp.1030470410.
- ↑ Kauzmann W (1959). "Some factors in the interpretation of protein denaturation". Advances in Protein Chemistry. Advances in Protein Chemistry 14: 1–63. ISBN 978-0-12-034214-3. PMID 14404936. doi:10.1016/S0065-3233(08)60608-7.
- ↑ Kalman SM, Linderstrom-Lang K, Ottesen M, Richards FM (1955). "Degradation of ribonuclease by subtilisin". Biochimica et Biophysica Acta 16 (2): 297–99. PMID 14363272. doi:10.1016/0006-3002(55)90224-9.
- ↑ Sanger F (1949). "The terminal peptides of insulin". Biochemical Journal 45 (5): 563–74. PMC 1275055. PMID 15396627.
- ↑ Muirhead H, Perutz M (1963). "Structure of hemoglobin. A three-dimensional fourier synthesis of reduced human hemoglobin at 5.5 Å resolution". Nature 199 (4894): 633–38. Bibcode:1963Natur.199..633M. PMID 14074546. doi:10.1038/199633a0.
- ↑ Kendrew J, Bodo G, Dintzis H, Parrish R, Wyckoff H, Phillips D (1958). "A three-dimensional model of the myoglobin molecule obtained by X-ray analysis". Nature 181 (4610): 662–66. Bibcode:1958Natur.181..662K. PMID 13517261. doi:10.1038/181662a0.
- ↑ "RCSB Protein Data Bank". Arquivado dende o orixinal o 27 de decembro de 2012. Consultado o 2009-04-14.
- ↑ Zhou ZH (2008). "Towards atomic resolution structural determination by single-particle cryo-electron microscopy". Current Opinion in Structural Biology 18 (2): 218–28. PMC 2714865. PMID 18403197. doi:10.1016/j.sbi.2008.03.004.
- ↑ Keskin O, Tuncbag N, Gursoy A (2008). "Characterization and prediction of protein interfaces to infer protein-protein interaction networks". Current Pharmaceutical Biotechnology 9 (2): 67–76. PMID 18393863. doi:10.2174/138920108783955191.
