Estrutura das biomoléculas
 |
 |
A estrutura das biomoléculas é a forma tridimensional con intricados pregamentos que adopta unha molécula de proteína, ADN, ARN ou outras biomoléculas, e que é importante para a súa función. A estrutura destas moléculas pode considerarse a calquera escala, desde o nivel de átomos individuais ata as relacións entre subunidades proteicas completas. Esta útil distinción entre escalas adoita expresarse descompoñendo a estrutura molecular en catro niveis: estrutura primaria, secundaria, terciaria e cuaternaria. O armazón de toda esta organización a multiescalas da molécula orixínase no nivel da estrutura secundaria, onde os elementos estruturais fundamentais son os diversos enlaces de hidróxeno que establece a molécula. Isto orixina varios dominios recoñecibles na estrutura das proteínas e dos ácidos nucleicos, incluíndo características da estrutura secundaria como as hélices alfa e láminas beta das proteínas e os bucles en forquita, protuberancias e bucles internos dos ácidos nucleicos. Os termos estrutura primaria, secundaria, terciaria e cuaternaria foron introducidos por Kaj Ulrik Linderstrøm-Lang na súa obra de 1951 Lane Medical Lectures na Universidade Stanford.
Estrutura primaria
[editar | editar a fonte]- Artigos principais: Estrutura primaria das proteínas e Estrutura primaria dos ácidos nucleicos.
A estrutura primaria dun biopolímero é a especificación exacta da súa composición atómica e dos enlaces químicos que conectan eses átomos (incluíndo a estereoquímica). Para un biopolímero típico non ramificado e sen enlaces cruzados (como unha molécula dunha proteína intracelular típica ou de ADN ou ARN), a estrutura primaria é equivalente á secuencia das súas subunidades monómeras, como son os aminoácidos ou os nucleótidos, respectivamente.
A estrutura primaria dunha proteína indícase empezando polo amino N-terminal e rematando no carboxilo C-terminal da súa secuencia de aminoácidos, mentres que a estrutura primaria dunha molécula de ADN ou ARN indícase empezando polo extremo 5' da súa secuencia de nucleótidos e rematando polo extremo 3'. A secuencia dun ácido nucleico comprende os nucleótidos de toda a molécula, pero a miúdo dentro da estrutura primaria hai motivos de secuencia que teñen importancia funcional. Algúns exemplos de tales motivos son: as caixas C/D[1] e H/ACA[2] dos ARN nucleolares pequenos, o sitio de unión LSm que se encontra nos ARNs do espliceosoma como U1, U2, U4, U5, U6, U12 e U3, a secuencia Shine-Dalgarno,[3] a secuencia consenso Kozak[4] e o trerminador da ARN polimerase III.[5]
Estrutura secundaria
[editar | editar a fonte]- Artigos principais: Estrutura secundaria das proteínas e Estrutura secundaria dos ácidos nucleicos.
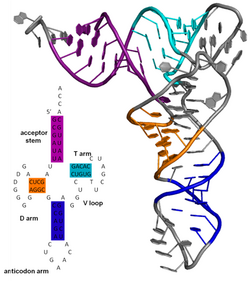
A estrutura secundaria das proteínas é o padrón de enlaces de hidróxeno das proteínas. Estes determinan a forma tridimensional xeral de segmentos locais da proteína, pero non describen a estrutura global da posición de átomos específicos no espazo tridimensional, que é o que se considera estrutura terciaria. A estrutura secundaria defínese formalmente como o padrón de enlaces de hidróxeno do biopolímero tal como se observa na estrutura a resolución atómica. Nas proteínas a estrutura secundaria defínese como os padróns de enlaces de hidróxeno entre os grupos amino e carboxilo do esqueleto da molécula (ao contrario, os enlaces de hidróxeno que se formen entre as cadeas laterais e a cadea principal ou entre dúas cadeas laterais son irrelevantes aquí), para o cal se utiliza a definición DSSP de enlace de hidróxeno.
A estrutura secundaria dos ácidos nucleicos está definida polos enlaces de hidróxeno que se establecen entre as bases nitroxenadas.
Porén, nas proteínas o padrón de enlaces de hidróxeno está correlacionado con outras características estruturais, que deron lugar a definicións menos formais pero moi utilizadas de estrutura secundaria. Por exemplo, as hélices poden adoptar ángulos diedros no seu esqueleto molecular nalgunhas rexións do gráfico de Ramachandran; deste modo, un segmento de residuos de aminoácidos con ditos ángulos diedros denomínase hélice, sen importar se ten o padrón correcto de enlaces de hidróxeno. Propuxéronse outras definicións menos formais, xeralmente aplicando conceptos de xeometría diferencial das curvas, como curvatura e torsión. Os biólogos estruturais que teñen que resolver unha nova estrutura con resolución atómica asígnanlle ás veces a súa estrutura secundaria a ollo e rexistran as súas asignacións no arquivo correspondente de Protein Data Bank (formato PDB).
A estrutura secundaria dun ácido nucleico refírese ás interaccións de apareamento de bases nunha molécula ou conxunto de moléculas que interaccionan. A estrutura secundaria dos ARNs biolóxicos pode a miúdo ser descomposta en soamente talos e bucles. A miúdo estes elementos ou combinacións deles poden ser clasificados en tipos, como tetrabucles, pseudonós e talos bucles. Hai moitos elementos de estrutura secundaria de importancia funcional nos ARNs biolóxicos. Exemplos famosos son os talos-bucles do terminador independente de rho e a folla de trevo do ARN de transferencia. Entre as estratexias usadas para determinar as estruturas secundarias dos ARNs están os métodos experimental e computacional.
Estrutura terciaria
[editar | editar a fonte]- Artigos principais: Estrutura terciaria das proteínas, Estrutura terciaria dos ácidos nucleicos e Aliñamento estrutural.
A estrutura terciaria dunha proteína ou calquera outra macromolécula é a súa estrutura tridimensional, definida polas súas coordenadas atómicas.[6] As proteínas e ácidos nucleicos préganse en complexas estruturas tridimensionais que son necesarias para o funcionamento das moléculas. Aínda que estas estruturas son diversas e complexas, a miúdo están compostas de motivos de estrutura terciaria e dominios recoñecibles que serven como bloques de construción molecular. A estrutura terciaria considérase que está determinada fundamentalmente pola sús estrutura primaria (a súa secuencia de aminoácidos ou nucleótidos).
Estrutura cuaternaria
[editar | editar a fonte]- Artigos principais: Estrutura cuaternaria das proteínas e Estrutura cuaternaria dos ácidos nucleicos.
A estrutura cuaternaria das proteínas é a disposición de múltiples moléculas proteicas que forman un complexo de multisubunidades.
O uso do termo é menos común en ácidos nucleicos, pero pode utilizarse para referirse á organización de alto nivel do ADN na cromatina,[7] incluíndo a súa interacción coas proteínas histonas, ou a interacción de unidades de ARN individuais no ribosoma[8][9] ou no espliceosoma.
Determinación da estrutura
[editar | editar a fonte]- Artigos principais: Estrutura das proteínas e Determinación da estrutura dos ácidos nucleicos.
A estrutura exploratoria é o proceso no cal se utilizan técnicas bioquímicas para determinar a estrutura biomolecular.[10] Esta análise pode utilizarse para definir os padróns que se poden usar para inferir a estrutura molecular, a análise exprerimental da estrutura molecular e a función, e comprender mellor o desenvolvemento de moléculas máis pequenas para futuras investigacións biolóxicas.[11] A análise exploratoria de estruturas pode facerse usando diferentes métodos, entre os que están o tenteo químico, o tenteo de radicais hidroxilo, o mapado de interferencia de análogos de nucleótidos (NAIM) e o tenteo en liña.[10]
As estruturas das proteínas e os ácidos nucleicos poden determinarse usando espectroscopia RMN ou cristalografía de raios X ou microscopia crioelectrónica dunha soa partícula (cryoEM). Os primeiros informes publicados para o ADN (feitos por Rosalind Franklin e Raymond Gosling en 1953) de padróns de difracción de raios X do ADN A (e tamén do B) usaron análises baseados nas transformadas da función de Patterson que proporcionaron soamente unha cantidade limitada de información estrutural para fibras orientadas do ADN illadas de timo de tenreiras.[12][13] Wilkins et al. en 1953 propuxeron despois unha análise alternativa para a difracción de raios X do ADN B e os padróns de difracción de febras de ADN hidratadas orientadas bacterianas e de espermatozoides de troita en termos de cadrados das funcións de Bessel.[14] Aínda que a forma B do ADN é a máis común baixo as condicións reinantes nas células,[15] non é unha conformación ben definida senón unha familia ou conxunto difuso de conformacións de ADN que aparecen aos altos niveis de hidratación presentes nunha ampla variedade de células vivas.[16] As súas correspondentes difraccións e dispersións de raios X son característicos dos paracristais moleculares cun grao significativo de desorde (dun 20%),[17][18] e a estrutura non se pode tratar usando só as análises estándar.
En contraste, a análise estándar, que implica só as transformadas de Fourier das funcións de Bessel[19] e os modelos moleculares do ADN, aínda se usa rutineiramente para analizar os padróns de difraccións dos ADNs A e Z.[20]
Predición da estrutura
[editar | editar a fonte]
- Artigos principais: Predición da estrutura das proteínas e Predición da estrutura dos ácidos nuclecicos.
A predición da estrutura biomolecular é a predición da estrutura tridimensional dunha proteína a partir dos seus aminoácidos, ou dun ácido nucleico a partir da súa secuencia de nucleobases. Noutras palabras, é a predición da estrutura secundaria e terciaria a partir da estrutura primaria. A predición de estruturas é o inverso do deseño biomolecular, como no deseño racional, deseño de proteínas, deseño de ácidos nucleicos e enxeñaría biomolecular.
A predición da estrutura das proteínas é un dos obxectivos máis importantes procurados pola bioinformática e a química teórica. A predición da estrutura das proteínas é de grande importancia en medicina (por exemplo, en deseño de fármacos) e biotecnoloxía (por exemplo, no deseño de novos encimas). Cada dous anos, avalíase o rendemento dos métodos actuais no experimento de Avaliación Crítica da Predición da Estrutura de Proteínas (ou CASP, do inglés Critical Assessment of protein Structure Prediction).
Houbo tamén un número significativo de investigacións bioinformáticas dirixida ao problema da predición da estrutura do ARN. Un problema común para os investigadores é determinar a estrutura tridimensional dun ácido nucleico sabendo só a súa secuencia de bases. Porén, no caso do ARN, gran parte da estrutura final está determinada pola estrutura secundaria ou as interaccións de apareamento de bases intramoleculares da molécula. Isto móstrase pola alta conservación do apareamento de bases entre distintas especies.
A estrutura secundaria de pequenas moléculas de ácidos nucleicos está determinada en gran parte por interaccións locais fortes como os enlaces de hidróxeno e o empillamento de bases. Sumando a enerxía libre desas interaccións, xeralmente usando un método do veciño máis próximo, proporciona unha aproximación para a estabilidade da estrutura dada.[21] O xeito máis directo de encontrar a estrutura de enerxía libre máis baixa sería xerar todas as estruturas posibles e calcular a enerxía libre para eles, pero o número de estruturas posibles para a secuencia increméntase exponencialmente coa lonxitude da molécula.[22] Para moléculas máis longas, o número de estruturas secundarias posibles é enorme.[21]
Os métodos de covariación da secuencia dependen da existencia dun conxunto de datos composto de múltiples secuencias de ARNs homólogos con secuencias relacionadas pero non semellantes. Estes métodos analizan a covariación dos sitios de bases individuais na evolución; o mantemento en dous sitios moi separados dun par de nucleótidos que se aparean indica a presenza dun enlace de hidróxeno requirido estruturalmente entre esas posicións. O problema xeral da predición de pseudonós mostrou ser NP-completo.[23]
Deseño
[editar | editar a fonte]- Artigos principais: Deseño de proteínas e Deseño de ácidos nucleicos.
O deseño biomolecular pode considerarse como o inverso da predición da estrutura. Na predición da estrutura esta determínase a partir dunha secuencia coñecida, mentres que no deseño de proteínas ou ácidos nucleicos xérase unha secuencia que formará unha estrutura desexada.
Outras biomoléculas
[editar | editar a fonte]- Véxase tamén: Bicapa lipídica.
Outras biomoléculas, como polisacáridos, polifenois e lípidos poden tamén ter estruturas de orde superior con consecuencias biolóxicas.
Notas
[editar | editar a fonte]- ↑ Samarsky DA, Fournier MJ, Singer RH, Bertrand E (xullo de 1998). "The snoRNA box C/D motif directs nucleolar targeting and also couples snoRNA synthesis and localization". The EMBO Journal 17 (13): 3747–57. PMC 1170710. PMID 9649444. doi:10.1093/emboj/17.13.3747.
- ↑ Ganot P, Caizergues-Ferrer M, Kiss T (abril de 1997). "The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation". Genes & Development 11 (7): 941–56. PMID 9106664. doi:10.1101/gad.11.7.941.
- ↑ Shine J, Dalgarno L (marzo de 1975). "Determinant of cistron specificity in bacterial ribosomes". Nature 254 (5495): 34–38. Bibcode:1975Natur.254...34S. PMID 803646. doi:10.1038/254034a0.
- ↑ Kozak M (outubro de 1987). "An analysis of 5'-noncoding sequences from 699 vertebrate messenger RNAs". Nucleic Acids Research 15 (20): 8125–48. PMC 306349. PMID 3313277. doi:10.1093/nar/15.20.8125.
- ↑ Bogenhagen DF, Brown DD (abril de 1981). "Nucleotide sequences in Xenopus 5S DNA required for transcription termination". Cell 24 (1): 261–70. PMID 6263489. doi:10.1016/0092-8674(81)90522-5.
- ↑ "tertiary structure". Compendium of Chemical Terminology (the "Gold Book") (2ª ed.). IUPAC. (2006–) [1997].
- ↑ Sipski ML, Wagner TE (marzo de 1977). "Probing DNA quaternary ordering with circular dichroism spectroscopy: studies of equine sperm chromosomal fibers". Biopolymers 16 (3): 573–82. PMID 843604. doi:10.1002/bip.1977.360160308.
- ↑ Noller HF (1984). "Structure of ribosomal RNA". Annual Review of Biochemistry 53: 119–62. PMID 6206780. doi:10.1146/annurev.bi.53.070184.001003.
- ↑ Nissen P, Ippolito JA, Ban N, Moore PB, Steitz TA (abril de 2001). "RNA tertiary interactions in the large ribosomal subunit: the A-minor motif". Proceedings of the National Academy of Sciences of the United States of America 98 (9): 4899–903. Bibcode:2001PNAS...98.4899N. PMC 33135. PMID 11296253. doi:10.1073/pnas.081082398.
- ↑ 10,0 10,1 Teunissen, A. W. M. (1979). RNA Structure Probing: Biochemical structure analysis of autoimmune-related RNA molecules. pp. 1–27. ISBN 978-90-901323-4-1.
- ↑ Pace NR, Thomas BC, Woese CR (1999). Probing RNA Structure, Function, and History by Comparative Analysis. Cold Spring Harbor Laboratory Press. pp. 113–17. ISBN 978-0-87969-589-7.
- ↑ Franklin RE, Gosling RG (6 de marzo de 1953). "The Structure of Sodium Thymonucleate Fibres (I. The Influence of Water Content, and II. The Cylindrically Symmetrical Patterson Function)" (PDF). Acta Crystallogr. 6 (8): 673–78. doi:10.1107/s0365110x53001939.
- ↑ Franklin RE, Gosling RG (abril de 1953). "Molecular configuration in sodium thymonucleate". Nature 171 (4356): 740–41. Bibcode:1953Natur.171..740F. PMID 13054694. doi:10.1038/171740a0.
- ↑ Wilkins MH, Stokes AR, Wilson HR (abril de 1953). "Molecular structure of deoxypentose nucleic acids". Nature 171 (4356): 738–40. Bibcode:1953Natur.171..738W. PMID 13054693. doi:10.1038/171738a0.
- ↑ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (outubro de 1980). "Polymorphism of DNA double helices". Journal of Molecular Biology 143 (1): 49–72. PMID 7441761. doi:10.1016/0022-2836(80)90124-2.
- ↑ Baianu, I. C. (1980). "Structural Order and Partial Disorder in Biological systems". Bull. Math. Biol. 42 (1): 137–41. doi:10.1007/BF02462372.
- ↑ Hosemann R, Bagchi RN (1962). Direct analysis of diffraction by matter. Amsterdam/New York: North-Holland.
- ↑ Baianu IC (1978). "X-ray scattering by partially disordered membrane systems". Acta Crystallogr. A 34 (5): 751–53. Bibcode:1978AcCrA..34..751B. doi:10.1107/s0567739478001540.
- ↑ "Bessel functions and diffraction by helical structures.". planetphysics.org.[Ligazón morta]
- ↑ "X-Ray Diffraction Patterns of Double-Helical Deoxyribonucleic Acid (DNA) Crystals". planetphysics.org. Arquivado dende o orixinal o 24 de xullo de 2009.
- ↑ 21,0 21,1 Mathews DH (xuño de 2006). "Revolutions in RNA secondary structure prediction". Journal of Molecular Biology 359 (3): 526–32. PMID 16500677. doi:10.1016/j.jmb.2006.01.067.
- ↑ Zuker M, Sankoff D (1984). "RNA secondary structures and their prediction". Bull. Math. Biol. 46 (4): 591–621. doi:10.1007/BF02459506.
- ↑ Lyngsø RB, Pedersen CN (2000). "RNA pseudoknot prediction in energy-based models". Journal of Computational Biology 7 (3–4): 409–27. PMID 11108471. doi:10.1089/106652700750050862.

