CRISPR (edición de xenes)
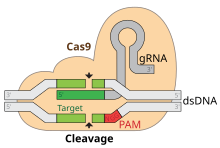
A edición de xenes por CRISPR (adoita pronunciarse crísper) é unha técnica de enxeñaría xenética en bioloxía molecular por medio da cal pode modificarse o xenoma dos organismos vivos. CRISPR é a abreviatura en inglés de Clustered Regularly Interspaced Short Palindromic Repeats, repeticións palindrómicas curtas agrupadas e espazadas regularmente, unha familia de secuencias de ADN dos procariotas derivadas de fragmentos de bacteriófagos que as infectaran. A técnica está baseada nunha versión simplificada do sistema de defensa antiviral bacteriano CRISPR-Cas9, usado polos procariotas para destruír ADN de bacteriófagos. Con este método, ao introducir nunha célula a nuclease Cas9 en complexo cun ARN guía (ARNg ou, en inglés, gRNA) sintético, pode cortarse o xenoma da célula no lugar desexado, o que permite eliminar xenes existentes ou engadir xenes novos in vivo.[1]
Esta técnica considérase moi importante en biotecnoloxía e medicina, xa que permite editar xenomas in vivo con gran precisión, facilidade e baixo custo. Pode utilizarse na creación de novas medicinas, produtos agrícolas e organismos modificados xeneticamente, ou como medio de controlar patóxenos e pragas. Tamén ten posibilidades no tratamento de enfermidades conxénitas herdadas xeneticamente, así como de doenzas que se orixinan por mutacións somáticas, como o cancro. Porén, o seu uso na modificación xenética da liña xerminal humana é moi polémica. O desenvolvemento da técnica serviulles a Jennifer Doudna e Emmanuelle Charpentier para gañar o Premio Nobel de Química de 2020.[2][3] O terceiro grupo de investigadores que compartiu o premio Kavli polo mesmo descubrimento,[4] liderado por Virginijus Šikšnys, non foi galardoado co premio Nobel.[5][6][7]
A nuclease Cas9, que funciona como tesoira xenética, abre ambas as febras dunha secuencia diana de ADN para introducir a modificación por medio dun de dous posibles métodos. As mutacións knock-in, facilitadas por reparación dirixida por homoloxía (HDR), son a vía tradicional usada polas estratexias de edición xenómica dirixida a unha diana.[1] Isto permite a introdución de danos e reparacións no ADN dirixidas a unha diana. A HDR emprega secuencias de ADN similares para reparar as roturas por incorporación de ADN exóxeno que funciona como molde para a reparación.[1] Este método depende de que ocorran danos no ADN periódicos e illados no sitio diana para que comece a reparación. As mutacións knock-out causadas por CRISPR-Cas9 orixínanse pola reparación da rotura de dobre febra no ADN por medio de unión de extremos non homólogos (NHEJ) ou unión de extremos mediada pola POLQ/ polimerase theta (TMEJ). Estas dúas vías de unión de extremos poden con frecuencia orixinar delecións ou insercións aleatorias no sitio de reparación, o cal pode impedir ou alterar o funcionamento do xene. Polo tanto, a enxeñaría xenómica por CRISPR-Cas9 dálle aos investigadores a capacidade de xerar a alteración aleatoria dun xene diana. Debido a isto, a precisión da edición do xenoma é unha gran preocupación. A edición do xenoma orixina cambios irreversibles no xenoma.
Aínda que a edición xenómica en células eucariotas foi posible usando varios métodos desde a década de 1980, os métodos empregados foran ineficientes e pouco prácticos para aplicalos a grande escala. Co descubrimento de CRISPR e especificamente a molécula de nuclease Cas9, a posibilidade dunha edición eficiente e moi selectiva é agora unha realidade. A Cas9 derivada da especie bacteriana Streptococcus pyogenes facilitou a modificación xenómica dirixida a diana en células eucariotas ao permitir un método fiable de crear unha rotura dirixida a un lugar específico designado polas febras guía de ARNtracr (en inglés tracrRNA) e o ARNcr (en inglés crRNA).[8] A facilidade coa cal os investigadores poden inserir a Cas9 e o molde de ARN para silenciar ou causar mutacións puntuais en loci específicos é moi valiosa para un rápido e eficiente mapado de modelos xenómicos e procesos biolóxicos asociados con varios xenes en diversos eucariotas. Desenvolvéronse variantes novas feitas por enxeñaría da nuclease Cas9 que reducen significativamente a actividade fóra de diana.[9]
As técnicas de edición do xenoma CRISPR-Cas9 teñen moitas aplicacións potenciais, incluíndo en medicina e agricultura. O uso do complexo CRISPR-Cas9-ARNg para a edición de xenomas[10] foi elixido pola Asociación Americana para o Avance da Ciencia (AAAS) como descubrimento do ano en 2015.[11] Expresáronse moitas cuestións bioéticas sobre a perspectiva do uso de CRISPR para a edición da liña xerminal, especialmente en embrións humanos.[12]
Historia[editar | editar a fonte]
Outros métodos[editar | editar a fonte]
A inicios da década de 2000 investigadores alemáns empezaron a desenvolver as nucleases de dedo de cinc (ZFNs), que son proteínas sintéticas cuxos dominios de unión ao ADN lles permiten crear roturas de dobre febra no ADN en sitios específicos. As nucleases de dedo de cinc teñen unha maior precisión e a vantaxe de ser menores que Cas9, pero non se usan tan comunmente coma os métodos baseados en CRISPR. En 2010, as nucleases sintéticas chamadas nucleases efectoras similares a un activador da transcrición (TALENs) proporcionaron un modo máis doado de tomar como diana unha rotura de dobre febra nunha localización específica da febra de ADN. Tanto as nucleases de dedo de cinc coma as TALENs requiren o deseño e creación dunha proteína á medida para cada secuencia diana de ADN, o cal é un proceso moito máis difícil e leva máis tempo que deseñar ARNs guías. Os CRISPRs son moito máis fáciles de deseñar porque o proceso require sintetizar só unha curta secuencia de ARN, un procedemento que xa se usa moito noutras técnicas de bioloxía molecular (por exemplo, para crear cebadores oligonucleótidos).[13]
Mentres que métodos como a interferencia de ARN (RNAi) non suprimen totalmente a función xénica, CRISPR, as ZFNs e as TALENs proporcionan un knockout de xenes completo e irreversible.[14] CRISPR pode tamén tomar como diana varios sitios do ADN simultaneamente simplemente introducindo diferentes ARNg. Ademais, o custo de empregar CRISPR é relativamente baixo.[14][15][16]
Descubrimento[editar | editar a fonte]
En 2012 Jennifer Doudna, Emmanuelle Charpentier, Martin Jínek, Krzysztof Chyliński, Ines Fonfara e Michael Hauer publicaron os seus descubrimentos de que o CRISPR-Cas9 podía programarse con ARN para editar o ADN xenómico, agora considerado un dos descubrimentos máis significativos na historia da bioloxía.[17][18]
Patentes e comercialización[editar | editar a fonte]
As patentes parqa CRISPR pasaron por varias vicisitudes.
En novembro de 2013, SAGE Labs (parte do grupo Horizon Discovery) tiña dereitos exclusivos dunha desas compañías para producir e vender ratos modificados xeneticamente e dereitos non exclusivos sobre modelos de ratos e coelllos.[19] En 2015 Thermo Fisher Scientific tiña licenza de propiedade intelectual de ToolGen para desenvolver kits de reactivos CRISPR.[20]
En decembro de 2014 os dereitos de patente para CRISPR foron impugnados. Formáronse varias compañías para desenvolver fármacos e ferramentas de investigación.[21] A medida que as compañías incrementaron o seu financiamento, empezaron a formularse dúbidas sobre se o CRISPR podería ter un rendemento económico rápido.[22] En 2014 Feng Zhang do Broad Institute do MIT e Harvard e outros nove recibiron a patente estadounidense número 8.697.359[23] sobre o uso da edición de xenes por CRISPR–Cas9 en eucariotas. Aínda que Charpentier e Doudna (grupo CVC) foron recoñecidos como os que concibiron CRISPR, o Broad Institute foi o primeiro que conseguiu unha "redución á práctica" de acordo cos xuíces e xuízas da patente Sally Gardner Lane, James T. Moore e Deborah Katz.[24]
O primeiro conxunto de patentes concedéuselle ao equipo do Broad Institute en 2015, o que motivou que os avogados do grupo CVC interpuxesen o primeiro procedemento de interferencia.[25] En febreiro de 2017 a Oficina de Patentes dos EUA (USPTO) xulgou un caso de interferencia de patente presentado pola Universidade de California con respecto a patentes concedidas ao Broad Institute, e atopou que as patentes de Broad, que afirmaban comprendian a aplicación de CRISPR-Cas9 en células eucariotas, eran distintas das invencións aducidas pola Universidade de California.[26][27][28]
Pouco despois, a Universidade de California presentou unha apelación a este pronunciamento.[29][30] En 2019 iniciouse un segundo preito de interferencia. Foi unha resposta a aplicacións da patente feitas por CVC que solicitou ao tribunal de apelación determinar cal era o inventor orixinal desta tecnoloxía. A USPTO sentenciou en marzo de 2022 contra a Universidade de California, establecendo que o Broad Institute foi o primeiro en rexistrala. A decisión afectou moitos dos acordos de licenzas para a tecnoloxía de edición por CRISPR que foran concedidas pola Universidade de Californa, Berkeley. A Universidade de California declarou a súa intención de apelar o dictame ao USPTO.[31]
En Europa[editar | editar a fonte]
En marzo de 2017 a Oficina de Patentes Europea (OPE) anunciou a súa intención de dar dereito de editar todos os tipos de células ao Instituto Max-Planck de Berlín, á Universidade de California e á Universidade de Viena,[32][33] e en agosto de 2017, a OPE anunciou a súa intención de dar dereitos sobre CRISPR nunha aplicación de patente presentada por MilliporeSigma.[32] En agosto de 2017 a situación da patente en Europa era complexa, con MilliporeSigma, ToolGen, a Universidade de Vilnius e Harvard disputándose os dereitos, xunto coa Universidade de Califora e Broad.[34]
En xullo de 2018, o Tribunal Europeo de Xustiza sentenciou que a edición de xenes de plantas era unha subcategoría dos alimentos OMG e, polo tanto, que a técnica CRISPR sería regulada de aí en adiante na Unión Europea polas súas regras e regulacións para os organismos modificados xeneticamente (OMG).[35]
Sucesos recentes[editar | editar a fonte]
En febreiro de 2020, un ensaio en EUA mostrou que a edición de xenes por CRISPR era segura en tres pacientes de cancro.[36]
En outubro de 2020 as investigadoras Emmanuelle Charpentier e Jennifer Doudna recibiron o Premio Noble de Química polo seu traballo no campo dos CRISPR.[37][38] Fixeron historia por ser as dúas primeiras mulleres que compartiron este premio sen compartilo tamén cun home.[39][5]
En xuño de 2021 concluíu un primeiro pequeno ensaio clínico da edición de xenes por CRISPR intravenosa en humanos con resultados prometedores.[40][41]
En setembro de 2021 o primeiro alimento editado con CRISPR saíu á venda pública no Xapón. Eran uns tomates modificados xeneticamente para teren unhas cinco veces a cantidade normal do neurotransmisor con posible efecto calmante[42] GABA (un neurotransmisor inhibitorio).[43] CRISPR fora aplicado por primeira vez en tomates en 2014.[44]
En decembro de 2021 informouse de que o primeiro alimento animal mariño con edición de xenes por CRISPR e un un segundo conxunto de alimentos editados con CRISPR saíron á venda no Xapón: creáronse dous peixes, un dos cales crecía case o dobre do normal, pola alteración da leptina, a cal controla o apetito, e o outro crecía ao 1,2 do seu tamaño natural coa mesma cantidade de comida debido á alteración da miostatina, que inhibe o crecemento muscular.[45][46][47]
Un estudo de 2022 sobre a preferencia dos consumidores de comprar tomates CRISPR atopou que darlles a coñecer máis sobre os tomates CRISPR tivo un forte efecto positivo sobre a prefeencia dos participantes. "Case a metade dos 32 participantes de Alemaña que eran científicos demonstraron eleccións constantes, mentres que a maioría mostrou un aumento na súa intención de comprar tomates CRISPR, principalmente non científicos."[48][49]
A Universidade de California, Berkeley anunciou en maio de 2021 o seu intento de poxa de token non funxible de ambas as patentes de edición de xenes por CRISPR así como da inmunoterapia do cancro, aínda que a universidade conservaría neste caso a propiedade das patentes.[50][51] O 85 % dos fondos reunidos polas vendas da colección chamada The Fourth Pillar (O cuarto piar) ían ser utilizados para financiar investigacións.[52][53] Vendeuse en xuño de 2022 por 54.000 dólares.[54]
Enxeñaría xenómica[editar | editar a fonte]

A edición do xenoma por CRISPR-Cas9 lévase a cabo cun sistema CRISPR de tipo II. Cando se utiliza para a edición do xenoma, este sistema inclúe unha ribonucleoproteína (RNP), que consta de Cas9, ARNcr (en inglés crRNA), e ARNtracr (en inglés tracrRNA), xunto cun molde para a reparación do ADN opcional.

Principais compoñentes[editar | editar a fonte]
| Compoñente | Función |
|---|---|
| ARNcr (ARN CRISPR) | Contén o ARN guía que localiza o segmento correcto do ADN do hóspede xunto cunha rexión que se une ao ARNtracr (xeralmente nunha forma de bucle en forquita), formando un complexo activo. |
| ARNtracr (ARNcr transactivante) | Únese ao ARNcr e forma un complexo activo. |
| ARNgs (sgRNA en inglés) | Os ARNs guías simples ou únicos (single guide RNA) son uns ARNs combinados que constan dun ARNtracr e polo menos un ARNcr. |
| Cas9 (o máis común) | Un encima cuxa forma activa pode modificar o ADN. Existen moitas variantes con diferentes funcións (é dicir, encima que forma unha amosega nunha soa das febras, que crea roturas de dobre febra, que se une ao ADN) debido á función do sitio de recoñecemento do ADN de cada encima. |
| Molde de reparación | Molécula de ADN usada como molde no proceso de reparación do ADN da célula hóspede, que permite a inserción dunha secuencia de ADN específica no segmento hóspede roto por Cas9. |
O CRISPR-Cas9 a miúdo emprega plásmidos, que codifican os compoñentes da RNP, para a transfección das células diana, ou a RNP ensámblase antes da adición ás células por medio de nucleofección.[55] Os principais compoñentes deste plásmido están representados na imaxe e listados na táboa. O ARNcr deséñase expresamente para cada aplicación, xa que esta é a secuencia que a Cas9 usa para identificar e unirse directamente a secuencias específicas dentro do ADN da célula hóspede. O ARNcr debe unirse soamente no lugar que se desexa. O molde de reparación é tamén deseñado expresamente para cada aplicación, xa que debe complementar ata certo punto as secuencias de ADN a cada lado do corte e tamén contén calquera secuencia que se desexe para a inserción no xenoma hóspede.
Poden empaquetarse xuntos múltiples ARNcr e o ARNtracr para formar un ARN guía simple ou único (ARNgs).[56] Este ARNgs pode incluírse xunto ao xene que codifica a proteína Cas9 e formar un plásmido para ser transfectado ás células. Disponse de moitas ferramentas on line para axudar no deseño de secuencias de ARNgs efectivas.[57][58]

Alternativas á Cas9[editar | editar a fonte]
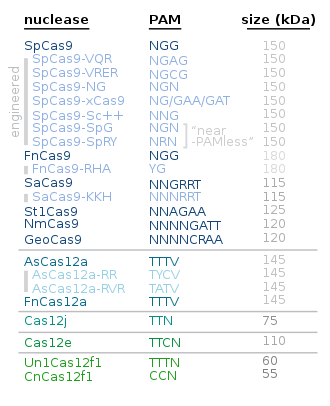
Entre as proteínas alternativas a Cas9 están as seguintes:
| Proteína | Principal uso / características | Ano/s |
|---|---|---|
| Cas12 | Cas12a é máis pequena e simple que Cas9; Cas12b i.a. para a enxeñaría xenética en plantas[59][60] | |
| Cas13 | para a edición do ARN[61] | |
| Cas3[62][63] | Crea un amplo tramo monocatenario[64] | 2019 |
| CasMINI | Unhas dúas veces máis compacta que a máis frecuentemente usada Cas9 e Cas12a.[65][66] | 2021 |
| SuperFi-Cas9 | Máis exacta sen deceleración da velocidade[67][68] | 2022 |
| Cas7-11 | Máis axeitada para a edición do ARN terapéutica que a Cas13[69] | 2022 |
| Reparación do ADN tomando como molde o cromosoma homólogo | Este método só é aplicable a organismos cuxo cromosoma correspondente ten os xenes desexados.
|
2022 |
Estrutura[editar | editar a fonte]
CRISPR-Cas9 ofrece un alto grao de fidelidade e a súa construción é relativamente simple. Depende de dous factores para a súa especificidade: a secuencia diana e a secuencia motivo adxacente protoespazador (PAM, do inglés protospacer adjacent motif). A secuencia diana é dunha lonxitude de 20 bases como parte de cada locus CRISPR na matriz de ARNcr.[55] Unha matriz de ARNcr típica ten múltiples secuencias diana únicas. As proteínas Cas9 seleccionan a localización correcta no xenoma do hóspede utilizando a secuencia para unirse cos pares de bases do ADN hóspede. A secuencia non forma parte da proteína Cas9 e como resultado é personalizable e pode ser sintetizada independentemente.[72][73]
A secuencia PAM no xenoma hóspede é recoñecida por Cas9. Cas9 non pode ser doadamente modificada para recoñecer unha secuencia PAM diferente. Porén, isto no fin de contas non é demasiado limitante, xa que é tipicamente unha secuencia moi curta e non específica que aparece frecuentemente en moitos lugares por todo o xenoma (por exemplo, a secuencia PAM SpCas9 é 5'-NGG-3', que no xenoma humano aparece aproximadamente cada 8 a 12 pares de bases).[55]
Unha vez que estas secuencias foron ensambladas nun plásmido e transfectadas nas células, a proteína Cas9 coa axuda do ARNcr encontra a secuencia correcta no ADN da célula hóspede e (dependendo da variante da Cas9) crea unha rotura do febra simple ou dobre na localización apropiada no ADN.[74]
As roturas dunha soa febra espazadas adecuadamente no ADN hóspede poden activar a reparación dirixida por homoloxía, a cal é menos tendente ao erro que a unión de extremos non homólogos que segue tipicamente a unha rotura de dore febra. Proporcionar un molde para a reparación permite a inserción dunha secuencia de ADN específica nunha localización exacta dentro do xenoma. O molde de reparaión debería estenderse de 40 a 90 pares de bases alén da rotura inducida por Cas9.[55] O obxectivo é que o proceso de reparación dirixida por homoloxía da célula utilice o molde de reparación proporcionado e, por tanto, incorpore a nova secuencia no xenoma. Unha vez incorporada, esta nova secuencia é agora parte do material xenético da célula e transmítese ás células fillas. A inhibición transitoria combinada da unión de extremos non homólogos por unha molécula de ARN interferente pequeno pode incrementar a eficiencia da reparación dirixida por homoloxía ata nun 93 % e impedir simultaneamente a edición fóra de diana.[75]
Entrega[editar | editar a fonte]
- Véxase tamén: Transfección.
A entrega ás células da Cas9, o ARNgs, e os complexos asociados pode realizarse por medio dun virus ou por sistemas non virais. A electroporación do ADN, ARN ou ribonucleocomplexos é unha técnica común, aínda que pode ter efectos daniños nas células diana.[76] Utilizáronse técnicas de transfección química que utilizan lípidos e péptidos para introducir nas células os ARNgs en complexo con Cas9.[77][78] A entrega baseada en nanopartículas tamén se utilizou para a transfección.[79] Os tipos de células que son máis difíciles de transfectar (por exemplo, células nais, neuronas e células hematopoéticas) requiren sistemas de entrega máis eficientes, como os baseados en lentivirus (LVs), adenovirus (AdV) e virus adeno-asociados (AAV).[80][81][82]
A eficiencia da CRISPR-Cas9 increméntase moito cando varios compoñentes do sistema incluíndo a estrutura CRISPR/Cas9 enteira e os complexos Cas9-ARNg son entregados ensamblados en vez de usando métodos transxénicos.[83][84] Isto ten especial valor en plantas agrícolas modificadas xeneticamente para a comercialización en masa.[85][86] Como non se necesita a maquinaria de replicación do hóspede para producir estas proteínas, as posibilidades da secuencia de recoñecemento do ARNgs son case nulas, diminuíndo a probabilidade de efectos fóra de diana.[79]
Edición do xenoma controlada[editar | editar a fonte]
Outras melloras e variantes do sistema CRISPR-Cas9 centráronse en maximizar o control no seu uso. Concretamente, preténdese mellorar a súa especificidade, eficiencia e granularidade do seu poder de edición. As técnicas poden ser subdivididas e clasificadas polo compoñente do sistema que modifican. Estas inclúen o uso de diferentes variantes ou novas creacións da proteína Cas, o uso dunha proteína efectora totalmente diferente, a modificación do ARNgs ou o uso dunha estratexia algorítmica para identificar as solucións óptimas.
A especificidade é un importante aspecto para mellorar o sistema CRISPR-Cas9 porque os efectos fóra de diana que xera teñen graves consecuencias para o xenoma da célula e aconsellan precaución no seu uso. Minimizar os efectos fóra de diana é así maximizar a seguridade do sistema. Novas variacións das proteínas Cas9 que incrementan a especificidade inclúen as proteínas efectoras con eficiencia e especificidade comparables ás da SpCas9 orixinal, que poden dirixirse a secuencias diana previamente non alcanzables, e unha variante que virtualmente non ten mutacións fóra de diana.[87][88] Tamén se investigou a preparación por enxeñaría de novas proteínas Cas9, incluíndo algunhas que substitúen parcialmente ribonucleótidos en ARNcr por desoxirribonucleótidos (de ADN), e un mutante de Cas9 guiado a estrutura, todos os cales teñen efectos fóra de diana reducidos.[89][90] Os ARNgs repetidamente truncados e os ARNg moi estabilizados tamén diminúen os efectos fóra de diana.[91][92] Os métodos computacionais incluíndo a aprendizaxe de máquina utilizáronse para predicir a afinidade e crear secuencias únicas para o sisema para maximizar a especificidade para unha diana dada.[93][94]
Varias variantes de CRISPR-Cas9 permiten a activación de xenes ou edición do xenoma cun causante externo como a luz ou pequenas moléculas.[95][96][97] Estes inclúen sistemas CRISPR fotoactivables desenvolvidos fusionando proteínas sensibles á luz cun dominio activador e unha dCas9 para a activación xénica,[98][99] ou fusionar dominios sensibles á luz similares con dous construtos de split-Cas9,[100][101] ou incorporando aminoácidos non naturais engaiolados á Cas9,[102] ou modificando os ARNs guías con complementos fotoactivables para a edición do xenoma.[103]
Entre os métodos de control da edición do xenoma con pequenas moléculas están unha Cas9 alostérica, sen ningunha edición de fondo detectable, que activa a unión e corte coa adición de 4-hidroxitamoxifén (4-HT),[95] unha Cas9 ligada a inteína sensible a 4-HT,[104] ou unha Cas9 que é sensible a 4-HT cando se fusiona a catro dominios ERT2.[105] A split-Cas9 inducible por inteína permite a dimerización de fragmentos de Cas9[106] e o sistema de split-Cas9 inducible por rapamicina foi desenvolvido fusionando dous construtos de split-Cas9 con fragmentos de FRB e FKBP.[107] Outros estudos conseguiron inducir a transcrición de Cas9 cunha pequena molécula, a doxiciclina.[108][109] Tamén se poden usar pequenas moléculas para mellorar a reparación dirixida por homoloxía,[110] a miúdo inhibindo a vía da unión de extremos non homólogos e/ou a vía da unión de extremos mediada por theta.[111][112] Creouse un sisema coa proteína efectora Cpf1 que é inducido polas pequenas moléculas VE-822 e AZD-7762.[113] Estes sistemas permiten o control condicional da actividade de CRISPR para mellorar a precisión, eficiencia e control espazotemporal. O control espazotemporal é unha forma de eliminar os efectos fóra de diana, xa que só certas células ou partes do organismo necesitan ser modificadas, e así poden utilizarse a luz ou pequenas moléculas como modo de realizalo. A eficiencia do sistema CRISPR-Cas9 aumentouse tamén grandemente coa entrega axeitada de instrucións do ADN para crear as proteínas e reactivos necesarios.[113]
O CRISPR tamén utiliza proteínas editoras dun só par de bases para crear edicións específicas nunha ou dúas bases da secuencia diana. Fusionouse CRISPR/Cas9 con encimas específicos que inicialmente só podían cambiar mutacións de C por T e G por A e as súas inversas. Isto realizouse finalmente sen necesidade de ningún corte no ADN.[114][115][116] Coa fusión doutro encima, o sistema CRISPR-Cas9 de edición de bases pode tamén editar C por G e o seu inverso.[117]
Cribado CRISPR[editar | editar a fonte]
As repeticións palindrómicas curtas agrupadas espazadas regularmente (CRISPR, do inglés clustered regularly interspaced short palindrome repeats) do sistema CRISPR/Cas9 son unha tecnoloxía de edición de xenes que pode inducir roturas de dobre febra en calquera punto no que se poidan unir ácidos ribonucleicos guías (ARNg) coa secuencia PAM (motivo adxacente protoespazador, en inglés protospacer adjacent motif).[118] Os mutantes de sitio activo de Cas9 poden inducir tamén amosegas nunha soa febra,[119] tamén coñecidos como nickases Cas9.[120] Simplemente cambiando a secuencia do ARNg, a endonuclease Cas9 pode entregarse a un xene de interese e inducir roturas de dobre febra.[121] A eficiencia da endonuclease Cas9 e a facilidade coa cal se poden alcanzar os xenes diana levou ao desenvolvemento de bibliotecas de CRISPR-knockout (KO) tanto en células de ratos coma humanas, as cales poden cubrir conxuntos de xenes específicos de interese ou mesmo o xenoma completo.[122][123] O cribado CRISPR axudou a crear unha perturbación xenética sistemática e de alto rendemento en organismos modelo. Esta perturbación xnética é necesaria para comprender a función do xene e a súa regulación epixenética.[124] A vantaxe das librarías CRISPR de pools é que poden alcanzarse máis xenes diana á vez.
As bibliotecas knock-out créanse de maneira que teñan igual representación e actuación con todos os ARNg e porten un marcador de selección fluorescente ou antibiótico que pode utilizarse para recuperar células transducidas.[118] Hai dous sistemas de plásmidos nas bibliotecas CRISPR/Cas9. No primeiro está todo nun plásmido, no que o ARNgs e o Cas9 son producidos simultaneamente nunha célula transfectada. O segundo é un sistema de dous vectores: o ARNgs e os plásmidos Cas9 son entregados separadamente.[124] É importante entregar miles de vectores que conteñen ARNgs únicos nun só recipiente de células por transdución viral a baixa multiplicidade de infección (tipicamente de 0,1–0,6), porque prevén a probabilidade de que un clon de células individual tome máis dun tipo de ARNgs, xa que doutro modo isto pode levar a unha asignación incorrecta de xenotipo a fenotipo.[122]
Unha vez que se prepara a bibliotecas de pools é necesario levar a cabo unha secuenciación profunda (secuenciación de seguinte xeración) de ADN de plásmido amplificado por PCR para revelar a abundancia de ARNgs. As células de interese poden ser infectadas consecuentemente pola biblioteca e despois seleccionadas de acordo co fenotipo. Hai dous tipos de selección: negativa e positiva. Por selección negativa detéctanse eficientemente as células mortas ou de lento crecemento. Poden identificar os xenes esenciais de supervivencia, que poden servir como candidatos para fármacos dirixidos a diana molecularmente. Por outra parte, a selección positiva dá unha conxunto de poboacións que adquiriron vantaxes de crecemento por mutaxénese aleatoria.[118] Despois da selección o ADN xenómico é recollido e secuenciado por secuenciación de seguinte xeración. O empobrecemento ou enriquecemento de ARNgs detéctase e compárase coa biblioteca de ARNgs orixinal, anotada co xene diana ao que corresponda o ARNgs. Despois, a análise estatística identifica xenes que é significativamente probable que sexan relevantes para o fenotipo de interese.[122]
| Biblioteca | ID | Especie | PI | Xenes diana | Número de ARNg por xene | ARNg totais |
|---|---|---|---|---|---|---|
| Biblioteca de CRISPR knockout de rato Bassik | 1000000121–1000000130 | Rato | Bassik | Varía (~23.000 en total) | ~10 | Varía |
| Biblioteca de CRISPR knockout do xene supresor de tumores de rato | 113584 EFS backbone
113585 TBG backbone |
Rato | Chen | 56 | ~4 | 286 |
| Biblioteca de todo o xenoma de ratos Brie | 73632 (1 plásmido)
73633 (2 plásmido) |
Rato | Doench and Root | 19.674 | 4 | 78.637 |
| Biblioteca de CRISPR knockout humana Bassik | 101926–101934 | Humanos | Bassik | Varía (~20.500 en total) | ~10 | Varía |
| Biblioteca de todo o xenoma humana Brunello | 73179 (1 plásmido)
73178 (2 plásmidos) |
Humanos | Doench and Root | 19.114 | 4 | 76.441 |
| Biblioteca de knockout de todo o xenoma mini-humana baseada en AsCpf1 | 130630 | Humanos | Draetta | 16.977 | 3–4 | 17.032 matrices |
Á parte das bibliotecas de knockout hai tamén bibliotecas de knock-down (CRISPRi) e de activación (CRISPRa), que usan a capacidade das Cas9-proteínas de fusión desactivadas proteoliticamente (dCas9) de unirse ao ADN diana, o que significa que o xene de interese non é cortado senón que é sobreexpresado ou reprimido. Isto fai ao sistema CRISPR/Cas9 aínda máis interesante para a edición de xenes. A proteína dCas9 inactiva modula a expresión xénica ao dirixir represores ou activadores de dCas9 cara a sitios de comezo da transcrición ou promotores dos xenes diana. Para reprimir xenes, Cas9 pode fusionarse ao dominio efector KRAB que fai un complexo co ARNg, mentres que CRISPRa utiliza dCas9 fusionada a diferentes dominios de activación transcricional, que son ademais dirixidos polo ARNg a rexións promotoras para regular á alza a expresión.[126][127][128]
Aplicacións[editar | editar a fonte]
Modelos de enfermidades[editar | editar a fonte]
A modificación xenética por Cas9 permite unha xeración rápida e eficiente de modelos transxénicos dentro do campo da xenética. Cas9 pode ser facilmente introducido nas células diana xunto co ARNgs por medio de transfección de plásmidos para modelizar o espallamento de enfermidades, a resposta das células e a defensa contra a infección.[129] A capacidade de Cas9 para ser introducida in vivo permite a creación de modelos máis axeitados da función xénica e efectos das mutacións, mentres que se evitan a mutacións fóra de diana observadas tipicamente nos métodos máis vellos de enxeñaria xenética.
A revolución que supuxo CRISPR e Cas9 nos modelos xenómicos non afecta só os mamíferos. Os modelos xenómicos tradicionais como Drosophila melanogaster, un dos primeiros organismos modelo, fixéronse máis requintados na súa resolución co uso de Cas9.[129] Cas9 usa promotores específicos de célula, o que permite un uso controlado de Cas9. O método da Cas9 é axeitado para tratar doenzas debido a que a acción do encima Cas9 só afecta a certos tipos celulares. As células que sofren a terapia Cas9 poden tamén ser extraídas e reintroducidas para proporcionar efectos amplificados da terapia.[130]
CRISPR-Cas9 pode utilizarse para editar o ADN de organismos in vivo e para eliminar xenes concretos ou mesmo cromosomas enteiros dun organismo en calquera momento do seu desenvolvemento. Entre os cromosomas que foron eliminados con éxito in vivo usando as técnicas CRISPR están o cromosoma Y e o X de ratos de laboratorio adultos e os cromosomas humanos 14 e 21; o primeiro en liñas de células nai embrionarias de ratos aneuploides cun cromosoma humano extra, e o segundo en células nais pluripotentes inducidas humanas. Este método podería ser útil para o tratamento de trastornos xenéticos causados por ter un número anormal de cromosomas, como a síndrome de Down e os trastornos intersexo.[131]
A edición do xenoma in vivo usando CRISPR-Cas9 realizouse con éxito en numerosos organismos modelo, incluíndo Escherichia coli,[132] Saccharomyces cerevisiae,[133] [134]Candida albicans, Methanosarcina acetivorans,[135][136] Caenorhabditis elegans,[137] Arabidopsis spp.,[138] Danio rerio,[139] e Mus musculus.[140][141] Outros éxitos conseguíronse no estudo da bioloxía básica, na creación de modelos de enfermidades,[137][142] e no tratamento experimental de modelos de enfermidades.[143]
Un problema é que os efectos da edición do xenoma fóra de diana (edición de xenes ao lado dos que realmente se pretendía) pode confundir os resultados dun experimento de edición de xenes por CRISPR, é dicir, o cambio fenotípico observado pode non deberse á modificación do xene diana, senón á dalgún outro xene. Fixéronse modificacións ao CRISPR para minimizar a posibilidade de efectos fóra de diana. Adoitan recomendarse experimentos de CRISPR ortogonal para confirmar os resultados dun experimento de edición de xenes.[144][145]
CRISPR simplifica a creación de organismos modificados xeneticamente para a investigación que imitan unha enfermidade ou mostran o que ocorre cando un xene é sometido a knock-down ou mutado. A CRISPR pode utilizarse na liña xerminal para crear organismos nos cales o xene diana queda cambiado en todas partes onde se encontre (é dicir, en todas as células/tecidos/órganos dun organismo multicelular), ou pode utilizarse en células non xerminais para crear cambios locais que só afectan a certas poboacións celulares do organismo.[146][147][148]
CRISPR pode utilizarse para crear modelos celulares humanos de enfermidades.[149] Por exemplo, cando se aplica a células nais pluripotentes humanas, CRISPR utilizouse para introducir mutacións dirixidas a diana en xenes relevantes para a enfermidade renal poliquística e a glomeruloesclerose segmentaria e focal.[150] Estas células nai pluripotentes modificadas por CRISPR fixéronse despois crecer en organoides de ril humano que mostraron fenotipos específicos de enfermidades. Os organoides de ril formados a partir de células nais con mutacións para a enfermidade renal poliquística formaron estruturas quísticas translúcidas grandes a partir dos túbulos renais. Os quistes podían acadar dimensións macroscópicas, de ata un centímetro de diámetro.[151] Os organoides de ril con mutacións nun xene ligadas á glomeruloesclerose segmentaria e focal desenvolveron defectos nas unións celulares entre os podocitos, que son as células filtradoras que están afectadas nesa doenza. Isto debíase á incapacidade dos podocitos de formar microvilosidades entre as células adxacentes.[152] Un dato importante é que estes fenotipos de enfermidades estaban ausentes nos organoides control de idéntico fondo xenético, pero que carecían de modificacións CRISPR.[150]
Seguiuse unha estratexia similar para modelizar a síndrome do QT longo en cardiomiocitos derivados de células nai pluripotentes.[153] Estes modelos celulares xerados por CRISPR, con controis isoxénicos, proporcionan un novo modo de estudar as doenzas humanas e de ensaiar fármacos.
Biomedicina[editar | editar a fonte]
A tecnoloxía CRISPR-Cas propúxose como tratamento para múltiples doenzas humanas, especialmente as que teñen unha causa xenética.[154] A súa capacidade de modificar secuencias específicas de ADN convértea nunha ferramenta coa potencial capacidade de arranxar mutacións que causan enfermidades. As primeiras investigacións sobre isto en modelos animais indican que as terapias baseadas na tecnoloxía CRISPR poderían utilizarse para tratar unha ampla gama de doenzas,[155] incluíndo o cancro,[156] a proxeria,[157] a beta-talasemia,[158][159][160] a anemia falciforme,[160][161] a hemofilia,[162] a fibrose quística,[163] a distrofia muscular de Duchenne,[164] a enfermidade de Huntington,[165][166] a amiloidose de transtiretina[41] e doenzas cardíacas.[167] A CRISPR tamén se utilizou para previr a malaria eliminando mosquitos, o cal podería eliminar o vector e a enfermidade en humanos.[168] A CRISPR pode tamén ter aplicacións na enxeñaría de tecidos e na medicina rexenerativa, como crear vasos sanguíneos humanos que carezan da expresión das proteínas MHC de clase II, que a miúdo causan o rexeitamento de transplantes.[169]
Ademais, os ensaios clínicos para curar a beta-talasemia e a anemia falciforme en pacientes humanos usando a tecnoloxía CRISPR-Cas9 tiveron resultados prometedores.[170][171]
Non obstante, seguen existindo algunhas limitacións no uso desta tecnoloxía en terapia xénica, como son: a frecuencia relativamente alta de efectos fóra de diana, a necesidade dunha secuencia PAM preto do sitio diana, a apoptose mediada por p53 por roturas de dobe febra inducidas por CRISPR e a toxicidade inmunoxénica debida ao sistema de entrega que normalmente é un virus.[172]
Cegueira[editar | editar a fonte]
O tratamento CRISPR para a LCA10 (a variante máis común da amaurose conxénita de Leber, que é a principal causa de cegueira herdada na nenez) modifica o xene de fotorreceptor defectuoso do paciente.
En marzo de 2020 nun estudo sobre esta doenza nos Estados Unidos, patrocinado por Editas Medicine, un primeiro paciente voluntario recibiu unha dose do tratamento para testar a súa seguiridade. En xuño de 2021 empezou o recrutamento de participantes para unha dose adulta alta e unha cohorte pediátrica con 4 pacientes voluntarios en cada unha. A administración de doses ás novas cohortes esperábase que estivese completa en xullo de 2022.[173] En novembro de 2022, Editas informou que o 20% dos pacientes tratados tiveran melloramentos importantes, pero tamén anunciou que a poboación diana resultante era demasiado pequena como para soportar un desenvolvemento continuado independente.[174]
Cancro[editar | editar a fonte]
A CRISPR tamén encontrou moitas aplicacións no desenvolvemento de inmunoterapias baseadas en células.[175] O primeiro ensaio clínico con CRISPR empezou en 2016. Consistiu en extraer células inmunitarias de persoas con cancro de pulmón, usar CRISPR para editar o xene expresado PD-1, despois administrar as células alteradas de volta á mesma persoa. Outros vinte ensaios estaban en marcha ou case listos, principalmente na China en 2017.[156]
En 2016 a FDA dos Estados Unidos aprobou un ensaio clínico con CRISPR no que se extraerían células T de persoas con diferentes tipos de cancro e despois se lles administrarían ás mesmas persoas esas células unha vez modificadas por enxeñaría.[176]
En novembro de 2020 en modelos animais de rato, utilizouse CRISPR para tratar o glioblastoma (tumor cerebral de rápido crecemento) e o cancro de ovario metastático, xa que eses son dous cancros con peor prognóstico e diagnostícanse normalmente en estadios avanzados. Os tratamentos orixinaron unha inhibición do crecemento do tumor, un incremento da supervivencia do 80% no caso do cancro ovárico metastático e a apoptose da célula tumoral, e no caso do glioblastoma unha inhibición do crecemento do tumor dun 50% e unha mellora da supervivencia do 30%.[177]
En outubro de 2021 CRISPR Therapeutics anunciou os resultados do seu ensaio en fase 1 en marcha nos Estados Unidos para unha terapia de célula T aloxénica. Estas células tomáronse de doantes sans e foron editadas para atacar células cancerosas e evitar ser vistas como unha ameaza polo sistema inmunitario do receptor, e despois multiplicáronse en enormes lotes que podían administrarse a un gran número de pacientes.[173]
En decembro de 2022 unha rapaza británica de 13 anos que fora diagnosticada de leucemia linfoblástica aguda de células T incurable foi curada no Great Ormond Street Hospital, no primeiro uso documentado de edición de xenes terapéutica para este propósito, despois de someterse a seis meses de tratamento experimental, cando outros tipos de tratamento fallaran previamente. O procedemento incluía reprogramar células T sas para destruír células T cancerosas para en primeiro lugar eliminar a leucemia, e despois reconstruír o sistema inmunitario da paciente desde cero usando células inmunitarias sas.[178] O equipo utilizou edición BASE e tratara previamente un caso de leucemia linfoblástica aguda en 2015 usando TALENs.[179]
Diabetes[editar | editar a fonte]
A diabetes tipo 1 é un trastorno endócrino que é o resultado da perda de células beta pancreáticas que producen insulina, unha hormona vital para que o azucre do sangue entre na células para producir enerxía. Os investigadores estiveron intentando o transplante de células beta sas. Utilizouse CRISPR para editar as células para reducir as posibilidades de que o corpo do paciente rexeite o transplante. En febreiro de 2022 realizouse un ensaio en fase 1 na cal recibiu o tratamento un paciente voluntario.[173][180]
VIH/SIDA[editar | editar a fonte]
O virus da inmunodeficiencia humana (VIH) é un virus que ataca o sistema inmunitario do corpo. Aínda que existen tratamentos efectivos para manter a raia a enfermidade e que permiten aos pacientes ter unha vida sa, o VIH é un retrovirus, o que significa que integra unha versión inactiva del no xenoma humano, que se pode activar en certos momentos. A CRISPR pode utilizarse para eliminar selectivamente o virus do xenoma deseñando un ARN guía que teña como diana o xenoma integrado do VIH. Un probelma con esta estratexia é que require a eliminación do xenoma integrado do VIH de case todas as células, o cal, sendo realista, pode ser difícil de conseguir.[173]
Infección[editar | editar a fonte]
As "nucleases guiadas por ARN" baseadas en CRISPR-Cas poden utilizarse para alterar factores de virulencia, xenes que codifican resistencia a antibióticos e outras secuencias medicamenrte relevantes de interese. Esta tecnoloxía representa unha nova forma de terapia antimicrobiana e unha estratexia para manipular poboacións bacterianas.[181][182] Estudos recentes indican unha correlación entre a interferencia do locus CRISPR-Cas e a adquisición e resistencia a antibióticos.[183] Este sistema proporciona protección á bacteria ante ADN alleo invasor, como o de transposóns, bacteriófagos e plásmidos. Este sistema exerce unha forte presión de selección para a adquisición de resistencia a antibióticos e factores de virulancia en patóxenos bacterianos.[183]
As terapias baseadas na tecnoloxía de edición de xenes por CRISPR–Cas3 entregadas por bacteriófagos preparados por enxeñaría poderían usarse para destruír o ADN diana en patóxenos.[184] Cas3 é máis destrutuiva que a máis coñecida Cas9.[185][186]
Diversas investigacións suxiren que CRISPR é un modo efectivo de limitar a replicación de múltiples herpesvirus. Conseguiu erradicar o ADN viral no caso do virus de Epstein–Barr (EBV). O uso de CRISPRs antiherpesvirus ten prometedoras aplicacións, como eliminar o EBV causante de cancro das células tumorais, axudando a eliminar os invasores víricos en órganos de doantes para pacientes inmunocomprometidos, ou impedir brotes de Herpes labialis e infeccións recorrentes no ollo ao bloquear a reactivación do HSV-1. En agosto de 2016 estes procedementos estaban esperando a ser testados.[187]
Os resultados iniciais no tratamento e cura do VIH foron bastante positivos, en outubro de 2021 9 dun total de 23 ratos humanizados tratados cunha combinación de anti-retrovirais e CRISPR/Cas-9 tiñan o virus indetectable, mesmo despois do usual período de rebote. Ningún dos dous tratamentos por si só tiña ese efecto.[188] Os ensaios clínicos en humanos empezaron en 2022.[189][190]
A CRISPR pode facer revivir o concepto de xenotransplante de órganos animais a persoas. Os retrovirus presentes en xenomas animais podía causar dano aos receptores do transplante. En 2015 un equipo eliminou 62 copias dunha determinada secuencia de ADN retroviral do xenoma do porco nunha célula epitelial de ril.[191] Demostrouse recentemente por primeira vez a capacidade de que nazan vivos espécimes de porcos despois de eliminar eses retrovirus dos xenomas dos embrións usando CRISPR.[192]
Antropoloxía xenética[editar | editar a fonte]
A CRISPR-Cas9 pode utilizarse para investigar e identificar as diferenzas xenéticas entre humanos e outros primates, como os simios, especialmente no cerebro. Por exemplo, ao reintroducir variantes de xenes arcaicos en organoides de cerebro para mostrar unha influencia na neuroxénese,[193] a lonxitude da metafase de proxenitores apicais do neocórtex en desenvolvemento,[194] ou polo knockout dun xene en células nais embrionarias para identificar un regulador xenético que por transicións da forma da célula temperás conduciron á expansión evolutiva do prosencéfalo humano.[195][196] Un estudo describiu unha influencia importante dunha variante dun xene arcaico no neurodesenvolvemento[197][198] que pode ser un artefacto dun efecto colateral do CRISPR,[199][200] xa que non puido ser replicado nun estudo posterior.[201]
Por técnica[editar | editar a fonte]
Knockdown/activación[editar | editar a fonte]
- Artigo principal: Interferencia por CRISPR.
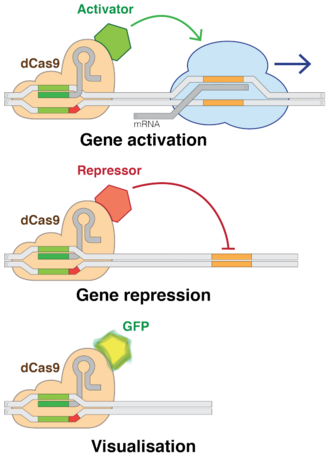
O uso de versións "mortas" de Cas9 (dCas9) elimina a capacidade de cortar o ADN de CRISPR, mentres que conserva esa capacidade para actuar sobre as secuencias desexadas. Múltiples grupos de investigadores engadiron varios factores reguladores a dCas9s, permitíndolles activar ou desactivar case calquera xene ou axustar o seu nivel de actividade.[191] Igual que a interferencia de ARN (RNAi), a interferencia por CRISPR (CRISPRi) desactiva xenes de forma reversible ao unirse á diana pero non cortar o sitio. O sitio diana é metilado, modificando así epixeneticamente o xene. Esta modificación inhibe a transcrición. Estas modificacións situadas con precisión poden usarse despois para regular os efectos na expresión xénica da dinámica do ADN despois da inhibición de certas secuencias xenómicas do ADN. Nos últimos anos, investigáronse intensamente as marcas epixenéticas en diferentes células humanas e atoparon que certos padróns nas marcas se correlacionaban con todas as cousas desde o crecemento do tumor á actividade cerebral.[10] Inversamente, a activación mediada por CRISPR (CRISPRa) promove a transcrición xénica.[202] Cas9 é unha forma efectiva de actuar sobre unha diana e silenciar xenes específicos a nivel do ADN.[203] En bacterias, a presenza de Cas9 soa é dabondo para bloquear a transcrición. Para aplicacións en mamíferos, engádese unha sección da proteína. O seu ARN guía únese a secuencias do ADN regulatorias chamadas promotores que preceden inmediatamente ao xene diana.[204]
Cas9 utilizouse para transportar factores de transcrición sintéticos que activaban xenes humanos específicos. A técnica conseguiu un forte efecto ao dirixir múltiples construtos CRISPR a localizacións lixeiramente diferentes do promotor do xene.[204]
Edición de ARN[editar | editar a fonte]
En 2016 os investigadores demostraron que o CRISPR procedente dunha bacteria común da boca podería usarse para editar o ARN. Os investigadores buscan en bases de datos que conteñen centos de millóns de secuencias xenéticas aquelas que lembran os xenes CRISPR. Estudaron a fusobacteria Leptotrichia shahii, a cal ten un grupo de xenes que lembran os xenes CRISPR, pero con importantes diferenzas. Cando os investigadores equiparon outras bacterias con eses xenes, aos cales chamaron C2c2, atoparon que os organismos adquirían unha nova defensa.[205] O C2c2 foi despois renomeado como Cas13a para axustarse á nomenclatura estándar dos xenes Cas.[206]
Moitos virus codifican a súa información xenética no ARN en vez de no ADN, que eles reprograman para facer novos virus. O VIH e o poliovirus son virus dese tipo. As bacterias con Cas13 producen moléculas que poden desfacer o ARN, destruíndo o virus. Preparar convenientemente estes xenes serve para editar calquera molécula de ARN.[205]
Os sistemas CRISPR-Cas poden tamén epregarse para editar xenes de microARN e ARN non codificante longo en plantas.[207]
Aplicacións terapéuticas[editar | editar a fonte]
As edicións dirixidas para corrixir secuencias mutadas foi proposta por primeira vez e demostrada en 1995.[208] Este traballo inicial usou oligonucleótidos de ARN antisentido sintético complementario dunha mutación de codón de stop premaduro nunha secuencia de distrofina para activar a edición de adenina (A) a inosina (I) do codón de stop xerando un codón que se pode ler nun sistema modelo de células de Xenopus.[208] Aínda que isto tamén causou transicións inadvertidas de A a I en lugares próximos, as transicións de A a I (lidas como G) poden corrixir os tres posibles codóns de stop, pero non poden crear un novo codón de stop. Por tanto, os cambios causaron >25 % de corrección do codón de stop diana con lectura dunha secuencia augas abaixo de luciferase reporteira. Posteriores traballos feitos por Rosenthal conseguiron editar unha secuencia de ARNm mutada en cultivos de células de mamíferos ao dirixir un oligonucleótido ligado á citidina desaminase para corrixir unha secuencia mutada de fibrose quística.[209] Máis recentemente, empregouse CRISPR-Cas13 fusionada a desaminases para dirixir a edición de ARNm.[210]
En 2022 informouse do uso de Cas7-11, máis apropiada para a edición de ARN terapéutica que Cas13.[211][212] Permite cortes suficientemente ben dirixidos a diana e unha verisón inicial xa foi utilizada para a edición in vitro en 2021.[213]
Comparación coa edición do ADN[editar | editar a fonte]
A diferenza da edición de ADN, que é permanente, os efectos da edición do ARN, incluíndo as posibles mutacións fóra de diana no ARN, son transitorias e non se herdan. A edición do ARN é, por tanto, considerada menos perigosa. Ademais, pode soamente necesitar un ARN guía ao usar a proteína ADAR xa atopada en células humanas e de moitos outros eucariotas en vez de ter que introducir unha proteína allea no corpo.[214]
Xenética dirixida[editar | editar a fonte]
A xenética dirixida (gene drive) pode proporcionar unha potente feramenta para restablecer o equilibrio dos ecosistemas ao eliminar especies invasoras. Hai dúbidas sobre a súa eficacia, e sinaláronse posibles consecuencias inesperadas nas especies diana así como nas especies non diana, especialmente pola posibilidade dunha liberación accidental dos laboratorios á natureza. Propuxéronse varias salvagardas para asegurar a contención da xenética dirixida experimental de tipo molecular, reprodutivo e ecolóxico.[215] Moitos recomendan que a inmunización e a xenética dirixida inversa sexan desenvolvidos en tándem coa xenética dirixida para sobreescribir os seus efectos se for necesario.[216] Hai consenso en que os efectos a longo prazo deben ser estudados máis detalladamente ante a posibilidade de alteracións ecolóxicas que non poidan ser corrixidas con xenética dirixida inversa.[217]
Empobrecemento xenético in vitro[editar | editar a fonte]
As bibliotecas de secuenciación empobrecidas a miúdo teñen abundantes secuencias non desexadas. Cas9 pode empobrecer especificamente as secuencias non desexadas con roturas de dobre febra con ata un 99 % de eficiencia e sen efectos fóra de diana significativos, como se viu con encimas de restrición. O tratamento con Cas9 pode empobrecer o ARNr abundante, mentres que incrementa a sensibilidade do patóxeno en bibliotecas de RNA-seq.[218]
Edición epixenómica[editar | editar a fonte]
A edición epixenómica ou enxeñaría epixenómica é un tipo de enxeñaría xenética na cal se modifica o epixenoma en sitios específicos usando moléculas preparadas por enxeñaría dirixidas a ditos sitios (en oposición ás modificacións en todo o xenoma). Mentres que a edición de xenes implica cambiar a propia secuencia de ADN, a edición epixenética só supón a modificación e presentación de secuencias de ADN a proteínas e outros factores que se unen ao ADN que inflúen na función do ADN. Ao "editar” características epixenómicas deste xeito, pódese determinar o papel biolóxico exacto dunha modificación epixenética no sitio en cuestión.
As proteínas preparadas por enxeñaría usadas para a edición epixenómica están compostas dun dominio de unión ao ADN que ten como diana secuencias específicas e un dominio efector que modifica características epixenómicas. Actualmente, están utilizándose predominantemente tres grandes grupos de proteínas que se unen ao ADN para a edición epixenómica, que son: proteínas de dedo de cinc, efectores similares a activadores de transcrición (TALEs) e fusións de Cas9 deficientes en nuclease (CRISPR).
Outras aplicacións[editar | editar a fonte]
A regulación dirixida de xenes relacionados con enfermidades podería permitir novas terapias para moitas doenzas, especialmente en casos nos que as terapias xénicas adecuadas non están aínda desenvolvidas ou son inapropiadas.[219] Aínda que as consecuencias a nivel transxeneracional e de poboación non se comprenden totalmente, pode converterse nunha importante ferramenta para aplicala á xenómica funcional e á medicina personalizada.[220] Igual que na edición de ARN, non implica cambios xenéticos e os riscos que os acompañan.[219] Un exemplo dun posible uso funcional da edición do epixenoma describiuse en 2021: a represión da expresión xénica de Nav1.7 por CRISPR-dCas9, que mostrou un potencial terapéutico en tres modelos de ratos de dor crónica.[221][222]
En 2022 fixeronse investigacións que avaliaron a súa utilidade na redución dos niveis de proteína tau, regulando unha proteína que intervén na enfermidade de Huntington, para tratar unha forma herdada de obesidade, e a síndrome de Dravet.[223]
Integrases dirixidas por CRISPR[editar | editar a fonte]
A combinación de CRISPR-Cas9 con integrases orixinou unha técnica para facer grandes edicións sen as problemáticas roturas de dobre febra, como se demostrou con PASTE[224] en 2022. Informouse que se podía usar para entregar xenes de ata 36.000 pares de bases de ADN a varios tipos de células humanas e podería usarse para tratar doenzas causadas por un gran número de mutacións.[225][226]
Prime editing[editar | editar a fonte]
A edición de calidade (prime editing en inglés)[227] ou edición de bases é un refinamento da CRISPR para poder inserir ou eliminar con exactitude seccións do ADN. As edicións de CRISPR non son sempre perfectas e os cortes poden acabar aparecendo no sitio incorrecto. Ambas cuestións son un problema á hora de usar esta tecnoloxía en medicina.[228] Esta edición de calidade non corta o ADN bicatenario senón que utiliza o aparato de busca de diana da CRISPR para transportar un encima adicional á secuencia desexada, onde este converte un só nucleótido noutro con precisión.[229] A nova molécula guía, chamada ARN guía de prime editing (pegRNA en inglés), contén un molde de ARN para que se engada unha nova secuencia de ADN ao xenoma na localización onde está a diana. Para isto cómpre unha segunda proteína, unida a Cas9: un encima transcriptase inversa, que pode facer unha nova febra de ADN a partir do molde de ARN e inserila no sitio onde se fixo a amosega.[230] Eses tres eventos emparellados independentemente proporcionan cada un unha oportunidade para impedir eventos fóra de secuencia, o cal incrementa significativamente a flexibilidade á hora de dirixirse á diana e a precisión da edición.[229] A prime editing foi desenvolvida por investigadores do Broad Institute da MIT e Harvard en Massachusetts.[231] Serán necesarios máis traballos para optimizar os métodos.[231][230]
Sociedade e cultura[editar | editar a fonte]
Modificación da liña xerminal humana[editar | editar a fonte]
En marzo de 2015 múltiples grupos de investigadores anunciaran que estaban realizando investigacións coa intención de establecer os fundamentos para aplicar a CRISPR a embrións humanos para a enxeñaría da liña xerminal humana, incluíndo laboratorios dos Estados Unidos, a China e o Reino Unido, así como a compañía biotecnolóxica estadounidense OvaScience.[232] Os científicos, entre eles o codescubridor do CRISPR, instaron unha moratoria mundial na aplicación do CRISPR á liña xerminal humana, especialmente para o uso clínico. Dixeron que "os científicos deberían evitar incluso o intento, en xurisdicións permisivas, da modificación do xenoma na liña xerminal para a aplicación clínica en humanos" ata que todas as implicacións "se discutan entre os científicos e as organizacións gobernamentais".[233][234] Estes científicos apoiaron unha maior investigación a baixo nivel sobre CRISPR e non consideralo como un método suficientemente desenvolvido de uso clínico para facer cambios herdables en seres humanos.[235]
En abril de 2015 científicos chineses informaron dos resultados dun intento de alterar o ADN de embrións humanos non viables usando CRISPR para corrixir unha mutación que causa a beta-talasemia, un trastorno herdable que pode ser mortal.[236][237] O estudo fora previamente rexeitado polas revistas Nature e Science en parte debido a consideracións éticas.[238] Os experimentos tiveron como resultado o cambio con éxito de só algúns dos xenes pretendidos, e presentaban efectos fóra de diana sobre outros xenes. Os investigadores afirmaron que o CRISPR non está listo para a aplicación clínica en medicina reprodutiva.[238] En abril de 2016 informouse que científicos chineses fixeran un segundo intento fracasado de alterar o ADN de embrións humanos non viables usando CRISPR; esta vez para alterar o xene CCR5 para facer que o embrión fose resistente á infección por VIH.[239]
En decembro de 2015 un Cumio Internacional sobre a Edición de Xenes Humanos tivo lugar en Washington baixo a dirección de David Baltimore. Membros das academias científicas nacionais dos Estados Unidos, o Reino Unido e a China discutiron os aspectos éticos da modificación da liña xerminal. Estiveron de acordo en apoiar a investigación básica e clínica baixo certas directrices legais e éticas. Fíxose unha distinción específica entre as células somáticas, nas que os efectos das edicións están limitadas a un só individuo, e as células da liña xerminal, nas que os cambios no xenoma poden herdarse polos descendentes. As modificacións herdables poderían ter consecuencias non pretendidas e de longo alcance para a evolución humana, tanto xeneticamente (por exemplo, as interaccións xene–ambiente) coma culturalmente (por exemplo, o darwinismo social). A alteración dos gametocitos e embrións para xerar cambios herdables en humanos definiuse como irresponsable. O grupo concordou en iniciar un foro internacional para tratar eses asuntos e harmonizar as regulacións entre os países.[240]
En febreiro de 2017 o Comité das Academias Nacionais de Ciencias, Enxeñaría e Medicina dos Estados Unidos (NASEM) sobre a Edición de Xenes Humanos publicou un informe revisando as cuestións éticas, legais e científicas da tecnoloxía de enxeñaria xenómica. A conclusión do informe afirmaba que a edición xenómica herdable non é permisible agora, pero podería ser xustificable para certas condicións médicas, pero non xustificaban o uso de CRISPR para a mellora das características humanas.[241]
En novembro de 2018 Jiankui He na China anunciou que editara dous embrións humanos para intentar desactivar o xene da CCR5, que codifica un receptor que usa o VIH para entrar nas células. Dixo que unhas poucas semanas antes naceran dúas nenas xemelgas tratadas para facelas invulnerables ao VIH, chamadas Lulu e Nana, pero que as nenas aínda portaban copias funcionais do CCR5 xunto con CCR5 desactivado (un caso de mosaicismo) e eran aínda vulnerables ao VIH. O traballo foi amplamente condenado como falto de ética, perigoso e prematuro.[242] Un grupo internacional de científicos solicitou unha moratoria mundial sobre a edición xenética de embrións humanos.[243]
Políticas regulatorias sobre enxeñaría xenética[editar | editar a fonte]
Os principios regulatorios para o sistema CRISPR-Cas9 varían arredor do mundo. En febreiro de 2016 científicos británicos recibiron permiso dos reguladores para modificar xeneticamente embrións humanos usando CRISPR-Cas9 e técnicas relacionadas. Porén, aos investigadores prohibíuselles implantar os embrións e ditos embrións foron destruídos sete días despois.[244]
Os Estados Unidos teñen un elaborarado sistema regulatorio interdepartmental para avaliar novos alimentos modificados xeneticamente. Por exemplo, a Lei de Protección de Riscos para a Agricultura de 2000 dá ao Departamento de Agricultura dos Estados Unidos a autoridade de supervisar a detección, control, erradicación, supresión, prevención ou demora do espallamento de pragas agrícolas ou malas herbas prexudiciais para protexer a agricultura, o medio ambiente e a economía dos Estados Unidos. A lei regula calquera organismo modificado xeneticamente que utilice o xenoma dunha "praga agrícola" predefinida ou calquera planta non previamente categorizada.[245] En 2015 Yinong Yang conseguiu desactivar 16 xenes específicos no fungo Agaricus bisporus (champiñón común) para facer que non se volvese marrón. Como non engadiran ningún ADN de especie allea (transxénico) a este organismo, o fungo non podía ser regulado polo USDA baixo a Section 340.2.[246] O champiñón de Yang foi o primeiro organismo modificado xeneticamente co sistema de proteínas de CRISPR-Cas9 que pasou a regulación dos Estados Unidos.[247]
En 2016 o USDA patrocinou un comité para considerar futuras políticas regulatorias para as técnicas de modificación xenética por vir. Coa axuda das Academias Nacionais de Ciencias, Enxeñaria e Medicina dos Estados Unidos, os grupos con interese especial xuntáronse o 15 de abril para considerar os posibles avances en enxeñaría xenética nos próximos cinco anos e calquera nova regulación que puidese necesitarse como resultado.[248] En 2017 a FDA propuxo unha regra pola que clasificarían as modificacións por enxeñaría xenética en animais como "fármacos animais", somenténdoos a estritas regulacións se se poñen á venda e reducindo a capacidade de individuos e pequenos negocios de sacarlles rendemento.[249][250]
Na China, onde as condicións sociais son moi diferentes das de Occidente, as enfermidades xenéticas teñen un forte estigma.[251] Isto levou á China a ter poucas barreiras regulatorias para o uso desta tecnoloxía.[252][253]
Recoñecementos[editar | editar a fonte]
En 2012 e 2013, a tecnoloxía CRISPR foi un dos finalistas no premio ao descubrimento do ano de Science Magazine. En 2015 foi a vencedora do premio.[191] CRISPR foi considerado un dos 10 descubrimentos tecnolóxicos que elixe a revista MIT Technology Review en 2014 e 2016.[254][255] En 2016 Jennifer Doudna e Emmanuelle Charpentier, xunto con Rudolph Barrangou, Philippe Horvath e Feng Zhang gañaron o premio Gairdner International. En 2017 Doudna e Charpentier recibiron o Premio Xapón en Toquio pola súa invención revolucionaria do CRISPR-Cas9. En 2016, Charpentier, Doudna e Zhang gañaron o premio Tang de ciencias biofarmacéuticas.[256] En 2020 Charpentier e Doudna foron galardoadas co premio Nobel de Química, o primeiro que recibiu un equipo integramente feminino, "polo desenvolvemento dun método para a edición do xenoma."[257]
Notas[editar | editar a fonte]
- ↑ 1,0 1,1 1,2 Bak RO, Gomez-Ospina N, Porteus MH (agosto de 2018). "Gene Editing on Center Stage". Trends in Genetics 34 (8): 600–611. PMID 29908711. doi:10.1016/j.tig.2018.05.004.
- ↑ "The Nobel Prize in Chemistry 2020". The Nobel Prize. Consultado o 2020-12-10.
- ↑ Cohen J (7 de outubro de 2020). "CRISPR, the revolutionary genetic "scissors," honored by Chemistry Nobel". Science. doi:10.1126/science.abf0540.
- ↑ Cohen J (2018-06-04). "With prestigious prize, an overshadowed CRISPR researcher wins the spotlight". Science | AAAS (en inglés). Consultado o 2020-05-02.
- ↑ 5,0 5,1 Owens R (8 de outubro de 2020). "Nobel prize: who gets left out?". The Conversation. Consultado o 13 de decembro de 2021.
- ↑ "Lithuanian scientists not awarded Nobel prize despite discovering same technology.". LRT.LT. 8 de outubro de 2020.
- ↑ Šikšnys V (2018-06-16). "Imam genų žirkles, iškerpam klaidą, ligos nelieka". Laisvės TV / Freedom TV. <...>Tai mes tą savo straipsnį išsiuntėm į redakciją pirmieji, bet laimės ten daug nebuvo. Viena redakcija pasakė, kad mes net recenzentam nesiųsim. Nusiuntėm į kitą redakciją – tai jis (straipsnis) pragulėjo kažkur ant redaktoriaus stalo labai ilgai. Na ir taip galų gale išsiuntėm į trečią žurnalą ir trečias žurnalas po kelių mėnesių jį išspausdino. Bet, aišku, Berklio universiteto mokslininkams sekėsi geriau – jie išsiuntė straipsnį į žurnalą Science – jį priėmė ir išspausdino per 2 savaites. Nors iš tikro jie tą straispnį išsiuntė pora mėnesių vėliau nei mes. 12:22 No minuto. LaisvėsTV. Consultado o 2018-06-30.
<...> Ben, nós fomos os que enviamos o artigo primeiro, mais non tivemos moita sorte.
- ↑ Zhang JH, Pandey M, Kahler JF, Loshakov A, Harris B, Dagur PK, et al. (novembro de 2014). "Improving the specificity and efficacy of CRISPR/CAS9 and gRNA through target specific DNA reporter". Journal of Biotechnology 189: 1–8. PMC 4252756. PMID 25193712. doi:10.1016/j.jbiotec.2014.08.033.
- ↑ Vakulskas CA, Dever DP, Rettig GR, Turk R, Jacobi AM, Collingwood MA, et al. (agosto de 2018). "A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells". Nature Medicine 24 (8): 1216–1224. PMC 6107069. PMID 30082871. doi:10.1038/s41591-018-0137-0.
- ↑ 10,0 10,1 10,2 Ledford H (marzo de 2016). "CRISPR: gene editing is just the beginning". Nature 531 (7593): 156–159. Bibcode:2016Natur.531..156L. PMID 26961639. doi:10.1038/531156a.
- ↑ Travis J (17 de decembro de 2015). "Breakthrough of the Year: CRISPR makes the cut". Science Magazine. American Association for the Advancement of Science.
- ↑ Ledford H (xuño de 2015). "CRISPR, the disruptor". Nature 522 (7554): 20–24. Bibcode:2015Natur.522...20L. PMID 26040877. doi:10.1038/522020a.
- ↑ Young S (11 de febreiro de 2014). "CRISPR and Other Genome Editing Tools Boost Medical Research and Gene Therapy's Reach". MIT Technology Review. Arquivado dende o orixinal o 12 de xaneiro de 2016. Consultado o 2014-04-13.
- ↑ 14,0 14,1 Heidenreich M, Zhang F (xaneiro de 2016). "Applications of CRISPR-Cas systems in neuroscience". Nature Reviews. Neuroscience 17 (1): 36–44. PMC 4899966. PMID 26656253. doi:10.1038/nrn.2015.2.
- ↑ Barrangou R, Doudna JA (setembro de 2016). "Applications of CRISPR technologies in research and beyond". Nature Biotechnology 34 (9): 933–941. PMID 27606440. doi:10.1038/nbt.3659.
- ↑ Cox DB, Platt RJ, Zhang F (febreiro de 2015). "Therapeutic genome editing: prospects and challenges". Nature Medicine 21 (2): 121–131. PMC 4492683. PMID 25654603. doi:10.1038/nm.3793.
- ↑ Pollack A (11 de maio de 2015). "Jennifer Doudna, a Pioneer Who Helped Simplify Genome Editing". New York Times. Consultado o 8 de outubro de 2020.
- ↑ Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E (agosto de 2012). "A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity". Science 337 (6096): 816–821. Bibcode:2012Sci...337..816J. PMC 6286148. PMID 22745249. doi:10.1126/science.1225829.
- ↑ "CRISPR Madness". GEN. 2013-11-08.
- ↑ Staff (1 April 2015). "News: Products & Services". Genetic Engineering & Biotechnology News (Paper) 35 (7): 8. doi:10.1089/gen.35.21.05.
- ↑ "Who Owns the Biggest Biotech Discovery of the Century? There's a bitter fight over the patents for CRISPR, a breakthrough new form of DNA editing". MIT Technology Review. Consultado o 25 de febreiro de 2015.
- ↑ Fye S. "Genetic Rough Draft: Editas and CRISPR". The Atlas Business Journal. Consultado o 19 de xaneiro de 2016.
- ↑ "CRISPR-Cas systems and methods for altering expression of gene products". Google Patents.
- ↑ Shaffer C (abril de 2022). "Broad defeats Berkeley CRISPR patent". Nature Biotechnology 40 (4): 445. PMID 35288688. doi:10.1038/d41587-022-00004-2.
- ↑ "CRISPR patents to go on trial". Nature Biotechnology 34 (2): 121. febreiro de 2016. PMID 26849500. doi:10.1038/nbt0216-121a.
- ↑ Pollack A (15 de febreiro de 2017). "Harvard and M.I.T. Scientists Win Gene-Editing Patent Fight". The New York Times.
- ↑ Akst J (15 de febreiro de 2017). "Broad Wins CRISPR Patent Interference Case". The Scientist Magazine.
- ↑ Noonan KE (16 de febreiro de 2017). "PTAB Decides CRISPR Interference in Favor of Broad Institute – Their Reasoning". Patent Docs.
- ↑ Potenza A (13 de abril de 2017). "UC Berkeley challenges decision that CRISPR patents belong to Broad Institute". The Verge. Consultado o 22 de setembro de 2017.
- ↑ Buhr S (26 de xullo de 2017). "The CRISPR patent battle is back on as UC Berkeley files an appeal". TechCrunch. Consultado o 22 de setembro de 2017.
- ↑ Westman N (1 de marzo de 2022). "UC Berkeley loses CRISPR patent case". The Verge. Consultado o 6 de marzo de 2022.
- ↑ 32,0 32,1 Philippidis A (7 de agosto de 2017). "MilliporeSigma to Be Granted European Patent for CRISPR Technology". Genetic Engineering & Biotechology News. Consultado o 22 de setembro de 2017.
- ↑ Akst J (24 de marzo de 2017). "UC Berkeley Receives CRISPR Patent in Europe". The Scientist. Consultado o 22 de setembro de 2017.
- ↑ Cohen J (4 de agosto de 2017). "CRISPR patent battle in Europe takes a 'wild' twist with surprising player". Science. doi:10.1126/science.aan7211.
- ↑ "Top EU court: GMO rules cover plant gene editing technique". Retuers. 25 de xullo de 2018.
- ↑ AFP (7 de febreiro de 2020). "US Trial Shows 3 Cancer Patients Had Their Genomes Altered Safely by CRISPR". ScienceAlert (en inglés). Consultado o 2020-02-09.
- ↑ Chamary JV. "These Scientists Deserved A Nobel Prize, But Didn't Discover Crispr". Forbes (en inglés). Consultado o 2020-07-10.
- ↑ Fischman J. "Nobel Prize in Chemistry Goes to Discovery of 'Genetic Scissors' Called CRISPR/Cas9". Scientific American (en inglés). Consultado o 2021-03-24.
- ↑ "Two women share chemistry Nobel in historic win for 'genetic scissors'". BBC News (en inglés). 2020-10-07. Consultado o 2020-12-06.
- ↑ Kaiser J (26 de xuño de 2021). "CRISPR injected into the blood treats a genetic disease for first time". Science | AAAS (en inglés). Consultado o 11 July 2021.
- ↑ 41,0 41,1 Gillmore JD, Gane E, Taubel J, Kao J, Fontana M, Maitland ML, et al. (agosto de 2021). "CRISPR-Cas9 In Vivo Gene Editing for Transthyretin Amyloidosis". The New England Journal of Medicine 385 (6): 493–502. PMID 34215024. doi:10.1056/NEJMoa2107454.
- ↑ Boonstra E, de Kleijn R, Colzato LS, Alkemade A, Forstmann BU, Nieuwenhuis S (6 de outubro de 2015). "Neurotransmitters as food supplements: the effects of GABA on brain and behavior". Frontiers in Psychology 6: 1520. PMC 4594160. PMID 26500584. doi:10.3389/fpsyg.2015.01520.
- ↑ "Tomato In Japan Is First CRISPR-Edited Food In The World To Go On Sale". IFLScience (en inglés). Consultado o 18 de outubro de 2021.
- ↑ Wang T, Zhang H, Zhu H (15 de xuño de 2019). "CRISPR technology is revolutionizing the improvement of tomato and other fruit crops". Horticulture Research 6 (1): 77. PMC 6570646. PMID 31240102. doi:10.1038/s41438-019-0159-x.
- ↑ "Japan embraces CRISPR-edited fish". Nature Biotechnology 40 (1): 10. xaneiro de 2022. PMID 34969964. doi:10.1038/s41587-021-01197-8.
- ↑ "Startup hopes genome-edited pufferfish will be a hit in 2022". The Japan Times. 5 de xaneiro de 2022. Arquivado dende o orixinal o 17 de xaneiro de 2022. Consultado o 17 de xaneiro de 2022.
- ↑ "Gene-edited sea bream set for sale in Japan". thefishsite.com (en inglés).
- ↑ Götz L, Svanidze M, Tissier A, Brand A (xaneiro de 2022). "Consumers' Willingness to Buy CRISPR Gene-Edited Tomatoes: Evidence from a Choice Experiment Case Study in Germany". Sustainability 14 (2): 971. doi:10.3390/su14020971.
- ↑ "Are Consumers Willing to Buy CRISPR Tomatoes?". Crop Biotech Update (en inglés). Consultado o 2022-02-21.
- ↑ Whitford E (2021-05-28). "UC Berkeley Will Auction NFTs for 2 Nobel Prize Patents". Inside Higher Ed. Consultado o 2023-02-21.
- ↑ Sestino A, Guido G, Peluso AM (2022). Non-Fungible Tokens (NFTs). Examining the Impact on Consumers and Marketing Strategies. p. 28. ISBN 978-3-031-07202-4. doi:10.1007/978-3-031-07203-1.
- ↑ Chang K (2021-05-27). "You Can Buy a Piece of a Nobel Prize-Winning Discovery". New York Times. Consultado o 2023-02-21.
- ↑ Trautman LJ (2022). "Virtual Art and Non-Fungible Tokens" (PDF). Hofstra Law Review 50 (361): 369 f. doi:10.2139/ssrn.3814087.
- ↑ Jones N (2021-06-18). "How scientists are embracing NFTs". Nature 594 (7864): 482. Bibcode:2021Natur.594..481J. PMID 34145410. doi:10.1038/d41586-021-01642-3.
- ↑ 55,0 55,1 55,2 55,3 55,4 Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F (novembro de 2013). "Genome engineering using the CRISPR-Cas9 system". Nature Protocols 8 (11): 2281–2308. PMC 3969860. PMID 24157548. doi:10.1038/nprot.2013.143. hdl:1721.1/102943.
- ↑ Ly J (2013). Discovering Genes Responsible for Kidney Diseases (Ph.D.). University of Toronto. Consultado o 26 de decembro de 2016.
- ↑ Mohr SE, Hu Y, Ewen-Campen B, Housden BE, Viswanatha R, Perrimon N (setembro de 2016). "CRISPR guide RNA design for research applications". The FEBS Journal 283 (17): 3232–3238. PMC 5014588. PMID 27276584. doi:10.1111/febs.13777.
- ↑ Brazelton VA, Zarecor S, Wright DA, Wang Y, Liu J, Chen K, et al. (2015). "A quick guide to CRISPR sgRNA design tools". GM Crops & Food 6 (4): 266–276. PMC 5033207. PMID 26745836. doi:10.1080/21645698.2015.1137690.
- ↑ "Researchers establish new viable CRISPR-Cas12b system for plant genome engineering". phys.org (en inglés). Consultado o 6 de abril de 2020.
- ↑ Ming M, Ren Q, Pan C, He Y, Zhang Y, Liu S, et al. (marzo de 2020). "CRISPR-Cas12b enables efficient plant genome engineering". Nature Plants 6 (3): 202–208. PMID 32170285. doi:10.1038/s41477-020-0614-6.
- ↑ Cox DB, Gootenberg JS, Abudayyeh OO, Franklin B, Kellner MJ, Joung J, Zhang F (novembro de 2017). "RNA editing with CRISPR-Cas13". Science 358 (6366): 1019–1027. Bibcode:2017Sci...358.1019C. PMC 5793859. PMID 29070703. doi:10.1126/science.aaq0180.
- ↑ "CRISPR-Cas3 innovation holds promise for disease cures, advancing science". Cornell Chronicle (en inglés). Consultado o 24 de outubro de 2021.
- ↑ Dolan AE, Hou Z, Xiao Y, Gramelspacher MJ, Heo J, Howden SE, et al. (xuño de 2019). "Introducing a Spectrum of Long-Range Genomic Deletions in Human Embryonic Stem Cells Using Type I CRISPR-Cas". Molecular Cell (en English) 74 (5): 936–950.e5. PMC 6555677. PMID 30975459. doi:10.1016/j.molcel.2019.03.014.
- ↑ Liu Z, Dong H, Cui Y, Cong L, Zhang D (setembro de 2020). "Application of different types of CRISPR/Cas-based systems in bacteria". Microbial Cell Factories 19 (1): 172. PMC 7470686. PMID 32883277. doi:10.1186/s12934-020-01431-z.
- ↑ "Researchers develop an engineered 'mini' CRISPR genome editing system". phys.org (en inglés). Consultado o 18 de outubro de 2021.
- ↑ Xu X, Chemparathy A, Zeng L, Kempton HR, Shang S, Nakamura M, Qi LS (outubro de 2021). "Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing". Molecular Cell 81 (20): 4333–4345.e4. PMID 34480847. doi:10.1016/j.molcel.2021.08.008.
- ↑ Bravo JP, Liu MS, Hibshman GN, Dangerfield TL, Jung K, McCool RS, et al. (marzo de 2022). "Structural basis for mismatch surveillance by CRISPR-Cas9". Nature 603 (7900): 343–347. Bibcode:2022Natur.603..343B. PMC 8907077. PMID 35236982. doi:10.1038/s41586-022-04470-1.
- ↑ "Protein tweak makes CRISPR gene editing 4,000 times less error-prone". New Atlas (en inglés). 2022-03-04. Consultado o 2022-03-07.
- ↑ Kato K, Zhou W, Okazaki S, Isayama Y, Nishizawa T, Gootenberg JS, et al. (xuño de 2022). "Structure and engineering of the type III-E CRISPR-Cas7-11 effector complex". Cell 185 (13): 2324–2337.e16. PMID 35643083. doi:10.1016/j.cell.2022.05.003.
- Resumo para non profesionais: Williams S. "Neuroscientists expand CRISPR toolkit with new, compact Cas7-11 enzyme". Massachusetts Institute of Technology (en inglés). Consultado o 22 de xuño de 2022.
- ↑ "'Softer' form of CRISPR may edit genes more accurately". New Scientist. Consultado o 21 de agosto de 2022.
- ↑ Roy S, Juste SS, Sneider M, Auradkar A, Klanseck C, Li Z, et al. (xullo de 2022). "Cas9/Nickase-induced allelic conversion by homologous chromosome-templated repair in Drosophila somatic cells". Science Advances 8 (26): eabo0721. Bibcode:2022SciA....8O.721R. PMID 35776792. doi:10.1126/sciadv.abo0721.
- ↑ Horvath P, Barrangou R (xaneiro de 2010). "CRISPR/Cas, the immune system of bacteria and archaea". Science 327 (5962): 167–170. Bibcode:2010Sci...327..167H. PMID 20056882. doi:10.1126/science.1179555.
- ↑ Bialk P, Rivera-Torres N, Strouse B, Kmiec EB (2015-06-08). "Regulation of Gene Editing Activity Directed by Single-Stranded Oligonucleotides and CRISPR/Cas9 Systems". PLOS ONE 10 (6): e0129308. Bibcode:2015PLoSO..1029308B. PMC 4459703. PMID 26053390. doi:10.1371/journal.pone.0129308.
- ↑ Sander JD, Joung JK (abril de 2014). "CRISPR-Cas systems for editing, regulating and targeting genomes". Nature Biotechnology 32 (4): 347–355. PMC 4022601. PMID 24584096. doi:10.1038/nbt.2842.
- ↑ Riesenberg, Stephan; Kanis, Philipp; Macak, Dominik; Wollny, Damian; Düsterhöft, Dorothee; Kowalewski, Johannes; Helmbrecht, Nelly; Maricic, Tomislav; Pääbo, Svante (2023-07-20). "Efficient high-precision homology-directed repair-dependent genome editing by HDRobust". Nature Methods (en inglés) 20 (9): 1388–1399. ISSN 1548-7091. PMC 10482697. PMID 37474806. doi:10.1038/s41592-023-01949-1.
- ↑ Lino CA, Harper JC, Carney JP, Timlin JA (novembro de 2018). "Delivering CRISPR: a review of the challenges and approaches". Drug Delivery 25 (1): 1234–1257. PMC 6058482. PMID 29801422. doi:10.1080/10717544.2018.1474964.
- ↑ Li L, Hu S, Chen X (xullo de 2018). "Non-viral delivery systems for CRISPR/Cas9-based genome editing: Challenges and opportunities". Biomaterials 171: 207–218. PMC 5944364. PMID 29704747. doi:10.1016/j.biomaterials.2018.04.031.
- ↑ Jain PK, Lo JH, Rananaware S, Downing M, Panda A, Tai M, et al. (novembro de 2019). "Non-viral delivery of CRISPR/Cas9 complex using CRISPR-GPS nanocomplexes". Nanoscale 11 (44): 21317–21323. PMC 7709491. PMID 31670340. doi:10.1039/C9NR01786K.
- ↑ 79,0 79,1 Yip BH (maio de 2020). "Recent Advances in CRISPR/Cas9 Delivery Strategies". Biomolecules 10 (6): 839. PMC 7356196. PMID 32486234. doi:10.3390/biom10060839.
- ↑ Bak RO, Porteus MH (xullo de 2017). "CRISPR-Mediated Integration of Large Gene Cassettes Using AAV Donor Vectors". Cell Reports 20 (3): 750–756. PMC 5568673. PMID 28723575. doi:10.1016/j.celrep.2017.06.064.
- ↑ Schmidt F, Grimm D (febreiro de 2015). "CRISPR genome engineering and viral gene delivery: a case of mutual attraction". Biotechnology Journal 10 (2): 258–272. PMID 25663455. doi:10.1002/biot.201400529.
- ↑ Waxmonsky N (24 de setembro de 2015). "CRISPR 101: Mammalian Expression Systems and Delivery Methods". Consultado o 11 de xuño de 2018.
- ↑ Mishra T, Bhardwaj V, Ahuja N, Gadgil P, Ramdas P, Shukla S, Chande A (xuño de 2022). "Improved loss-of-function CRISPR-Cas9 genome editing in human cells concomitant with inhibition of TGF-β signaling". Molecular Therapy. Nucleic Acids 28: 202–218. PMC 8961078. PMID 35402072. doi:10.1016/j.omtn.2022.03.003.
- ↑ Adli M (maio de 2018). "The CRISPR tool kit for genome editing and beyond". Nature Communications 9 (1): 1911. Bibcode:2018NatCo...9.1911A. PMC 5953931. PMID 29765029. doi:10.1038/s41467-018-04252-2.
- ↑ Svitashev S, Schwartz C, Lenderts B, Young JK, Mark Cigan A (novembro de 2016). "Genome editing in maize directed by CRISPR-Cas9 ribonucleoprotein complexes". Nature Communications 7 (1): 13274. Bibcode:2016NatCo...713274S. PMC 5116081. PMID 27848933. doi:10.1038/ncomms13274.
- ↑ Liang Z, Chen K, Li T, Zhang Y, Wang Y, Zhao Q, et al. (xaneiro de 2017). "Efficient DNA-free genome editing of bread wheat using CRISPR/Cas9 ribonucleoprotein complexes". Nature Communications 8 (1): 14261. Bibcode:2017NatCo...814261L. PMC 5253684. PMID 28098143. doi:10.1038/ncomms14261.
- ↑ Cui Z, Tian R, Huang Z, Jin Z, Li L, Liu J, et al. (marzo de 2022). "FrCas9 is a CRISPR/Cas9 system with high editing efficiency and fidelity". Nature Communications 13 (1): 1425. Bibcode:2022NatCo..13.1425C. PMC 8931148. PMID 35301321. doi:10.1038/s41467-022-29089-8.
- ↑ Kleinstiver BP, Pattanayak V, Prew MS, Tsai SQ, Nguyen NT, Zheng Z, Joung JK (xaneiro de 2016). "High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects". Nature 529 (7587): 490–495. Bibcode:2016Natur.529..490K. PMC 4851738. PMID 26735016. doi:10.1038/nature16526.
- ↑ Slaymaker IM, Gao L, Zetsche B, Scott DA, Yan WX, Zhang F (xaneiro de 2016). "Rationally engineered Cas9 nucleases with improved specificity". Science 351 (6268): 84–88. Bibcode:2016Sci...351...84S. PMC 4714946. PMID 26628643. doi:10.1126/science.aad5227.
- ↑ Yin H, Song CQ, Suresh S, Kwan SY, Wu Q, Walsh S, et al. (marzo de 2018). "Partial DNA-guided Cas9 enables genome editing with reduced off-target activity". Nature Chemical Biology 14 (3): 311–316. PMC 5902734. PMID 29377001. doi:10.1038/nchembio.2559.
- ↑ Riesenberg S, Helmbrecht N, Kanis P, Maricic T, Pääbo S (xaneiro de 2022). "Improved gRNA secondary structures allow editing of target sites resistant to CRISPR-Cas9 cleavage". Nature Communications 13 (1): 489. Bibcode:2022NatCo..13..489R. PMC 8789806. PMID 35078986. doi:10.1038/s41467-022-28137-7.
- ↑ Fu Y, Sander JD, Reyon D, Cascio VM, Joung JK (marzo de 2014). "Improving CRISPR-Cas nuclease specificity using truncated guide RNAs". Nature Biotechnology 32 (3): 279–284. PMC 3988262. PMID 24463574. doi:10.1038/nbt.2808.
- ↑ Thean DG, Chu HY, Fong JH, Chan BK, Zhou P, Kwok CC, et al. (abril de 2022). "Machine learning-coupled combinatorial mutagenesis enables resource-efficient engineering of CRISPR-Cas9 genome editor activities". Nature Communications 13 (1): 2219. Bibcode:2022NatCo..13.2219T. PMC 9039034. PMID 35468907. doi:10.1038/s41467-022-29874-5.
- ↑ Cho SW, Kim S, Kim Y, Kweon J, Kim HS, Bae S, Kim JS (xaneiro de 2014). "Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases". Genome Research 24 (1): 132–141. PMC 3875854. PMID 24253446. doi:10.1101/gr.162339.113.
- ↑ 95,0 95,1 Oakes BL, Nadler DC, Flamholz A, Fellmann C, Staahl BT, Doudna JA, Savage DF (xuño de 2016). "Profiling of engineering hotspots identifies an allosteric CRISPR-Cas9 switch". Nature Biotechnology 34 (6): 646–651. PMC 4900928. PMID 27136077. doi:10.1038/nbt.3528.
- ↑ Nuñez JK, Harrington LB, Doudna JA (marzo de 2016). "Chemical and Biophysical Modulation of Cas9 for Tunable Genome Engineering". ACS Chemical Biology 11 (3): 681–688. PMID 26857072. doi:10.1021/acschembio.5b01019.
- ↑ Zhou W, Deiters A (abril de 2016). "Conditional Control of CRISPR/Cas9 Function". Angewandte Chemie 55 (18): 5394–5399. PMID 26996256. doi:10.1002/anie.201511441.
- ↑ Polstein LR, Gersbach CA (marzo de 2015). "A light-inducible CRISPR-Cas9 system for control of endogenous gene activation". Nature Chemical Biology 11 (3): 198–200. PMC 4412021. PMID 25664691. doi:10.1038/nchembio.1753.
- ↑ Nihongaki Y, Yamamoto S, Kawano F, Suzuki H, Sato M (febreiro de 2015). "CRISPR-Cas9-based photoactivatable transcription system". Chemistry & Biology 22 (2): 169–174. PMID 25619936. doi:10.1016/j.chembiol.2014.12.011.
- ↑ Wright AV, Sternberg SH, Taylor DW, Staahl BT, Bardales JA, Kornfeld JE, Doudna JA (marzo de 2015). "Rational design of a split-Cas9 enzyme complex". Proceedings of the National Academy of Sciences of the United States of America 112 (10): 2984–2989. Bibcode:2015PNAS..112.2984W. PMC 4364227. PMID 25713377. doi:10.1073/pnas.1501698112.
- ↑ Nihongaki Y, Kawano F, Nakajima T, Sato M (xullo de 2015). "Photoactivatable CRISPR-Cas9 for optogenetic genome editing". Nature Biotechnology 33 (7): 755–760. PMID 26076431. doi:10.1038/nbt.3245.
- ↑ Hemphill J, Borchardt EK, Brown K, Asokan A, Deiters A (maio de 2015). "Optical Control of CRISPR/Cas9 Gene Editing". Journal of the American Chemical Society 137 (17): 5642–5645. PMC 4919123. PMID 25905628. doi:10.1021/ja512664v.
- ↑ Jain PK, Ramanan V, Schepers AG, Dalvie NS, Panda A, Fleming HE, Bhatia SN (seembro de 2016). "Development of Light-Activated CRISPR Using Guide RNAs with Photocleavable Protectors". Angewandte Chemie 55 (40): 12440–12444. PMC 5864249. PMID 27554600. doi:10.1002/anie.201606123.
- ↑ Davis KM, Pattanayak V, Thompson DB, Zuris JA, Liu DR (maio de 2015). "Small molecule-triggered Cas9 protein with improved genome-editing specificity". Nature Chemical Biology 11 (5): 316–318. PMC 4402137. PMID 25848930. doi:10.1038/nchembio.1793.
- ↑ Liu KI, Ramli MN, Woo CW, Wang Y, Zhao T, Zhang X, et al. (novembro de 2016). "A chemical-inducible CRISPR-Cas9 system for rapid control of genome editing". Nature Chemical Biology 12 (11): 980–987. PMID 27618190. doi:10.1038/nchembio.2179.
- ↑ Truong DJ, Kühner K, Kühn R, Werfel S, Engelhardt S, Wurst W, Ortiz O (xullo de 2015). "Development of an intein-mediated split-Cas9 system for gene therapy". Nucleic Acids Research 43 (13): 6450–6458. PMC 4513872. PMID 26082496. doi:10.1093/nar/gkv601.
- ↑ Zetsche B, Volz SE, Zhang F (febreiro de 2015). "A split-Cas9 architecture for inducible genome editing and transcription modulation". Nature Biotechnology 33 (2): 139–142. PMC 4503468. PMID 25643054. doi:10.1038/nbt.3149.
- ↑ González F, Zhu Z, Shi ZD, Lelli K, Verma N, Li QV, Huangfu D (agosto de 2014). "An iCRISPR platform for rapid, multiplexable, and inducible genome editing in human pluripotent stem cells". Cell Stem Cell 15 (2): 215–226. PMC 4127112. PMID 24931489. doi:10.1016/j.stem.2014.05.018.
- ↑ Dow LE, Fisher J, O'Rourke KP, Muley A, Kastenhuber ER, Livshits G, et al. (abril de 2015). "Inducible in vivo genome editing with CRISPR-Cas9". Nature Biotechnology 33 (4): 390–394. PMC 4390466. PMID 25690852. doi:10.1038/nbt.3155.
- ↑ Yu C, Liu Y, Ma T, Liu K, Xu S, Zhang Y, et al. (febreiro de 2015). "Small molecules enhance CRISPR genome editing in pluripotent stem cells". Cell Stem Cell 16 (2): 142–147. PMC 4461869. PMID 25658371. doi:10.1016/j.stem.2015.01.003.
- ↑ Schimmel, J.; Muñoz-Subirana, N.; Kool, H.; Van Schendel, R.; Van Der Vlies, S.; Kamp, J. A.; De Vrij, F.; Kushner, S. A.; Smith GCM; Boulton, S. J.; Tijsterman, M. (2023). "Modulating mutational outcomes and improving precise gene editing at CRISPR-Cas9-induced breaks by chemical inhibition of end-joining pathways". Cell Reports 42 (2): 112019. PMID 36701230. doi:10.1016/j.celrep.2023.112019.
- ↑ Maruyama T, Dougan SK, Truttmann MC, Bilate AM, Ingram JR, Ploegh HL (maio de 2015). "Increasing the efficiency of precise genome editing with CRISPR-Cas9 by inhibition of nonhomologous end joining". Nature Biotechnology 33 (5): 538–542. PMC 4618510. PMID 25798939. doi:10.1038/nbt.3190.
- ↑ 113,0 113,1 Ma X, Chen X, Jin Y, Ge W, Wang W, Kong L, et al. (abril de 2018). "Small molecules promote CRISPR-Cpf1-mediated genome editing in human pluripotent stem cells". Nature Communications 9 (1): 1303. Bibcode:2018NatCo...9.1303M. PMC 5880812. PMID 29610531. doi:10.1038/s41467-018-03760-5.
- ↑ Nishida K, Arazoe T, Yachie N, Banno S, Kakimoto M, Tabata M, et al. (setembro de 2016). "Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems". Science 353 (6305): aaf8729. PMID 27492474. doi:10.1126/science.aaf8729.
- ↑ Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR (maio de 2016). "Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage". Nature 533 (7603): 420–424. Bibcode:2016Natur.533..420K. PMC 4873371. PMID 27096365. doi:10.1038/nature17946.
- ↑ Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR (novembro de 2017). "Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage". Nature 551 (7681): 464–471. Bibcode:2017Natur.551..464G. PMC 5726555. PMID 29160308. doi:10.1038/nature24644.
- ↑ Chen L, Park JE, Paa P, Rajakumar PD, Prekop HT, Chew YT, et al. (marzo de 2021). "Programmable C:G to G:C genome editing with CRISPR-Cas9-directed base excision repair proteins". Nature Communications 12 (1): 1384. Bibcode:2021NatCo..12.1384C. PMC 7925527. PMID 33654077. doi:10.1038/s41467-021-21559-9.
- ↑ 118,0 118,1 118,2 Kurata M, Yamamoto K, Moriarity BS, Kitagawa M, Largaespada DA (febreiro de 2018). "CRISPR/Cas9 library screening for drug target discovery". Journal of Human Genetics 63 (2): 179–186. PMID 29158600. doi:10.1038/s10038-017-0376-9.
- ↑ Gasiunas G, Barrangou R, Horvath P, Siksnys V (setembro de 2012). "Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria". Proceedings of the National Academy of Sciences of the United States of America 109 (39): E2579–E2586. PMC 3465414. PMID 22949671. doi:10.1073/pnas.1208507109.
- ↑ Satomura A, Nishioka R, Mori H, Sato K, Kuroda K, Ueda M (maio de 2017). "Precise genome-wide base editing by the CRISPR Nickase system in yeast". Scientific Reports 7 (1): 2095. Bibcode:2017NatSR...7.2095S. PMC 5437071. PMID 28522803. doi:10.1038/s41598-017-02013-7.
- ↑ Hiranniramol K, Chen Y, Liu W, Wang X (maio de 2020). "Generalizable sgRNA design for improved CRISPR/Cas9 editing efficiency". Bioinformatics 36 (9): 2684–2689. PMC 7203743. PMID 31971562. doi:10.1093/bioinformatics/btaa041.
- ↑ 122,0 122,1 122,2 Agrotis A, Ketteler R (2015-09-24). "A new age in functional genomics using CRISPR/Cas9 in arrayed library screening". Frontiers in Genetics 6: 300. PMC 4585242. PMID 26442115. doi:10.3389/fgene.2015.00300.
- ↑ Yu JS, Yusa K (xullo de 2019). "Genome-wide CRISPR-Cas9 screening in mammalian cells". Methods. 164-165: 29–35. PMID 31034882. doi:10.1016/j.ymeth.2019.04.015.
- ↑ 124,0 124,1 Joung J, Konermann S, Gootenberg JS, Abudayyeh OO, Platt RJ, Brigham MD, et al. (abril de 2017). "Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening". Nature Protocols 12 (4): 828–863. PMC 5526071. PMID 28333914. doi:10.1038/nprot.2017.016.
- ↑ "Addgene: Pooled Libraries". www.addgene.org. Consultado o 2020-01-31.
- ↑ McDade JR, Waxmonsky NC, Swanson LE, Fan M (xullo de 2016). "Practical Considerations for Using Pooled Lentiviral CRISPR Libraries". Current Protocols in Molecular Biology 115 (1): 31.5.1–31.5.13. PMID 27366891. doi:10.1002/cpmb.8.
- ↑ Cheng AW, Wang H, Yang H, Shi L, Katz Y, Theunissen TW, et al. (outubro de 2013). "Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system". Cell Research 23 (10): 1163–1171. PMC 3790238. PMID 23979020. doi:10.1038/cr.2013.122.
- ↑ Gilbert LA, Horlbeck MA, Adamson B, Villalta JE, Chen Y, Whitehead EH, et al. (outubro de 2014). "Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation". Cell 159 (3): 647–661. PMC 4253859. PMID 25307932. doi:10.1016/j.cell.2014.09.029.
- ↑ 129,0 129,1 Dow LE (outuro de 2015). "Modeling Disease In Vivo With CRISPR/Cas9". Trends in Molecular Medicine 21 (10): 609–621. PMC 4592741. PMID 26432018. doi:10.1016/j.molmed.2015.07.006.
- ↑ Doudna J, Mali P (2016). CRISPR-Cas: A Laboratory Manual. Cold Spring Harbor, New York. ISBN 978-1-62182-130-4. OCLC 922914104.
- ↑ Zuo E, Huo X, Yao X, Hu X, Sun Y, Yin J, et al. (novembro de 2017). "CRISPR/Cas9-mediated targeted chromosome elimination". Genome Biology 18 (1): 224. PMC 5701507. PMID 29178945. doi:10.1186/s13059-017-1354-4.
- "CRISPR Used to Eliminate Targeted Chromosomes in New Study". Genome Web. 27 de novembro de 2017.
- ↑ Javed MR, Sadaf M, Ahmed T, Jamil A, Nawaz M, Abbas H, Ijaz A (decembro de 2018). "CRISPR-Cas System: History and Prospects as a Genome Editing Tool in Microorganisms". review. Current Microbiology 75 (12): 1675–1683. PMID 30078067. doi:10.1007/s00284-018-1547-4.
- ↑ DiCarlo JE, Norville JE, Mali P, Rios X, Aach J, Church GM (abril de 2013). "Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems". Nucleic Acids Res 41 (7): 4336–43. PMC 3627607. PMID 23460208. doi:10.1093/nar/gkt135.
- ↑ Giersch RM, Finnigan GC (decembro de 2017). "Yeast Still a Beast: Diverse Applications of CRISPR/Cas Editing Technology in S. cerevisiae". The Yale Journal of Biology and Medicine 90 (4): 643–651. PMC 5733842. PMID 29259528.
- ↑ Dhamad AE, Lessner DJ (outubro de 2020). Atomi H, ed. "A CRISPRi-dCas9 System for Archaea and Its Use To Examine Gene Function during Nitrogen Fixation by Methanosarcina acetivorans". Applied and Environmental Microbiology 86 (21): e01402–20. Bibcode:2020ApEnM..86E1402D. PMC 7580536. PMID 32826220. doi:10.1128/AEM.01402-20.
- ↑ Raschmanová H, Weninger A, Glieder A, Kovar K, Vogl T (2018). "Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts: Current state and future prospects". review. Biotechnology Advances 36 (3): 641–665. PMID 29331410. doi:10.1016/j.biotechadv.2018.01.006.
- ↑ 137,0 137,1 Ma D, Liu F (decembro de 2015). "Genome Editing and Its Applications in Model Organisms". review. Genomics, Proteomics & Bioinformatics 13 (6): 336–344. PMC 4747648. PMID 26762955. doi:10.1016/j.gpb.2015.12.001.
- ↑ Khurshid H, Jan SA, Shinwari ZK, Jamal M, Shah SH (2018). "An Era of CRISPR/ Cas9 Mediated Plant Genome Editing". review. Current Issues in Molecular Biology 26: 47–54. PMID 28879855. doi:10.21775/cimb.026.047.
- ↑ Simone BW, Martínez-Gálvez G, WareJoncas Z, Ekker SC (novembro de 2018). "Fishing for understanding: Unlocking the zebrafish gene editor's toolbox". review. Methods 150: 3–10. PMC 6590056. PMID 30076892. doi:10.1016/j.ymeth.2018.07.012.
- ↑ Singh P, Schimenti JC, Bolcun-Filas E (xaneiro de 2015). "A mouse geneticist's practical guide to CRISPR applications". review. Genetics 199 (1): 1–15. PMC 4286675. PMID 25271304. doi:10.1534/genetics.114.169771.
- ↑ Soni D, Wang DM, Regmi SC, Mittal M, Vogel SM, Schlüter D, Tiruppathi C (maio de 2018). "Deubiquitinase function of A20 maintains and repairs endothelial barrier after lung vascular injury". Cell Death Discovery 4 (60): 60. PMC 5955943. PMID 29796309. doi:10.1038/s41420-018-0056-3.
- ↑ Birling MC, Herault Y, Pavlovic G (agosto de 2017). "Modeling human disease in rodents by CRISPR/Cas9 genome editing". Mammalian Genome 28 (7–8): 291–301. PMC 5569124. PMID 28677007. doi:10.1007/s00335-017-9703-x.
- ↑ Gao X, Tao Y, Lamas V, Huang M, Yeh WH, Pan B, et al. (xaneiro de 2018). "Treatment of autosomal dominant hearing loss by in vivo delivery of genome editing agents". Nature 553 (7687): 217–221. Bibcode:2018Natur.553..217G. PMC 5784267. PMID 29258297. doi:10.1038/nature25164.
- ↑ Kadam US, Shelake RM, Chavhan RL, Suprasanna P (outubro de 2018). "Concerns regarding 'off-target' activity of genome editing endonucleases". review. Plant Physiology and Biochemistry 131: 22–30. PMID 29653762. doi:10.1016/j.plaphy.2018.03.027.
- ↑ Kimberland ML, Hou W, Alfonso-Pecchio A, Wilson S, Rao Y, Zhang S, Lu Q (outubro de 2018). "Strategies for controlling CRISPR/Cas9 off-target effects and biological variations in mammalian genome editing experiments". review. Journal of Biotechnology 284: 91–101. PMID 30142414. doi:10.1016/j.jbiotec.2018.08.007.
- ↑ van Erp PB, Bloomer G, Wilkinson R, Wiedenheft B (xuño de 2015). "The history and market impact of CRISPR RNA-guided nucleases". Current Opinion in Virology 12: 85–90. PMC 4470805. PMID 25914022. doi:10.1016/j.coviro.2015.03.011.
- ↑ Maggio I, Gonçalves MA (maio de 2015). "Genome editing at the crossroads of delivery, specificity, and fidelity". Trends in Biotechnology 33 (5): 280–291. PMID 25819765. doi:10.1016/j.tibtech.2015.02.011.
- ↑ Rath D, Amlinger L, Rath A, Lundgren M (outubro de 2015). "The CRISPR-Cas immune system: biology, mechanisms and applications". Biochimie 117: 119–128. PMID 25868999. doi:10.1016/j.biochi.2015.03.025.
- ↑ "What Is CRISPR? How Does It Work? Is It Gene Editing?". LiveScience.Tech (en inglés). 2018-04-30. Arquivado dende o orixinal o 2020-02-06. Consultado o 2020-02-06.
- ↑ 150,0 150,1 Freedman BS, Brooks CR, Lam AQ, Fu H, Morizane R, Agrawal V, et al. (outubro de 2015). "Modelling kidney disease with CRISPR-mutant kidney organoids derived from human pluripotent epiblast spheroids". Nature Communications 6: 8715. Bibcode:2015NatCo...6.8715F. PMC 4620584. PMID 26493500. doi:10.1038/ncomms9715.
- ↑ Cruz NM, Song X, Czerniecki SM, Gulieva RE, Churchill AJ, Kim YK, et al. (novembro de 2017). "Organoid cystogenesis reveals a critical role of microenvironment in human polycystic kidney disease". Nature Materials 16 (11): 1112–1119. Bibcode:2017NatMa..16.1112C. PMC 5936694. PMID 28967916. doi:10.1038/nmat4994.
- ↑ Kim YK, Refaeli I, Brooks CR, Jing P, Gulieva RE, Hughes MR, et al. (decembro de 2017). "Gene-Edited Human Kidney Organoids Reveal Mechanisms of Disease in Podocyte Development". Stem Cells 35 (12): 2366–2378. PMC 5742857. PMID 28905451. doi:10.1002/stem.2707.
- ↑ Bellin M, Casini S, Davis RP, D'Aniello C, Haas J, Ward-van Oostwaard D, et al. (decembro de 2013). "Isogenic human pluripotent stem cell pairs reveal the role of a KCNH2 mutation in long-QT syndrome". The EMBO Journal 32 (24): 3161–3175. PMC 3981141. PMID 24213244. doi:10.1038/emboj.2013.240.
- ↑ Cai L, Fisher AL, Huang H, Xie Z (decembro de 2016). "CRISPR-mediated genome editing and human diseases". Genes & Diseases 3 (4): 244–251. PMC 6150104. PMID 30258895. doi:10.1016/j.gendis.2016.07.003.
- ↑ "Seven Diseases That CRISPR Technology Could Cure". Labiotech.eu. 2018-06-25. Consultado o 2018-08-22.
- ↑ 156,0 156,1 "CRISPR/Cas9 and Cancer". Immuno-Oncology News (en inglés). 2018-04-27. Consultado o 2019-02-18.
- ↑ Crossley M (febreiro de 2021). "New CRISPR technology could revolutionise gene therapy, offering new hope to people with genetic diseases". The Conversation (en inglés). Consultado o 2021-02-03.
- ↑ Cromer MK, Camarena J, Martin RM, Lesch BJ, Vakulskas CA, Bode NM, et al. (abril de 2021). "Gene replacement of α-globin with β-globin restores hemoglobin balance in β-thalassemia-derived hematopoietic stem and progenitor cells". Nature Medicine 27 (4): 677–687. PMC 8265212. PMID 33737751. doi:10.1038/s41591-021-01284-y.
- ↑ Xie F, Ye L, Chang JC, Beyer AI, Wang J, Muench MO, Kan YW (setembro de 2014). "Seamless gene correction of β-thalassemia mutations in patient-specific iPSCs using CRISPR/Cas9 and piggyBac". Genome Research 24 (9): 1526–1533. PMC 4158758. PMID 25096406. doi:10.1101/gr.173427.114.
- ↑ 160,0 160,1 Frangoul H, Altshuler D, Cappellini MD, Chen YS, Domm J, Eustace BK, et al. (xaneiro de 2021). "CRISPR-Cas9 Gene Editing for Sickle Cell Disease and β-Thalassemia". The New England Journal of Medicine 384 (3): 252–260. PMID 33283989. doi:10.1056/NEJMoa2031054.
- ↑ Dever DP, Bak RO, Reinisch A, Camarena J, Washington G, Nicolas CE, et al. (novembro de 2016). "CRISPR/Cas9 β-globin gene targeting in human haematopoietic stem cells". Nature 539 (7629): 384–389. Bibcode:2016Natur.539..384D. PMC 5898607. PMID 27820943. doi:10.1038/nature20134.
- ↑ "CRISPR 'One Shot' Cell Therapy for Hemophilia Developed". GEN. 2018-05-02. Consultado o 2018-08-22.
- ↑ Marangi M, Pistritto G (2018-04-20). "Innovative Therapeutic Strategies for Cystic Fibrosis: Moving Forward to CRISPR Technique". Frontiers in Pharmacology 9: 396. PMC 5920621. PMID 29731717. doi:10.3389/fphar.2018.00396.
- ↑ Bengtsson NE, Hall JK, Odom GL, Phelps MP, Andrus CR, Hawkins RD, et al. (febreiro de 2017). "Muscle-specific CRISPR/Cas9 dystrophin gene editing ameliorates pathophysiology in a mouse model for Duchenne muscular dystrophy". Nature Communications 8: 14454. Bibcode:2017NatCo...814454B. PMC 5316861. PMID 28195574. doi:10.1038/ncomms14454.
- ↑ Eisenstein M (maio de 2018). "CRISPR takes on Huntington's disease". Nature 557 (7707): S42–S43. Bibcode:2018Natur.557S..42E. PMID 29844549. doi:10.1038/d41586-018-05177-y.
- ↑ Dabrowska M, Juzwa W, Krzyzosiak WJ, Olejniczak M (2018). "Precise Excision of the CAG Tract from the Huntingtin Gene by Cas9 Nickases". Frontiers in Neuroscience 12: 75. PMC 5834764. PMID 29535594. doi:10.3389/fnins.2018.00075.
- ↑ King A (marzo de 2018). "A CRISPR edit for heart disease". Nature 555 (7695): S23–S25. Bibcode:2018Natur.555.....K. PMID 29517035. doi:10.1038/d41586-018-02482-4.
- ↑ Scudellari M (xullo de 2019). "Self-destructing mosquitoes and sterilized rodents: the promise of gene drives". Nature 571 (7764): 160–162. Bibcode:2019Natur.571..160S. PMID 31289403. doi:10.1038/d41586-019-02087-5.
- ↑ Abrahimi P, Chang WG, Kluger MS, Qyang Y, Tellides G, Saltzman WM, Pober JS (xullo de 2015). "Efficient gene disruption in cultured primary human endothelial cells by CRISPR/Cas9". Circulation Research 117 (2): 121–128. PMC 4490936. PMID 25940550. doi:10.1161/CIRCRESAHA.117.306290.
- ↑ "A Year In, 1st Patient To Get Gene Editing For Sickle Cell Disease Is Thriving". NPR.org (en inglés). Consultado o 2021-02-03.
- ↑ "CRISPR technology to cure sickle cell disease". ScienceDaily (en inglés). Consultado o 2021-02-03.
- ↑ Uddin F, Rudin CM, Sen T (agosto 2020). "CRISPR Gene Therapy: Applications, Limitations, and Implications for the Future". Frontiers in Oncology 10: 1387. PMC 7427626. PMID 32850447. doi:10.3389/fonc.2020.01387.
- ↑ 173,0 173,1 173,2 173,3 "CRISPR Clinical Trials: A 2022 Update". Innovative Genomics Institute (IGI) (en inglés). Consultado o 2022-05-02.
- ↑ "Editas Hits Pause on LCA10 Program in Search of Partner".
- ↑ Jensen TI, Axelgaard E, Bak RO (xuño de 2019). "Therapeutic gene editing in haematological disorders with CRISPR/Cas9". British Journal of Haematology 185 (5): 821–835. PMID 30864164. doi:10.1111/bjh.15851.
- ↑ Reardon S (2016). "First CRISPR clinical trial gets green light from US panel". Nature. doi:10.1038/nature.2016.20137.
- ↑ Rosenblum D, Gutkin A, Kedmi R, Ramishetti S, Veiga N, Jacobi AM, et al. (novembro de 2020). "CRISPR-Cas9 genome editing using targeted lipid nanoparticles for cancer therapy". Science Advances 6 (47): eabc9450. Bibcode:2020SciA....6.9450R. PMC 7673804. PMID 33208369. doi:10.1126/sciadv.abc9450.
- ↑ "Base editing: Revolutionary therapy clears girl's incurable cancer". BBC News. 11 de decembro de 2022. Consultado o 11 de decembro de 2022.
- ↑ "'Designer cells' reverse one-year-old's cancer". BBC News (en inglés). 2015-11-05. Consultado o 2022-12-11.
- ↑ "CRISPR Therapeutics and ViaCyte, Inc. Announce First Patient Dosed in…". CRISPR (en inglés). Consultado o 2022-05-02.
- ↑ Gomaa AA, Klumpe HE, Luo ML, Selle K, Barrangou R, Beisel CL (xaneiro de 2014). "Programmable removal of bacterial strains by use of genome-targeting CRISPR-Cas systems". mBio 5 (1): e00928–e00913. PMC 3903277. PMID 24473129. doi:10.1128/mBio.00928-13.
- ↑ Citorik RJ, Mimee M, Lu TK (novembro de 2014). "Sequence-specific antimicrobials using efficiently delivered RNA-guided nucleases". Nature Biotechnology 32 (11): 1141–1145. PMC 4237163. PMID 25240928. doi:10.1038/nbt.3011. hdl:1721.1/100834.
- ↑ 183,0 183,1 Gholizadeh P, Aghazadeh M, Asgharzadeh M, Kafil HS (novembro de 2017). "Suppressing the CRISPR/Cas adaptive immune system in bacterial infections". European Journal of Clinical Microbiology & Infectious Diseases 36 (11): 2043–2051. PMID 28601970. doi:10.1007/s10096-017-3036-2.
- ↑ Gibney E (xaneiro de 2018). "What to expect in 2018: science in the new year". Nature 553 (7686): 12–13. Bibcode:2018Natur.553...12G. PMID 29300040. doi:10.1038/d41586-018-00009-5.
- ↑ Taylor P (3 de xaneiro de 2019). "J&J takes stake in Locus' CRISPR-based 'Pac-Man' antimicrobials". Fierce Biotech. Consultado o 27 de febreiro de 2019.
- ↑ Reardon S (xuño de 2017). "Modified viruses deliver death to antibiotic-resistant bacteria". Nature 546 (7660): 586–587. Bibcode:2017Natur.546..586R. PMID 28661508. doi:10.1038/nature.2017.22173.
- ↑ van Diemen FR, Kruse EM, Hooykaas MJ, Bruggeling CE, Schürch AC, van Ham PM, et al. (xuño de 2016). "CRISPR/Cas9-Mediated Genome Editing of Herpesviruses Limits Productive and Latent Infections". PLOS Pathogens 12 (6): e1005701. PMC 4928872. PMID 27362483. doi:10.1371/journal.ppat.1005701.
- "Using CRISPR to combat viral infections: a new way to treat herpes?". PLOS Media. 4 de agosto de 2016 – vía YouTube.
- ↑ National Institute on Drug Abuse (2020-02-14). "Antiretroviral Therapy Combined With CRISPR Gene Editing Can Eliminate HIV Infection in Mice". National Institute on Drug Abuse (en inglés). Consultado o 2022-04-18.
- ↑ "First Clinical Trial of CRISPR-Based HIV Therapy Founded on Breakthrough Research at Lewis Katz School of Medicine | Lewis Katz School of Medicine at Temple University". medicine.temple.edu (en inglés). Consultado o 2022-04-18.
- ↑ Excision BioTherapeutics (2022-03-24). "A Phase 1/2a, Sequential Cohort, Single Ascending Dose Study of the Safety, Tolerability, Biodistribution, and Pharmacodynamics of EBT 101 in Aviremic HIV-1 Infected Adults on Stable Antiretroviral Therapy".
- ↑ 191,0 191,1 191,2 Science News Staff (17 de decembro de 2015). "And Science's Breakthrough of the Year is ...". news.sciencemag.org. Consultado o 2015-12-21.
- ↑ Mullin E. "Using CRISPR on pigs could make their organs safer for human transplant". MIT Technology Review. Consultado o 2017-09-09.
- ↑ Pinson, Anneline; Xing, Lei; Namba, Takashi; Kalebic, Nereo; Peters, Jula; Oegema, Christina Eugster; Traikov, Sofia; Reppe, Katrin; Riesenberg, Stephan; Maricic, Tomislav; Derihaci, Razvan; Wimberger, Pauline; Pääbo, Svante; Huttner, Wieland B. (2022-09-09). "Human TKTL1 implies greater neurogenesis in frontal neocortex of modern humans than Neanderthals". Science (en inglés) 377 (6611): eabl6422. ISSN 0036-8075. PMID 36074851. doi:10.1126/science.abl6422.
- ↑ Mora-Bermúdez, Felipe; Kanis, Philipp; Macak, Dominik; Peters, Jula; Naumann, Ronald; Xing, Lei; Sarov, Mihail; Winkler, Sylke; Oegema, Christina Eugster; Haffner, Christiane; Wimberger, Pauline; Riesenberg, Stephan; Maricic, Tomislav; Huttner, Wieland B.; Pääbo, Svante (2022-07-29). "Longer metaphase and fewer chromosome segregation errors in modern human than Neanderthal brain development". Science Advances (en inglés) 8 (30): eabn7702. Bibcode:2022SciA....8N7702M. ISSN 2375-2548. PMC 9337762. PMID 35905187. doi:10.1126/sciadv.abn7702.
- ↑ "Scientists discover how humans develop larger brains than other apes". phys.org (en inglés). Arquivado dende o orixinal o 19 de abril de 2021. Consultado o 19 de abril de 2021.
- ↑ Benito-Kwiecinski S, Giandomenico SL, Sutcliffe M, Riis ES, Freire-Pritchett P, Kelava I, et al. (abril de 2021). "An early cell shape transition drives evolutionary expansion of the human forebrain". Cell (en English) 184 (8): 2084–2102.e19. PMC 8054913. PMID 33765444. doi:10.1016/j.cell.2021.02.050.
 Dispoñible baixo licenza CC BY 4.0 Arquivado 2017-10-16 en Wayback Machine..
Dispoñible baixo licenza CC BY 4.0 Arquivado 2017-10-16 en Wayback Machine..
- ↑ Sawal I. "Mini brains genetically altered with CRISPR to be Neanderthal-like". New Scientist. Arquivado dende o orixinal o 10 de marzo de 2021. Consultado o 7 de marzo de 2021.
- ↑ Trujillo CA, Rice ES, Schaefer NK, Chaim IA, Wheeler EC, Madrigal AA, et al. (febreiro de 2021). "Reintroduction of the archaic variant of NOVA1 in cortical organoids alters neurodevelopment". Science 371 (6530). PMC 8006534. PMID 33574182. doi:10.1126/science.aax2537.
- ↑ Maricic, Tomislav; Helmbrecht, Nelly; Riesenberg, Stephan; Macak, Dominik; Kanis, Philipp; Lackner, Martin; Pugach-Matveeva, Agrafena Daria; Pääbo, Svante (2021-10-15). "Comment on "Reintroduction of the archaic variant of NOVA1 in cortical organoids alters neurodevelopment"". Science (en inglés) 374 (6565): eabi6060. ISSN 0036-8075. PMID 34648345. doi:10.1126/science.abi6060.
- ↑ Herai, Roberto H.; Szeto, Ryan A.; Trujillo, Cleber A.; Muotri, Alysson R. (2021-10-15). "Response to Comment on "Reintroduction of the archaic variant of NOVA1 in cortical organoids alters neurodevelopment"". Science (en inglés) 374 (6565): eabi9881. ISSN 0036-8075. PMID 34648331. doi:10.1126/science.abi9881.
- ↑ Riesenberg, Stephan; Kanis, Philipp; Macak, Dominik; Wollny, Damian; Düsterhöft, Dorothee; Kowalewski, Johannes; Helmbrecht, Nelly; Maricic, Tomislav; Pääbo, Svante (2023-07-20). "Efficient high-precision homology-directed repair-dependent genome editing by HDRobust". Nature Methods (en inglés) 20 (9): 1388–1399. ISSN 1548-7091. PMC 10482697. PMID 37474806. doi:10.1038/s41592-023-01949-1.
- ↑ Dominguez AA, Lim WA, Qi LS (xaneiro de 2016). "Beyond editing: repurposing CRISPR-Cas9 for precision genome regulation and interrogation". Nature Reviews. Molecular Cell Biology 17 (1): 5–15. PMC 4922510. PMID 26670017. doi:10.1038/nrm.2015.2.
- ↑ Shalem O, Sanjana NE, Hartenian E, Shi X, Scott DA, Mikkelson T, et al. (xaneiro 2014). "Genome-scale CRISPR-Cas9 knockout screening in human cells". Science 343 (6166): 84–87. Bibcode:2014Sci...343...84S. PMC 4089965. PMID 24336571. doi:10.1126/science.1247005.
- ↑ 204,0 204,1 Pennisi E (agosto de 2013). "The CRISPR craze". News Focus. Science 341 (6148): 833–836. Bibcode:2013Sci...341..833P. PMID 23970676. doi:10.1126/science.341.6148.833.
- ↑ 205,0 205,1 Zimmer C (2016-06-03). "Scientists Find Form of CRISPR Gene Editing With New Capabilities". The New York Times. ISSN 0362-4331. Consultado o 2016-06-10.
- ↑ Pickar-Oliver A, Gersbach CA (agosto de 2019). "The next generation of CRISPR-Cas technologies and applications". Nature Reviews. Molecular Cell Biology 20 (8): 490–507. PMC 7079207. PMID 31147612. doi:10.1038/s41580-019-0131-5.
- ↑ Basak J, Nithin C (2015). "Targeting Non-Coding RNAs in Plants with the CRISPR-Cas Technology is a Challenge yet Worth Accepting". Frontiers in Plant Science 6: 1001. PMC 4652605. PMID 26635829. doi:10.3389/fpls.2015.01001.
- ↑ 208,0 208,1 Woolf TM, Chase JM, Stinchcomb DT (agosto de 1995). "Toward the therapeutic editing of mutated RNA sequences". Proceedings of the National Academy of Sciences of the United States of America 92 (18): 8298–8302. Bibcode:1995PNAS...92.8298W. PMC 41144. PMID 7545300. doi:10.1073/pnas.92.18.8298.
- ↑ Montiel-Gonzalez MF, Vallecillo-Viejo I, Yudowski GA, Rosenthal JJ (novembro de 2013). "Correction of mutations within the cystic fibrosis transmembrane conductance regulator by site-directed RNA editing". Proceedings of the National Academy of Sciences of the United States of America 110 (45): 18285–18290. Bibcode:2013PNAS..11018285M. PMC 3831439. PMID 24108353. doi:10.1073/pnas.1306243110.
- ↑ Cox DB, Gootenberg JS, Abudayyeh OO, Franklin B, Kellner MJ, Joung J, Zhang F (novembro de 2017). "RNA editing with CRISPR-Cas13". Science 358 (6366): 1019–1027. Bibcode:2017Sci...358.1019C. PMC 5793859. PMID 29070703. doi:10.1126/science.aaq0180.
- ↑ Williams S. "Neuroscientists expand CRISPR toolkit with new, compact Cas7-11 enzyme". Massachusetts Institute of Technology (en inglés). Consultado o 22 de xuño de 2022.
- ↑ Kato K, Zhou W, Okazaki S, Isayama Y, Nishizawa T, Gootenberg JS, et al. (xuño de 2022). "Structure and engineering of the type III-E CRISPR-Cas7-11 effector complex". Cell 185 (13): 2324–2337.e16. PMID 35643083. doi:10.1016/j.cell.2022.05.003.
- ↑ Özcan A, Krajeski R, Ioannidi E, Lee B, Gardner A, Makarova KS, et al. (setembro de 2021). "Programmable RNA targeting with the single-protein CRISPR effector Cas7-11". Nature 597 (7878): 720–725. Bibcode:2021Natur.597..720O. PMID 34489594. doi:10.1038/s41586-021-03886-5.
- ↑ Cross R (25 de marzo de 2019). "Watch out, CRISPR. The RNA editing race is on". Chemical & Engineering News (en inglés) 97 (12). Consultado o 30 de setembro de 2020.
- ↑ Akbari OS, Bellen HJ, Bier E, Bullock SL, Burt A, Church GM, et al. (agosto de 2015). "BIOSAFETY. Safeguarding gene drive experiments in the laboratory". Science 349 (6251): 927–929. Bibcode:2015Sci...349..927A. PMC 4692367. PMID 26229113. doi:10.1126/science.aac7932.
- ↑ Caplan AL, Parent B, Shen M, Plunkett C (novembro de 2015). "No time to waste—the ethical challenges created by CRISPR: CRISPR/Cas, being an efficient, simple, and cheap technology to edit the genome of any organism, raises many ethical and regulatory issues beyond the use to manipulate human germ line cells". EMBO Reports 16 (11): 1421–1426. PMC 4641494. PMID 26450575. doi:10.15252/embr.201541337.
- ↑ Oye KA, Esvelt K, Appleton E, Catteruccia F, Church G, Kuiken T, et al. (agosto de 2014). "Biotechnology. Regulating gene drives". Science 345 (6197): 626–628. Bibcode:2014Sci...345..626O. PMID 25035410. doi:10.1126/science.1254287.
- ↑ Gu W, Crawford ED, O'Donovan BD, Wilson MR, Chow ED, Retallack H, DeRisi JL (marzo de 2016). "Depletion of Abundant Sequences by Hybridization (DASH): using Cas9 to remove unwanted high-abundance species in sequencing libraries and molecular counting applications". Genome Biology 17: 41. PMC 4778327. PMID 26944702. doi:10.1186/s13059-016-0904-5.
- ↑ 219,0 219,1 Kungulovski, Goran; Jeltsch, Albert (1 de febreiro de 2016). "Epigenome Editing: State of the Art, Concepts, and Perspectives". Trends in Genetics (en inglés) 32 (2): 101–113. ISSN 0168-9525. PMID 26732754. doi:10.1016/j.tig.2015.12.001. Consultado o 30 de abril de 2021.
- ↑ Laufer, Benjamin I.; Singh, Shiva M. (17 de setembro de 2015). "Strategies for precision modulation of gene expression by epigenome editing: an overview". Epigenetics & Chromatin 8 (1): 34. ISSN 1756-8935. PMC 4574080. PMID 26388942. doi:10.1186/s13072-015-0023-7.
- ↑ "Unique CRISPR gene therapy offers opioid-free chronic pain treatment". New Atlas. 11 de marzo de 2021. Consultado o 18 de abril de 2021.
- ↑ Moreno, Ana M.; Alemán, Fernando; Catroli, Glaucilene F.; Hunt, Matthew; Hu, Michael; Dailamy, Amir; Pla, Andrew; Woller, Sarah A.; Palmer, Nathan; Parekh, Udit; McDonald, Daniella; Roberts, Amanda J.; Goodwill, Vanessa; Dryden, Ian; Hevner, Robert F.; Delay, Lauriane; Santos, Gilson Gonçalves dos; Yaksh, Tony L.; Mali, Prashant (10 de marzo de 2021). "Long-lasting analgesia via targeted in situ repression of NaV1.7 in mice". Science Translational Medicine (en inglés) 13 (584): eaay9056. ISSN 1946-6234. PMC 8830379. PMID 33692134. doi:10.1126/scitranslmed.aay9056.
- ↑ KAISER, JOCELYN (1 de xuño de 2022). "Better than CRISPR? Another way to fix gene problems may be safer and more versatile". www.science.org (en inglés). Consultado o 2022-08-21.
- ↑ PASTE = Programmable Addition via Site-specific Targeting Elements
- ↑ McDonnell S. "New CRISPR-based tool inserts large DNA sequences at desired sites in cells". Massachusetts Institute of Technology via phys.org (en inglés). Consultado o 18 de decembro de 2022.
- ↑ Yarnall MT, Ioannidi EI, Schmitt-Ulms C, Krajeski RN, Lim J, Villiger L, et al. (novembro de 2022). "Drag-and-drop genome insertion of large sequences without double-strand DNA cleavage using CRISPR-directed integrases". Nature Biotechnology 41 (4): 500–512. PMC 10257351. PMID 36424489. doi:10.1038/s41587-022-01527-4. Parámetro descoñecido
|biorxiv=ignorado (Axuda) - ↑ Anzalone AV, Randolph PB, Davis JR, Sousa AA, Koblan LW, Levy JM, et al. (decembro de 2019). "Search-and-replace genome editing without double-strand breaks or donor DNA". Nature 576 (7785): 149–157. Bibcode:2019Natur.576..149A. PMC 6907074. PMID 31634902. doi:10.1038/s41586-019-1711-4.
- ↑ Thulin L (21 de outubro de 2019). "A New Gene Editing Tool Could Make CRISPR More Precise.". The Smithsonian Magazine.
- ↑ 229,0 229,1 Cohen J (21 de outubro de 2019). "New 'prime' genome editor could surpass CRISPR.". Science Magazine.
- ↑ 230,0 230,1 Yasinski E (21 de outubro de 2019). "New "Prime Editing" Method Makes Only Single-Stranded DNA Cuts.". The Scientist.
- ↑ 231,0 231,1 Gallagher J (21 de outubro de 2019). "Prime editing: DNA tool could correct 89% of genetic defects". BBC News.
- ↑ Regalado A (5 de marzo de 2015). "Engineering the Perfect Baby". MIT Technology Review. Arquivado dende o orixinal o 13 de marzo de 2015.
- ↑ Baltimore D, Berg P, Botchan M, Carroll D, Charo RA, Church G, et al. (abril de 2015). "Biotechnology. A prudent path forward for genomic engineering and germline gene modification". Science 348 (6230): 36–38. Bibcode:2015Sci...348...36B. PMC 4394183. PMID 25791083. doi:10.1126/science.aab1028.
- ↑ Lanphier E, Urnov F, Haecker SE, Werner M, Smolenski J (marzo de 2015). "Don't edit the human germ line". Nature 519 (7544): 410–411. Bibcode:2015Natur.519..410L. PMID 25810189. doi:10.1038/519410a.
- ↑ Wade N (19 de marzo de 2015). "Scientists Seek Ban on Method of Editing the Human Genome". The New York Times. Consultado o 20 de marzo de 2015.
Os biólogos que escriben en Science apoian continuar a investigación de laboratorio con esta técnica e poucos ou ningún cren que estea lista para o uso clínico.
- ↑ Liang P, Xu Y, Zhang X, Ding C, Huang R, Zhang Z, et al. (maio de 2015). "CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes". Protein & Cell 6 (5): 363–372. PMC 4417674. PMID 25894090. doi:10.1007/s13238-015-0153-5.
- ↑ Kolata G (23 de abril de 2015). "Chinese Scientists Edit Genes of Human Embryos, Raising Concerns". The New York Times. Consultado o 24 de abril 2015.
- ↑ 238,0 238,1 Cyranoski D, Reardon S (2015). "Chinese scientists genetically modify human embryos". Nature. doi:10.1038/nature.2015.17378.
- ↑ Regalado A (2016-05-08). "Chinese Researchers Experiment with Making HIV-Proof Embryos". MIT Technology Review. Consultado o 2016-06-10.
- ↑ "International Summit on Gene Editing". National Academies of Sciences, Engineering, and Medicine. 3 de decembro de 2015. Consultado o 3 de decembro de 2015.
- ↑ Brokowski C (abril de 2018). "Do CRISPR Germline Ethics Statements Cut It?". The CRISPR Journal 1 (2): 115–125. PMC 6694771. PMID 31021208. doi:10.1089/crispr.2017.0024.
- ↑ Begley S (28 de novembro de 2018). "Amid uproar, Chinese scientist defends creating gene-edited babies". STAT.
- ↑ Sample I (13 de marzo de 2019). "Scientists call for global moratorium on gene editing of embryos". Theguardian.com. Consultado o 14 de marzo de 2019.
- ↑ Callaway E (febreiro de 2016). "UK scientists gain licence to edit genes in human embryos". Nature 530 (7588): 18. Bibcode:2016Natur.530...18C. PMID 26842037. doi:10.1038/nature.2016.19270.
- ↑ McHughen A, Smyth S (xaneiro de 2008). "US regulatory system for genetically modified [genetically modified organism (GMO), rDNA or transgenic] crop cultivars". Plant Biotechnology Journal 6 (1): 2–12. PMID 17956539. doi:10.1111/j.1467-7652.2007.00300.x.
- ↑ USDA. "Re: Request to confirm" (PDF). Arquivado dende o orixinal (PDF) o 11 de xuño de 2023. Consultado o 01 de novembro de 2023.
- ↑ Waltz E (abril de 2016). "Gene-edited CRISPR mushroom escapes US regulation". Nature 532 (7599): 293. Bibcode:2016Natur.532..293W. PMID 27111611. doi:10.1038/nature.2016.19754.
- ↑ Ledford H (abril de 2016). "Gene-editing surges as US rethinks regulations". Nature 532 (7598): 158–159. Bibcode:2016Natur.532..158L. PMID 27075074. doi:10.1038/532158a.
- ↑ Brown KV (1 de febreiro de 2017). "The FDA Is Cracking Down On Rogue Genetic Engineers". Gizmodo. Consultado o 5 de febreiro de 2017.
- ↑ "Guidance for Industry #187 / Regulation of Intentionally Altered Genomic DNA in Animals" (PDF). Food and Drug Administration. 2020-02-11.
- ↑ Cyranoski D (agosto de 2017). "China's embrace of embryo selection raises thorny questions". Nature 548 (7667): 272–274. Bibcode:2017Natur.548..272C. PMID 28816265. doi:10.1038/548272a.
- ↑ Peng Y (xuño de 2016). "The morality and ethics governing CRISPR-Cas9 patents in China". Nature Biotechnology 34 (6): 616–618. PMID 27281418. doi:10.1038/nbt.3590.
- ↑ Rana P, Marcus A, Fan W (2018-01-21). "China, Unhampered by Rules, Races Ahead in Gene-Editing Trials". Wall Street Journal. ISSN 0099-9660. Consultado o 2018-01-23.
- ↑ Talbot D (2016). "Precise Gene Editing in Plants/ 10 Breakthrough Technologies 2016". MIT Technology review (Massachusetts Institute of Technology). Arquivado dende o orixinal o 5 de novembro de 2017. Consultado o 18 de marzo de 2016.
- ↑ Larson C, Schaffer A (2014). "Genome Editing/ 10 Breakthrough Technologies 2014". Massachusetts Institute of Technology. Arquivado dende o orixinal o 5 de decembro de 2016. Consultado o 18 de marzo de 2016.
- ↑ "Tang Prize Laureates". www.tang-prize.org (en inglés). Consultado o 2018-08-05.
- ↑ "Press release: The Nobel Prize in Chemistry 2020". Nobel Foundation. Consultado o 7 de outubro de 2020.
