Bacteriófago


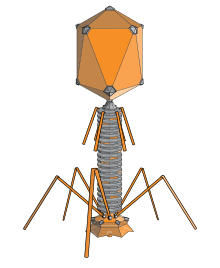



Un bacteriófago, abreviado informalmente como fago, é un virus que infecta e se replica dentro de bacterias e arqueas. O termo deriva de "bacteria" e do grego φαγεῖν (fagein), 'devorar'. Os bacteriófagos están compostos de proteínas que encapsulan o seu xenoma de ADN ou ARN, e poden ter estruturas simples ou moi elaboradas. Os seus xenomas poden codificar desde só catro xenes (caso do bacteriófago MS2) ata centos. Miden de 20 a 200 nm aproximadamente.[1] Os fagos replícanse dentro da célula procariota despois de inxectar o seu xenoma no seu citoplasma.
Os bacteriófagos están entre as entidades máis comúns e diversas da biosfera.[2] Son un tipo de virus que se encontra en todas os sitios en que existan bacterias. Estímase que hai máis de 1031 bacteriófagos no planeta, máis que calquera outro organismo da Terra, incluídas todas as bacterias combinadas.[3] Unha das fontes naturais máis densas de fagos e outros virus é a auga do mar, onde se atoparon ata 9·108 virións por mililitro en tapetes microbianos na superficie,[4] e ata un 70% das bacterias mariñas poden ser infectadas por fagos.[5]
Os fagos infectan só bacterias específicas. Algúns fagos son virulentos e destrúen inmediatamente a bacteria na que se reproducen, pero outros, chamados fagos temperados poden integrar o seu material xenético no ADN da bacteria e ser inocuos ou establecerse dentro da célula como se fosen plásmidos. Estes fagos endóxenos denomínanse profagos e replícanse xunto con todo o ADN cando a célula divide, polo que cada bacteria filla os leva. En certos momentos os fagos endóxenos actívanse e replícanse como virus virulentos destruíndo a célula. Nalgúns casos, os profagos poden ser mesmo beneficiosos para o hospedador, dándalle novas funcionalidades ao xenoma da bacteria, o que se chama conversión lisoxénica. Un exemplo é a bacteria Vibrio cholerae, que contén un fago, que codifica a toxina do cólera.[6]
Os fagos foron utilizados desde finais do século XX como alternativa aos antibióticos na antiga Unión Soviética e en Europa Central e tamén en Francia.[7][8] Considéranse unha posible terapia contra as cepas resistentes a multifármacos de moitas bacterias (terapia de fagos).[9] Por outra parte, os fagos do grupos dos Inoviridae observouse que complican as biopelículas implicadas na pneumonía e fibrose quística e protexen as bacterias dos fármacos que se usan para erradicar esas doenzas, promovendo así infeccións persistentes.[10]
Utilízanse tamén en investigación biolóxica en técnicas como o phage display, vectores de clonación e outras, e teñen aplicacións na industria alimentaria, farmacolóxica, diagnósticos clínicos e asepsia.
Clasificación[editar | editar a fonte]
Os bacteriófagos son abundantes na biosfera, teñen diferentes xenomas, morfoloxías e estilos de vida. Os fagos clasifícanse polo Comité Internacional de Taxonomía de Virus (ICTV) segundo a súa morfoloxía e ácido nucleico.

| Orde | Familia | Morfoloxía | Ácido nucleico | Exemplos |
|---|---|---|---|---|
| Belfryvirales | Turriviridae | Envolto, isométrico | ADN bicatenario linear | |
| Caudovirales | Ackermannviridae | Non envolto, cola contráctil | ADN bicatenario linear | |
| Myoviridae | Non envolto, cola contráctil | ADN bicatenario linear | T4, Mu, P1, P2 | |
| Siphoviridae | Non envolto, cola longa non contráctil | ADN bicatenario linear | λ, T5, HK97, N15 | |
| Podoviridae | Non envolto, cola curta non contráctil | ADN bicatenario linear | T7, T3, Φ29, P22 | |
| Halopanivirales | Sphaerolipoviridae | Envolto, isométrico | ADN bicatenario linear | |
| Haloruvirales | Pleolipoviridae | Envolto, pleomórfico | ADN monocatenario circular, ou ADN bicatenario linear | |
| Kalamavirales | Tectiviridae | Non envolto, isométrico | ADN bicatenario linear | |
| Levivirales | Leviviridae | Non envolto, isométrico | ARN monocatenario linear | MS2, Qβ |
| Ligamenvirales | Lipothrixviridae | Envolto, con forma de barra (=bacilar) | ADN bicatenario linear | Virus filamentoso de Acidianus 1 (Acidianus filamentous virus 1) |
| Rudiviridae | Non envolto, forma de barra | ADN bicatenario linear | Virus con forma de barra de Sulfolobus islandicus 1 (Sulfolobus islandicus rod-shaped virus 1) | |
| Mindivirales | Cystoviridae | Envolto, esférico | ARN bicatenario segmentado | |
| Petitvirales | Microviridae | Non envolto, isométrico | ADN monocatenario circular | ΦX174 |
| Tubulavirales | Inoviridae | Non envolto, filamentoso | ADN monocatenario circular | M13 |
| Vinavirales | Corticoviridae | Non envolto, isométrico | ADN bicatenario circular | PM2 |
| Non asignado | Ampullaviridae | Envolto, forma de botella | ADN bicatenario linear | |
| Bicaudaviridae | Non envolto, forma de limón | ADN bicatenario circular | ||
| Clavaviridae | Non envolto, forma de barra | ADN bicatenario circular | ||
| Finnlakeviridae | ADN bicatenario | FLiP[11] | ||
| Fuselloviridae | Non envolto, forma de limón | ADN bicatenario circular | ||
| Globuloviridae | Envolto, isométrico | ADN bicatenario linear | ||
| Guttaviridae | Non envolto, ovoide | ADN bicatenario circular | ||
| Plasmaviridae | Envolto, pleomórfico | ADN bicatenario circular | ||
| Portogloboviridae | Envolto, isométrico | ADN bicatenario circular | ||
| Spiraviridae | Non envolto, forma de barra | ADN monocatenario circular | ||
| Tristromaviridae | Envolto, forma de barra | ADN bicatenario linear |
Suxeriuse que os membros de Picobirnaviridae infectan bacterias, pero non mamíferos.[12]
Outra familia proposta é a "Autolykiviridae" (de ADN bicatenario).[13]
Historia[editar | editar a fonte]
En 1896, Ernest Hanbury Hankin informou que algo que estaba presente nas augas dos ríos Ganxes e Yamuna da India tiña unha marcada acción antibacteriana contra o axente do cólera e podía atravesar filtros de porcelana moi finos.[14] En 1915, o bacteriólogo británico Frederick Twort, superintendente da Brown Institution de Londres, descubriu un pequeno axente que infectaba e mataba bacterias. El pensaba que o axente debía ser algunha das seguintes cousas:
- Un estadio do ciclo biolóxico das bacterias.
- Un encima producido polas propias bacterias.
- Un virus que crecía nas bacterias e as destruía.[15]
As investigacións de Twort foron interompidas polo comezo da Primeira guerra mundial, e por unha escaseza de fondos e o descubrimento dos antibióticos.

Independentemente, o microbiólogo franco-canadense Félix d'Hérelle, que traballaba no Instituto Pasteur de París, anunciou o 3 de setembro de 1917, que descubrira "un microbio invisible antagonista do bacilo da disentería". Para d’Hérelle, non había dúbida sobre a natureza do descubrimento: "Nun instante o comprendera: o que causaba os meus puntos claros [nos cultivos bacterianos] era de feito un microbio invisible … un virus parasito das bacterias."[16] D'Hérelle chamou ao virus bacteriófago, un "comedor de bacterias" (do grego phagein, 'devorar'). Tamén informou do caso dun home que padecía disentería e que recuperou a saúde grazas aos bacteriófagos.[17] D'Herelle foi quen realizou boa parte das investigacións sobre bactgeriófagos daquela época e introduciu o concepto de terapia de fagos.[18]
Máis de medio século despois, en 1969, Max Delbrück, Alfred Hershey e Salvador Luria foron galardoados co Premio Nobel de Medicina polos seus descubrimentos da replicación de virus e a súa estrutura xenética.[19]
Evolución[editar | editar a fonte]
Estuos filoxenéticos indican que a maioría dos bacteriófagos que infectan actualmente os procariotas orixináronse antes que o último antepasado común universal (LUCA) e, por tanto, infectaron o último antepasado común universal e os protobiontes, (estruturas lipidícas compostas por material xenético que precederon ás células). A orixe dos bacteriófagos como primeiros virus é insegura, e suxeriuse que pudideron orixinarse no mundo de ARN ou a partir dalgún deses protobiontes ou por unha combinación de ambos.[20] No momento da orixe dos eucariotas os bacteriófagos precederían a varios virus que actualmente infectan os eucariotas. Por exemplo, os bacteriófagos con cola (Caudovirales) serían os devanceiros dos Herpesvirales, entre os que están os virus do herpes e a varicela.
Usos[editar | editar a fonte]
Terapia de fagos[editar | editar a fonte]
- Artigo principal: Terapia de fagos.
Como se descubrira que os fagos eran axentes antibacterianos, foron usados na antiga República Soviética de Xeorxia durante as décadas de 1920 e 1930 para tratar infeccións bacterianas. O pioneiro alí foi Giorgi Eliava coa axuda do codescubridor dos bacteriófagos Félix d'Herelle. Tiveron un amplo uso, incluíndo o tratamento de soldados do Exécito Vermello. Porén, foron despois abandonados para o seu uso xeral en Occidente por varias razóns:
- Descubríronse os antibióticos, que foron moi comercializados, eran máis fáciles de obter, almacenar e prescribir.
- Realízáronse ensaios médicos con fagos, pero a falta dunha comprensión básica suscitaba cuestións sobre a validez destes ensaios.[21]
- A publicación de investigacións na Unión Soviética facíase principalmente en lingua rusa ou xeorxiana e durante moitos anos non foi seguida internacionalmente.
O uso de fagos continuou desde o final da guerra fría en Rusia,[22] Xeorxia e noutros lugares de Europa central e do leste. O primeiro ensaio clínico regulado, aleatorizado de dobre cego publicouse no Journal of Wound Care en xuño de 2009, onde se avaliaba a seguridade e eficacia dun cóctel de bacteriófagos para tratar as úlceras venosas infectadas de pernas de pacientes humanos.[23] A FDA aprobou o estudo como un ensaio clínico en fase I. Os resultados do estudo demostraron a seguridade da aplicación terapéutica dos bacteriófagos, pero non mostraron ser eficaces. Os autores explicaron que o uso de certos compostos químicos que forman parte do coidado das feridas (por exemplo, a lactoferrina ou prata) puido interferir coa viabilidade dos bacteriófagos.[23] Pouco despois, publicouse outro ensaio controlado en Europa occidental (tratamento de infeccións de oído causadas por Pseudomonas aeruginosa) na revista Clinical Otolaryngology en agosto de 2009.[24] O estudo concluíu que as preparacións de bacteriófagos eran seguras e efectivas para o tratamento de infeccións crónicas de oído en humanos. Ademais, houbo ensaios clínicos en animais e doutro tipo que avaliaron a eficacia dos bacteriófagos contra varias doenzas, como as queimaduras infectadas e feridas, e a fibrose quística asociada con infeccións de pulmón, entre outras.[24]
Mentres tanto, os investigadores no eido dos bacteriófagos desenvolveran virus transformados por enxeñaría para superar a resistencia a antibióticos, e prepararon por enxeñería xenes de fagos que codificaban encimas que degradaban a matriz das biopelículas, as proteínas estruturais de fagos e os encimas responsables da lise da parede celular bacteriana.[4][5][7] Algúns resultados mostraron que os fagos T4 que son de pequeno tamaño e cola curta, poden ser unha boa axuda para detectar a bacteria Escherichia coli no corpo humano.[25]
Avaliouse a eficacia terapéutica dun cóctel de fagos nun modelo de ratos coa infección nasal pola bacteria resistente a multidrogas Acinetobacter baumannii. Os ratos tratados co cóctel de fagos mostraron unha taxa de supervivencia 2,3 veces maior que os non tratados aos sete días despois da infección.[26] En 2017 un paciente co páncreas afectado por A. baumannii resistente a multidrogas recibiu tratamento con varios antibióticos; malia iso a saúde do paciente continuou deteriorándose durante un período de catro meses. Xa sen administración de antibióticos o paciente foi sometido a terapia de fagos usando un cóctel de nove fagos que demostraran ser efctivos ante A. baumannii resistente a multidrogas, e o paciente mellorou ata recuperar a saúde.[27]
D'Herelle "aprendeu rapidamente que os bacteriófagos se encontran onde queira que prosperen as bacterias: en cloacas, en ríos que reciben escorreduras residuais de tubaxes e nas feces dos pacientes convalecentes."[28] Isto inclúe ríos que tradicionalmente se cría que tiñan poderes curativos, como o río Ganxes da India (pero que hoxe en día está moi contaminado).[29]
Outros[editar | editar a fonte]
Industria alimentaria.– Desde 2006,a Administración de Medicinas e Alimentos (FDA) e o Departamento de Agricultura dos Estados Unidos (USDA) aprobaron varios produtos bacteriófagos. O LMP-102 (Intralytix) aprobouse para tratar produtos de aves e outros produtos cárnicos "listos para comer". Ese mesmo ano a FDA aprobou LISTEX (desenvolvido e producido por Micreos) para usar bacteriófagos co quiexo para matar a bacteria Listeria monocytogenes e deulle a consideración de produto xeralmene recoñecido como seguro.[30] En xullo de 2007, o mesmo bacteriófago foi aprobado para o seu uso en todos os produtos alimenticios.[31] En 2011 o USDA confirmou que LISTEX é unha axuda ao procesamento de etiqueta limpa e está incluído na lista da USDA.[32] Os investigadores no campo da seguridade alimentaria seguen examinando se os fagos líticos son unha opción viable para controlar outros patóxenos que se poden encontrar nos alimentos.
Industria láctea.– Os bacteriófagos presentes no ambiente poden causar fallos na fermentación dos cultivos iniciadores na produción de queixo. Para evitalo, poden usarse cultivos iniciadores de cepas mesturadas e réximes de rotación destes cultivos.[33]
Diagnósticos.– En 2011, a FDA autorizou o primeiro produto baseado en bacteriófagos para o uso en diagnósticos in vitro.[34] O KeyPath MRSA/MSSA Blood Culture Test utiliza un cóctel de bacteriófagos para detectar Staphylococcus aureus en cultivos sanguíneos positivos e determinar a súa resistencia ou susceptibilidade á meticilina. O test dá os resultados nunhas cinco horas, comparadas cos dous ou tres días dos métodos dos test estándar de identificación e susceptibilidade microbiana. Foi o primeiro test de susceptibilidade a antibióticos acelerado aprobado pola FDA.[35]
Contrarrestación de armas biolóxicas e toxinas.– As axencias gobernamentais de países occidentais estiveron durante varios anos pedindo axuda a Xeorxia e á antiga Unión Soviética para aproveitar os fagos para contrarrestar as armas biolóxicas e toxinas, como o ántrax e o botulismo.[36] Continúan facéndose desenvolvementos en grupos de investigación nos EUA. Outros usos inclúen a aplicación en spray en horticultura para protexer as plantas e produtos vexetais da podremia e do espallamento de enfermidades bacterianas. Outras aplicacións para os bacteriófagos son os biocidas para superficies de cdertos ambientes, por exemplo en hospitais, e como tratamento preventivo para catéteres e aparellos médicos antes do seu uso en instalacións médicas. Existe actualmetne unha tecnoloxía dos fagos que pode ser aplicada a superficies secas, por exemplo, uniformes, cortinas ou mesmo suturas cirúrxicas. Os ensaios clínicos publicados en Clinical Otolaryngology[24] indican un éxito no tratamento veterinario de cans con otite.
SEPTIC.- O método de detección e identificación bacteriana SEPTIC (Sensing of phage-triggered ion cascades, Detección de fervenzas iónicas desencadeadas por fagos) usa a emisión de ións e as súas dinámicas durante a infección de fagos e ofrece unha alta especificidade para a detección.[37]
Phage display.- Na técnica do phage display faise un uso diferente dos fagos en investigación, que implica a creación dunha biblioteca de fagos cun péptido variable unido a unha proteína superficial. Cada xenoma dos fagos codifica a variante da proteína exposta na súa superficie, proporcionando un vínculo entre a variante do péptido e o xene que o codifica. As variantes dos fagos na biblioteca poden ser seleccionadas pola súa afinidade de unión a unha molécula inmobilizada (por exemplo, a toxina botúlica) para neutralizala. Os fagos seleccionados unidos poden multiplicarse reinfectando unha cepa bacteriana susceptible, o que permite recuperar os péptidos codificados neles para posteriores estudos.[38]
Descubrimento de fármacos antimicrobianos – As proteínas dos fagos adoitan ter actividade antimicrobiana e poden servir para a peptidomimética, é dicir, fármacos que imitan os péptidos.[39] A tecnoloxía fago-ligando utiliza as proteínas dos fagos en varias aplicacións, como unirse a bacterias ou compoñentes bacterianos (por exemplo a endotoxinas) e a lise das bacterias.[40]
Vectores de clonación.- Os fagos teñen un importante papel na bioloxía molecular como vectores de clonación para inserir ADN en bacterias. Combinando material xenético de fagos con plásmidos orixínanse un tipo de plásmidos artificiais denominados faxémidos (ou fásmidos), usados como vectores.
Investigación básica.– Os bacteriófagos son organismos modelo importantes para estudar os principios da evolución e a ecoloxía.[41]
Replicación[editar | editar a fonte]

Os bacteriófagos poden ter un ciclo lítico ou un ciclo lisoxénico. Con fagos líticos como o fago T4, as células bacterianas son rotas e destruídas (lisadas) despois da replicación dovirión no seu interior, que dese modo libera a proxenie, que busca novos hóspedes que infectar. Os fagos líticos son máis axeitados para a terapia de fagos. Algúns fagos líticos experimentan un fenómeno chamado inhibición da lise, na que a proxenie do fago xa completada non lisa inmediatemente a célula se as concentracións extracelular de fagos son altas. Este mecanismo non é igual que o dos fagos temperados, que permanecen dormentes integrados no ADN da célula, e xeralmente é temporal.
A diferenza do anterior, o ciclo lisoxénico non orixina unha lise inmediata da célula hóspede. Os fagos que poden realizar a lisoxenia denomínanse fagos temperados. O seu xenoma viral intégrase no ADN do hóspede e replícase xunto con el, de maneira relativamente inofensiva para a célula, ou pode establecerse como un plásmido intracelular. Deste modo as bacterias fillas levan todas o virus. O virus permanece doemente ata que as condicións do hóspede se deterioran, quizais por unha escaseza de nutrientes, e entón, os fagos endoxenos (chamados profagos) fanse activos. Nese momento inician un ciclo reprodutivo, que ten como resultado a lise da célula hóspede. Como o ciclo lisoxénico permite á célula hóspede continuar sobrevivindo e reproducíndose, o virus replícase en todas as células fillas da célula hóspede orixinal. Un exemplo de bacteriófago que segue un ciclo lisoxénico e un ciclo lítico é o fago lambda de Escherichia coli.[42]
Ás veces os profagos poden ser beneficiosos para a bacteria hóspede porque están dormentes e á vez proporcionan novas funcións ao xenoma bacteriano, fenómeno denominado conversión lisoxénica. Exemplos son a conversión de cepas inofensivas de Corynebacterium diphtheriae ou Vibrio cholerae por acción dos bacteriófagos, noutras altamente virulentas que causan a difteria ou o cólera, respectivamente.[43][44] Propuxéronse estratexias para combater certas infeccións bacterianas tomando como diana os profagos que codifican toxinas.[45]
Adhesión e penetración[editar | editar a fonte]
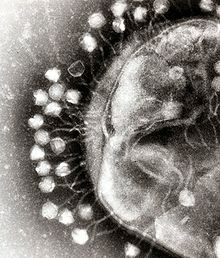
As células bacterianas están protexidas por unha parede celular de polisacáridos, que son importantes factores de virulencia que protexen as células bacterianas contra as defensas inmunitarias do hóspede e algúns antibióticos.[46] Para entraren na célula hóspede, os bacteriófagos únense a receptores específicos situados na superficie da bacteria, como os lipopolisacáridos, ácidos teicoicos, proteínas ou mesmo flaxelos. Esta especificidade significa que un bacteriófago só pode infectar certas bacterias que leven os receptores axeitados aos cales se pode unir, o cal á súa vez determina o rango de hóspedes do fago. Os encimas que degradan os polisacáridos, como as endolisinas, son proteínas asociadas a virións que degradan encimaticamente a capa externa capsular do hóspede, na etapa inicial do proceso estritamente programado de infección do fago. As condicións de crecemento do hóspede tamén inflúen na capacidade do fago de adherirse a el e invadilo.[47] Como os virións de fagos non se moven independentemente, dependen de encontros casuais cos receptores correctos cando están en solución, como no sangue, linfa, augas de rego, auga do solo, mar etc.
Os bacteriófagos miovirus usan un mecanismo de tipo xiringa hipodérmica para inxectar o seu material xenético dentro da célula. Unha vez que contactan co receptor apropiado, as fibras da cola flexiónanse achegando a placa basal moi preto da superficie da célula, o cal se denomina unión reversible. Unha vez que se adheriu completamente, iníciase a unión irreversible e a cola contráese, posiblemente coa axuda da enerxía do ATP, presente na cola,[5] inxectando o material xenético a través da membrana bacteriana.[48] A inxección realízase por medio dunha especie de movemento de dobramento no talo da cola, indo primeiro cara a un lado, contraéndose achegándose máis á célula e empuxando para arriba. Os podovirus carecen dunha vaíña da cola alongada como a dos miovirus, polo que no canto de faceren iso, usan as fibras da súa pequena cola con forma de dente para que degraden encimaticamente unha porción da membrana celular antes de inseriren o seu material xenético.
Síntese de proteínas e ácidos nucleicos[editar | editar a fonte]
En cuestión de minutos, os ribosomas bacterianos empezan a traducir o ARNm viral a proteínas. Nos fagos de ARN, sintetízase temperanmente durante o proceso unha ARN replicase. As proteínas modifican a ARN polimerase bacteriana para que transcriba preferencialmente o ARNm viral. A síntese de proteínas normal do hóspede e a de ácidos nucleicos queda alterada, e a célula é forzada a fabricar os produtos virais. Estes produtos acabarán converténdose en partes dos novos virións que se forman dentro da célula, en proteínas axudantes que contribúen á ensamblaxe dos novos virións ou en proteínas implicadas na lise da célula. En 1972, Walter Fiers (Universidade de Gante, Bélxica) foi o primeiro en establecer a secuencia nucleotídica completa dun xene de fago e, en 1976, do xenoma viral completo do bacteriófago MS2.[49] Algúns bacteriófagos de ADN bicatenario codifican proteínas ribosómicas, que se pensan que modulan a tradución de proteínas durante a infección do fago.[50]
Ensamblaxe do virión[editar | editar a fonte]
No caso do fago T4, a construción de novas partículas víricas implica a asistencia de proteínas axudantes. As placas basais son as que primeiro se ensamblan, e as colas constrúense despois sobre elas. As cápsides das cabezas, que se constrúen por separado, ensámblanse despois espontaneamente coas colas. O ADN empaquétase eficientemente dentro das cabezas. O proceso completo tarda uns 15 minutos.
Liberación de virións[editar | editar a fonte]
Os fagos poden liberarse pola lise da célula, pero tamén por extrusión e, nuns poucos casos, por evaxinación. A lise dos fagos con cola realízase pola acción do encima endolisina, que ataca e destrúe a parede celular de peptidoglicano. Un tipo de fago completamente diferente, o fago filamentoso, fai que a célula hóspede segregue continuamente novas partículas víricas. Os virións liberados descríbense como libres e, a non ser que sexan defectuosos, con capacidade de infectar novas bacterias. A evaxinación está asociada con certos fagos de Mycoplasma (unha bacteria que, excepcionalmente, non ten parede). A diferenza da liberación dos virións, os fagos que presentan un ciclo lisoxénico non matan a célla hóspede, senón que se converten en residentes a longo prazo en forma de profagos.
Comunicación[editar | editar a fonte]
Investigacións feitas en 2017 revelaron que o bacteriófago Φ3T fabrica unha proteína viral curta que sinaliza a outros bacteriófagos que queden durmentes en vez de mataren a bacteria hóspede. A esta proteína déuselle onome de arbitrium.[51][52]
Estrutura xenómica[editar | editar a fonte]
Hai millóns de fagos no medio ambiente e os seus xenomas poden ter diversas formas e tamaños. Os fagos de ARN, como o MS2 teñen os xenomas máis pequenos, de só unhas poucas quilobases. Porén, algúns fagos de ADN como o fago T4 poden ter xenomas grandes, con centros de xenes; o tamaño e forma da cápside varía en consonancia co xenoma.[53] Os xenomas de bacteriófagos máis grandes chegan a tamaños de 735 kb.[54]
Os xenomas de bacteriofagos poden ter un elevado mosaicismo, é dicir, o xenoma de moitas especies de fagos parece estar composto por numerosos módulos individuais. Estes módulos poden encontrarse noutras especies de fagos en diferentes arranxos. Os micobacteriófagos, que son bacteriófagos con hóspedes micobacterianos, proporcionaron excelentes exemplos deste mosaicismo. Nestes micobacteriófagos, a segregación xenética pode ser o resultado de repetidos casos de recombinación específica de sitio e recombinación ilexítima (o resultado da adquisición polo fago de secuencias xenéticas do hóspede bacteriano).[55] Os mecanismos evolutivos que deron forma aos xenomas dos virus bacterianos varían entre diferentes familias e dependen do tipo de ácido nucleico, características da estrutura do virión, así como do tipo de ciclo vital do virión.[56]
Bioloxía de sistemas[editar | editar a fonte]
Os fagos adoitan ter drásticos efectos sobre os seus hóspedes. Como consecuencia, o padrón de transcrición da bacteria infectada pode cambiar considerablemente. Por exemplo, a infección que sofre Pseudomonas aeruginosa polo fago temperado PaP3 cambia a expresión do 38% (2160/5633) dos xenes do hóspede. Moitos destes efectos son probablemente indirectos, polo que é un reto identificar as interaccións indirectas entre a bacteria e o fago.[57]
Fixéronse varios intentos de mapar as interaccións proteína-proteína entre os fagos e o seu hóspede. Por exemplo, o bacteriófago lambda sabíase que interaccionaba co seu hóspede, E. coli, por medio de 31 interaccións. Porén, un estudo a grande escala revelou 62 interaccións, a maioría das cales eran novas. De novo, a impotancia de moitas destas interaccións segue sendo pouco clara, pero estes estudos suxiren que hai moi probablemente varias interaccións clave e moitas interaccións indirectas cuxo papel permanece sen determinar.[58]
No medio ambiente[editar | editar a fonte]
- Artigo principal: Bacteriófagos mariños.
A metaxenómica permitiu a detección na auga de bacteriófagos, o que non era posible previamente.[59]
Ademais, os bacteriófagos foron utilizados en rastreos hidrolóxicos e modelización de sistemas fluviais, especialmente onde hai interaccións entre as augas superficiais e as subterráneas. Prefírese o uso de fagos ao dun marcador de tinguidura máis convencional porque son absorbidos menos significativamente cando pasan a través de augas subterráneas e son doadamente detectados a concentracións moi baixas.[60] As augas non polucionadas poden conter aproximadamente 2·108 bacteriófagos por mL.[61]
Pénsase que os bacteriófagos contribúen amplamente á transferencia horizontal de xenes en ambientes naturais, principalmente por transdución, pero tamén por transformación.[62] Os estudos baseados na metaxenmica tamén revelaron que os viromas dunha variedade de ambientes albergan xenes de resistencia a antibióticos, incluíndo os que poden dar resistencia a multifármacos.[63]
No mar os bacteriófagos son as entidades biolóxicas máis abundante e parasitan as bacterias mariñas, principalmente cianobacterias,[64] [65] chegando a niveis de 250 millóns de bacteriófagos por mililitro de auga de mar.[66] As bacterias deféndense producindo encimas (endonucleases de restrición) que destrúen o ADN dos bacteriófagos[67] ou por unha interferencia de ARN que utiliza o sistema CRISPR,[68][69] o cal lles dá certa inmunidade adquirida á infección.[70] As bacterias mariñas son fundamentais nos ciclos da materia nos ecosistemas da Terra,[71] e os bacteriófagos mariños modulan estes ciclos no mar ao afectaren á poboación de bacterias.[72][73] Por exemplo, inflúen no ciclo do carbono ao controlaren as poboacións de cianobacterias como Prochlorococcus, que é un importante produtor primario.[74] Ademais, os fagos mariños inflúen no metabolismo e evolución dos seus hóspedes, contribuíndo o intercambio e incorporación de xenes.[73]
Bacteriófagos modelo[editar | editar a fonte]
Os seguintes bacteriófagos foron amplamente estudados:
Notas[editar | editar a fonte]
- ↑ Stephen., Mc Grath,; van., Sinderen, Douwe (2007). Caister Academic Press, ed. Bacteriophage : genetics and molecular biology. ISBN 9781904455141. OCLC 86168751.
- ↑ 2,0 2,1 McGrath S and van Sinderen D (editors). (2007). Bacteriophage: Genetics and Molecular Biology (1st ed.). Caister Academic Press. ISBN 978-1-904455-14-1.
- ↑ "Novel Phage Therapy Saves Patient with Multidrug-Resistant Bacterial Infection". UC Health – UC San Diego. Consultado o 13 May 2018.
- ↑ 4,0 4,1 Wommack, K. E.; Colwell, R. R. (2000). "Virioplankton: Viruses in Aquatic Ecosystems". Microbiology and Molecular Biology Reviews 64 (1): 69–114. PMC 98987. PMID 10704475. doi:10.1128/MMBR.64.1.69-114.2000.
- ↑ 5,0 5,1 5,2 Prescott, L. (1993). Microbiology, Wm. C. Brown Publishers, ISBN 0-697-01372-3
- ↑ McLeod, S. M.; Kimsey, H. H.; Davis, B. M.; Waldor, M. K. (2005). "CTXφ and Vibrio cholerae: exploring a newly recognized type of phage-host cell relationship". Molecular Microbiology 57: 347–356. PMID 15978069. doi:10.1111/j.1365-2958.04676.x.
- ↑ 7,0 7,1 BBC Horizon (1997): The Virus that Cures – Documentary about the history of phage medicine in Russia and the West
- ↑ Borrell, Brendan (August 2012). "Science talk: Phage factor". Scientific American: 80–83.
- ↑ Keen, E. C. (2012). "Phage Therapy: Concept to Cure". Frontiers in Microbiology 3: 238. PMC 3400130. PMID 22833738. doi:10.3389/fmicb.2012.00238.
- ↑ "Bacteria and bacteriophages collude in the formation of clinically frustrating biofilms".
- ↑ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas, Ashley Gillum, David I. Stuart, Janne J. Ravantti, Juha Huiskonen, Lotta-Riina Sundberg: Virus found in a boreal lake links ssDNA and dsDNA viruses. In: Proceedings of the National Academy of Sciences 114(31), July 2017, doi:10.1073/pnas.1703834114
- ↑ Krishnamurthy SR, Wang D (2018). "Extensive conservation of prokaryotic ribosomal binding sites in known and novel picobirnaviruses". Virology 516: 108–114. PMID 29346073. doi:10.1016/j.virol.2018.01.006.
- ↑ Kathryn M. Kauffman, Fatima A. Hussain, Joy Yang, Philip Arevalo, Julia M. Brown, William K. Chang, David VanInsberghe, Joseph Elsherbini, Radhey S. Sharma, Michael B. Cutler, Libusha Kelly, Martin F. Polz: A major lineage of non-tailed dsDNA viruses as unrecognized killers of marine bacteria. In: Nature Vol. 554, pp. 118–122. January 24th, 2018. doi:10.1038/nature25474
- ↑ Hankin E H. (1896). "L'action bactericide des eaux de la Jumna et du Gange sur le vibrion du cholera". Annales de l'Institut Pasteur (en French) 10: 511–523.
- ↑ Twort, F. W. (1915). "An Investigation on the Nature of Ultra-Microscopic Viruses". The Lancet 186 (4814): 1241–1243. doi:10.1016/S0140-6736(01)20383-3.
- ↑ d'Hérelles, Félix (1917). "Sur un microbe invisible antagoniste des bacilles dysentériques" (PDF). Comptes Rendus de l'Académie des Sciences de Paris 165: 373–5. Arquivado dende o orixinal (PDF) o 11 May 2011. Consultado o 5 September 2010.
- ↑ d'Hérelles, Félix (1949). "The bacteriophage" (PDF). Science News 14: 44–59. Consultado o 5 September 2010.
- ↑ Keen EC (2012). "Felix d'Herelle and Our Microbial Future". Future Microbiology 7 (12): 1337–1339. PMID 23231482. doi:10.2217/fmb.12.115.
- ↑ "The Nobel Prize in Physiology or Medicine 1969". Nobel Foundation. Consultado o 28 July 2007.
- ↑ Mart Krupovic, Valerian V. Dolja, Eugene V. Koonin (2020). The LUCA and its complex virome. Nature.
- ↑ Kutter, Elizabeth; De Vos, Daniel; Gvasalia, Guram; Alavidze, Zemphira; Gogokhia, Lasha; Kuhl, Sarah; Abedon, Stephen (1 January 2010). "Phage Therapy in Clinical Practice: Treatment of Human Infections". Current Pharmaceutical Biotechnology 11 (1): 69–86. PMID 20214609. doi:10.2174/138920110790725401.
- ↑ Сергей Головин Бактериофаги: убийцы в роли спасителей // Наука и жизнь. – 2017. – № 6. – С. 26–33
- ↑ 23,0 23,1 Rhoads, DD; Wolcott, RD; Kuskowski, MA; Wolcott, BM; Ward, LS; Sulakvelidze, A (June 2009). "Bacteriophage therapy of venous leg ulcers in humans: results of a phase I safety trial". Journal of Wound Care 18 (6): 237–8, 240–3. PMID 19661847. doi:10.12968/jowc.2009.18.6.42801.
- ↑ 24,0 24,1 24,2 Wright, A.; Hawkins, C.H.; Änggård, E.E.; Harper, D.R. (August 2009). "A controlled clinical trial of a therapeutic bacteriophage preparation in chronic otitis due to antibiotic-resistant Pseudomonas aeruginosa; a preliminary report of efficacy". Clinical Otolaryngology 34 (4): 349–357. PMID 19673983. doi:10.1111/j.1749-4486.2009.01973.x.
- ↑ Tawil, Nancy (April 2012). "Surface plasmon resonance detection of E. coli and methicillin-resistant S. aureus using bacteriophages" (PDF). Biosensors and Bioelectronics 37 (1): 24–29. PMID 22609555. doi:10.1016/j.bios.2012.04.048.
- ↑ Cha, Kyoungeun; Oh, Hynu K.; Jang, Jae Y.; Jo, Yunyeol; Kim, Won K.; Ha, Geon U.; Ko, Kwan S.; Myung, Heejoon (10 April 2018). "Characterization of Two Novel Bacteriophages Infecting Multidrug-Resistant (MDR) Acinetobacter baumannii and Evaluation of Their Therapeutic Efficacy in Vivo". Frontiers in Microbiology 9: 696. ISSN 1664-302X. PMC 5932359. PMID 29755420. doi:10.3389/fmicb.2018.00696.
- ↑ Schooley, Robert T.; Biswas, Biswajit; Gill, Jason J.; Hernandez-Morales, Adriana; Lancaster, Jacob; Lessor, Lauren; Barr, Jeremy J.; Reed, Sharon L.; Rohwer, Forest (October 2017). "Development and Use of Personalized Bacteriophage-Based Therapeutic Cocktails To Treat a Patient with a Disseminated Resistant Acinetobacter baumannii Infection". Antimicrobial Agents and Chemotherapy 61 (10). ISSN 0066-4804. PMC 5610518. PMID 28807909. doi:10.1128/AAC.00954-17.
- ↑ Kuchment, Anna (2012). The Forgotten Cure: The past and future of phage therapy. Springer. p. 11. ISBN 978-1-4614-0250-3.
- ↑ Deresinski, Stan (15 April 2009). "Bacteriophage Therapy: Exploiting Smaller Fleas" (PDF). Clinical Infectious Diseases 48 (8): 1096–1101. PMID 19275495. doi:10.1086/597405.
- ↑ U.S. FDA/CFSAN: Agency Response Letter, GRAS Notice No. 000198
- ↑ (U.S. FDA/CFSAN: Agency Response Letter, GRAS Notice No. 000218)
- ↑ "FSIS Directive 7120" (PDF). Arquivado dende o orixinal (PDF) o 18 de outubro de 2011. Consultado o 11 de setembro de 2020.
- ↑ Atamer, Zeynep; Samtlebe, Meike; Neve, Horst; J. Heller, Knut; Hinrichs, Joerg (16 July 2013). "Review: elimination of bacteriophages in whey and whey products". Frontiers in Microbiology 4: 191. PMC 3712493. PMID 23882262. doi:10.3389/fmicb.2013.00191.
- ↑ FDA 510(k) Premarket Notification
- ↑ FDA clears first test to quickly diagnose and distinguish MRSA and MSSA. FDA (6 May 2011)
- ↑ Vaisman, Daria (25 May 2007) Studying anthrax in a Soviet-era lab – with Western funding. The New York Times
- ↑ Dobozi-King, M.; Seo, S.; Kim, J.U.; Young, R.; Cheng, M.; Kish, L.B. (2005). "Rapid detection and identification of bacteria: SEnsing of Phage-Triggered Ion Cascade (SEPTIC)" (PDF). Journal of Biological Physics and Chemistry 5: 3–7. doi:10.4024/1050501.jbpc.05.01. Arquivado dende o orixinal (PDF) o 26 de setembro de 2018. Consultado o 11 de setembro de 2020.
- ↑ Smith GP, Petrenko VA (April 1997). "Phage Display". Chem. Rev. 97 (2): 391–410. PMID 11848876. doi:10.1021/cr960065d.
- ↑ Liu, Jing; Dehbi, Mohammed; Moeck, Greg; Arhin, Francis; Bauda, Pascale; Bergeron, Dominique; Callejo, Mario; Ferretti, Vincent; Ha, Nhuan (February 2004). "Antimicrobial drug discovery through bacteriophage genomics". Nature Biotechnology 22 (2): 185–191. PMID 14716317. doi:10.1038/nbt932.
- ↑ Technological background Phage-ligand technology
- ↑ Keen, E. C. (2014). "Tradeoffs in bacteriophage life histories". Bacteriophage 4 (1): e28365. PMC 3942329. PMID 24616839. doi:10.4161/bact.28365.
- ↑ Mason, Kenneth A., Jonathan B. Losos, Susan R. Singer, Peter H Raven, and George B. Johnson. (2011). Biology, p. 533. McGraw-Hill, New York. ISBN 978-0-07-893649-4.
- ↑ Mokrousov I (2009). "Corynebacterium diphtheriae: genome diversity, population structure and genotyping perspectives". Infection, Genetics and Evolution 9 (1): 1–15. PMID 19007916. doi:10.1016/j.meegid.2008.09.011.
- ↑ Charles RC, Ryan ET (October 2011). "Cholera in the 21st century". Current Opinion in Infectious Diseases 24 (5): 472–7. PMID 21799407. doi:10.1097/QCO.0b013e32834a88af.
- ↑ Keen, E. C. (December 2012). "Paradigms of pathogenesis: Targeting the mobile genetic elements of disease". Frontiers in Cellular and Infection Microbiology 2: 161. PMC 3522046. PMID 23248780. doi:10.3389/fcimb.2012.00161.
- ↑ Drulis-Kawa, Zuzanna; Majkowska-Skrobek, Grazyna; MacIejewska, Barbara (2015). "Bacteriophages and Phage-Derived Proteins – Application Approaches". Current Medicinal Chemistry 22 (14): 1757–1773. PMC 4468916. PMID 25666799. doi:10.2174/0929867322666150209152851.
- ↑ Gabashvili, I.; Khan, S.; Hayes, S.; Serwer, P. (1997). "Polymorphism of bacteriophage T7". Journal of Molecular Biology 273 (3): 658–67. PMID 9356254. doi:10.1006/jmbi.1997.1353.
- ↑ Maghsoodi, A.; Chatterjee, A.; Andricioaei, I.; Perkins, N.C. (2019-11-25). "How the phage T4 injection machinery works including energetics, forces, and dynamic pathway". Proceedings of the National Academy of Sciences 116 (50): 25097–25105. ISSN 0027-8424. PMC 6911207. PMID 31767752. doi:10.1073/pnas.1909298116.
- ↑ Fiers, W.; Contreras, R.; Duerinck, F.; Haegeman, G.; Iserentant, D.; Merregaert, J.; Min Jou, W.; Molemans, F.; Raeymaekers, A.; Van Den Berghe, A.; Volckaert, G.; Ysebaert, M. (1976). "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene". Nature 260 (5551): 500–507. Bibcode:1976Natur.260..500F. PMID 1264203. doi:10.1038/260500a0.
- ↑ Mizuno, CM; Guyomar, C; Roux, S; Lavigne, R; Rodriguez-Valera, F; Sullivan, MB; Gillet, R; Forterre, P; Krupovic, M (2019). "Numerous cultivated and uncultivated viruses encode ribosomal proteins.". Nature Communications 10 (1): 752. Bibcode:2019NatCo..10..752M. PMC 6375957. PMID 30765709. doi:10.1038/s41467-019-08672-6.
- ↑ Callaway, Ewen (2017). "Do you speak virus? Phages caught sending chemical messages". Nature. doi:10.1038/nature.2017.21313.
- ↑ Erez, Zohar; Steinberger-Levy, Ida; Shamir, Maya; Doron, Shany; Stokar-Avihail, Avigail; Peleg, Yoav; Melamed, Sarah; Leavitt, Azita; Savidor, Alon; Albeck, Shira; Amitai, Gil; Sorek, Rotem (26 January 2017). "Communication between viruses guides lysis–lysogeny decisions". Nature 541 (7638): 488–493. Bibcode:2017Natur.541..488E. ISSN 0028-0836. PMC 5378303. PMID 28099413. doi:10.1038/nature21049.
- ↑ Black, LW; Thomas, JA (2012). Condensed genome structure. Advances in Experimental Medicine and Biology 726. pp. 469–87. ISBN 978-1-4614-0979-3. PMC 3559133. PMID 22297527. doi:10.1007/978-1-4614-0980-9_21.
- ↑ Al-Shayeb, Basem; Sachdeva, Rohan; Chen, Lin-Xing; Ward, Fred; Munk, Patrick; Devoto, Audra; Castelle, Cindy J.; Olm, Matthew R.; Bouma-Gregson, Keith; Amano, Yuki; He, Christine (February 2020). "Clades of huge phages from across Earth's ecosystems". Nature (en inglés) 578 (7795): 425–431. ISSN 1476-4687. PMC 7162821. PMID 32051592. doi:10.1038/s41586-020-2007-4.
- ↑ Morris P, Marinelli LJ, Jacobs-Sera D, Hendrix RW, Hatfull GF (March 2008). "Genomic characterization of mycobacteriophage Giles: evidence for phage acquisition of host DNA by illegitimate recombination". Journal of Bacteriology 190 (6): 2172–82. PMC 2258872. PMID 18178732. doi:10.1128/JB.01657-07.
- ↑ Krupovic M, Prangishvili D, Hendrix RW, Bamford DH (December 2011). "Genomics of bacterial and archaeal viruses: dynamics within the prokaryotic virosphere". Microbiology and Molecular Biology Reviews 75 (4): 610–35. PMC 3232739. PMID 22126996. doi:10.1128/MMBR.00011-11.
- ↑ Zhao X, Chen C, Shen W, Huang G, Le S, Lu S, Li M, Zhao Y, Wang J, Rao X, Li G, Shen M, Guo K, Yang Y, Tan Y, Hu F (2016). "Global Transcriptomic Analysis of Interactions between Pseudomonas aeruginosa and Bacteriophage PaP3". Sci Rep 6: 19237. Bibcode:2016NatSR...619237Z. PMC 4707531. PMID 26750429. doi:10.1038/srep19237.
- ↑ Blasche S, Wuchty S, Rajagopala SV, Uetz P (2013). "The protein interaction network of bacteriophage lambda with its host, Escherichia coli". J. Virol. 87 (23): 12745–55. PMC 3838138. PMID 24049175. doi:10.1128/JVI.02495-13.
- ↑ Breitbart M, Salamon P, Andresen B, Mahaffy JM, Segall AM, Mead D, Azam F, Rohwer F (October 2002). "Genomic analysis of uncultured marine viral communities". Proc. Natl. Acad. Sci. U.S.A. 99 (22): 14250–5. Bibcode:2002PNAS...9914250B. PMC 137870. PMID 12384570. doi:10.1073/pnas.202488399.
- ↑ Martin, C. (1988). "The Application of Bacteriophage Tracer Techniques in South West Water". Water and Environment Journal 2 (6): 638–642. doi:10.1111/j.1747-6593.1988.tb01352.x.
- ↑ Bergh, O (1989). "High abundance of viruses found in aquatic environments". Nature 340 (6233): 467–468. Bibcode:1989Natur.340..467B. PMID 2755508. doi:10.1038/340467a0.
- ↑ Keen, Eric C.; Bliskovsky, Valery V.; Malagon, Francisco; Baker, James D.; Prince, Jeffrey S.; Klaus, James S.; Adhya, Sankar L.; Groisman, Eduardo A. (2017). "Novel "Superspreader" Bacteriophages Promote Horizontal Gene Transfer by Transformation". mBio 8 (1): e02115–16. PMC 5241400. PMID 28096488. doi:10.1128/mBio.02115-16.
- ↑ Lekunberri, Itziar; Subirats, Jessica; Borrego, Carles M.; Balcazar, Jose L. (2017). "Exploring the contribution of bacteriophages to antibiotic resistance". Environmental Pollution 220 (Pt B): 981–984. PMID 27890586. doi:10.1016/j.envpol.2016.11.059. hdl:10256/14115.
- ↑ Mann, NH (2005-05-17). "The Third Age of Phage". PLOS Biology 3 (5): 753–755. PMC 1110918. PMID 15884981. doi:10.1371/journal.pbio.0030182.
- ↑ Wommack KE, Colwell RR (March 2000). "Virioplankton: viruses in aquatic ecosystems". Microbiology and Molecular Biology Reviews 64 (1): 69–114. PMC 98987. PMID 10704475. doi:10.1128/MMBR.64.1.69-114.2000.
- ↑ Bergh O, Børsheim KY, Bratbak G, Heldal M (August 1989). "High abundance of viruses found in aquatic environments". Nature 340 (6233): 467–68. Bibcode:1989Natur.340..467B. PMID 2755508. doi:10.1038/340467a0.
- ↑ Bickle TA, Krüger DH (June 1993). "Biology of DNA restriction". Microbiological Reviews 57 (2): 434–50. PMC 372918. PMID 8336674. doi:10.1128/MMBR.57.2.434-450.1993.
- ↑ Barrangou R, Fremaux C, Deveau H, Richards M, Boyaval P, Moineau S, et al. (March 2007). "CRISPR provides acquired resistance against viruses in prokaryotes". Science 315 (5819): 1709–12. Bibcode:2007Sci...315.1709B. PMID 17379808. doi:10.1126/science.1138140.
- ↑ Brouns SJ, Jore MM, Lundgren M, Westra ER, Slijkhuis RJ, Snijders AP, et al. (August 2008). "Small CRISPR RNAs guide antiviral defense in prokaryotes". Science 321 (5891): 960–64. Bibcode:2008Sci...321..960B. PMC 5898235. PMID 18703739. doi:10.1126/science.1159689.
- ↑ Mojica FJ, Rodriguez-Valera F (September 2016). "The discovery of CRISPR in archaea and bacteria". The FEBS Journal 283 (17): 3162–69. PMID 27234458. doi:10.1111/febs.13766.
- ↑ Falkowski, P.G.; Fenchel, T.; Delong, E.F. (2008). "The microbial engines that drive Earth's biogeochemical cycles". Science 320 (5879): 1034–1039. Bibcode:2008Sci...320.1034F. PMID 18497287. doi:10.1126/science.1153213.
- ↑ Brum, J.R.; Sullivan, M.B. (2015). "Rising to the challenge: accelerated pace of discovery transforms marine virology". Nature Reviews Microbiology 13 (3): 147–159. PMID 25639680. doi:10.1038/nrmicro3404.
- ↑ 73,0 73,1 Murata, K.; Zhang, Q.; Galaz-Montoya, J.G.; Fu, C.; Coleman, M.L.; Osburne, M.S.; Schmid, M.F.; Sullivan, M.B.; Chisholm, S.W.; Chiu, W. (2017). "Visualizing adsorption of cyanophage P-SSP7 onto marine Prochlorococcus". Scientific Reports 7: 44176. Bibcode:2017NatSR...744176M. PMC 5345008. PMID 28281671. doi:10.1038/srep44176.
- ↑ Bouman, H.A.; Ulloa, O.; Scanlan, D.J.; Zwirglmaier, K.; Li, W.K.; Platt, T.; Stuart, V.; Barlow, R.; Leth, O.; Clementson, L.; Lutz, V. (2006). "Oceanographic basis of the global surface distribution of Prochlorococcus ecotypes". Science 312 (5775): 918–921. Bibcode:2006Sci...312..918B. PMID 16690867. doi:10.1126/science.1122692.
- ↑ Strauss, James H.; Sinsheimer, Robert L. (July 1963). "Purification and properties of bacteriophage MS2 and of its ribonucleic acid". Journal of Molecular Biology 7 (1): 43–54. PMID 13978804. doi:10.1016/S0022-2836(63)80017-0.
- ↑ Miller, ES; Kutter, E; Mosig, G; Arisaka, F; Kunisawa, T; Rüger, W (March 2003). "Bacteriophage T4 genome". Microbiology and Molecular Biology Reviews 67 (1): 86–156, table of contents. PMC 150520. PMID 12626685. doi:10.1128/MMBR.67.1.86-156.2003.
- ↑ Ackermann, H.-W.; Krisch, H. M. (6 April 2014). "A catalogue of T4-type bacteriophages". Archives of Virology 142 (12): 2329–2345. PMID 9672598. doi:10.1007/s007050050246.
Véxase tamén[editar | editar a fonte]
Outros artigos[editar | editar a fonte]
- Virófago, virus que infectan outros virus
- Bacterívoro
- Faxémido
- CrAssphage
- Ecoloxía de fagos
- Polifago
- Transdución
- Virioma
- CRISPR
Bibliografía[editar | editar a fonte]
- Hauser, AR (2016). "Beyond Antibiotics: New Therapeutic Approaches for Bacterial Infections". Clinical Infectious Diseases 63 (1): 89–95. PMC 4901866. PMID 27025826. doi:10.1093/cid/ciw200.
- Strathdee, Steffanie; Patterson, Tom (2019). The Perfect Predator. Hachette Books. ISBN 978-0316418089.
Ligazóns externas[editar | editar a fonte]
- Häusler, T. (2006) "Viruses vs. Superbugs", Macmillan
- Animación de bacteriófagos atacando E. coli
- Phage.org co información xeral sobrfe bacteriófagos
- Ilustracións e xenómica de bacteriófagos Arquivado 29 de outubro de 2013 en Wayback Machine.
- Bacteriophages get a foothold on their prey
- NPR Science Friday podcast, "Using 'Phage' Viruses to Help Fight Infection", abril 2008
- Animación cientificamente corrfecta do bacteriófago T4 atacando E. coli
- Animación de Hybrid Animation Medical dun bacteriófago T4 atacando E. coli
- Bacteriophages: What are they. Presentación do profesor Graham Hatfull, Universidade de Pittsburgh no YouTube
