Mutación

En bioloxía, unha mutación é unha alteración na secuencia de nucleótidos do xenoma dun organismo, virus ou ADN extracromosómico.[1] As mutacións que se orixinan por erros durante a replicación do ADN, mitose e meiose ou outros tipos de danos no ADN (como dímeros de pirimidina que poden ser causados pola exposición á radiación ou aos carcinóxenos). Eses danos poden despois sufrir unha reparación tendente ao erro (especialmente a unión de extremos mediada por microhomoloxía[2]) ou pode orixinarse un erro durante outras formas de reparación[3][4] ou mesmo durante a replicación (síntese translesión). As mutacións poden tamén ser o resultado dunha inserción ou deleción de segmentos do ADN debido a elementos xenéticos móbiles.[5][6][7]
As mutacións non sempre producen cambios discernibles nas características observables do fenotipo dun organismo. As mutacións desempeñan un papel tanto en procesos biolóxicos normais coma anormais, como a evolución, cancro e o desenvolvemento do sistema inmunitario, incluíndo a diversidade de unión (junctional diversity).
Os xenomas de virus de ARN están baseados no ARN en vez de no ADN. O xenoma viral de ARN pode ser bicatenario (como no ADN) ou monocatenario. Nalgúns destes virus (como o monocatenario VIH causante da SIDA), a replicación ocorre rapidamente, e non hai mecanismos para comprobar a exactitude da copia do xenoma. Este proceso tendente ao erro adoita orixinar moitas mutacións. Os virus de ADN sofren un menor número de mutacións.
As mutaciones poden ser moi diferentes tipos de cambios nas secuencias. As mutacións nun xene poden ás veces non ter efectos, ou alterar o produto xénico ou impedir que o xene funcione correctamente ou completamente. Poden ocorrer tamén en rexións non xénicas. Un estudo de 2007 sobre a variación xenética entre varias especies da mosca Drosophila indicou que se unha mutación cambia unha proteína producida por un xene, o resultado é probablemente nocivo, e estimouse un 70% de polimorfismo de aminoácidos que teñen efectos prexudiciais e o resto son ou neutros ou marxinalmente beneficiosos.[8] Debido aos efectos daniños que poden ter as mutacións nos xenes, os seres vivos teñen mecanismos como a reparación do ADN para impedir ou corrixir as mutacións, revertendo a secuencia mutada ao seu estado orixinal.[5]
Introdución
[editar | editar a fonte]As mutacións poden consistir en duplicacións de grandes seccións do ADN, xeralmente por recombinación xenética.[9] Estas duplicacións son unha das principais fontes para a evolución de novos xenes, na que están implicados centos de xenes duplicados nos xenomas animais cada millóns de anos.[10] A maioría dos xenes pertencen a familias xénicas grandes que descenden dun mesmo xene, detectables pola súa homoloxía de secuencia.[11] Os novos xenes son producidos por varios métodos, comunmente pola duplicación e mutación dun xene ancestral ou por partes recombinads de diferentes xenes para formar novas combinacións con novas funcións.[12][13]
Aquí, os dominios proteicos actúan como módulos, cada un dos cales ten unha función particular e independente, que pode ser mesturada para producir xenes que codifican novas proteínas con novas propiedades.[14] Por exemplo, o ollo humano usa catro xenes para construír estruturas sensibles á luz: tres para os conos da retina ou visión das cores e unha para os bastóns ou visión nocturna; os catro orixináronse a partir dun único xene ancestral.[15] Outra vantaxe de duplicar un xene (ou mesmo un xenoma enteiro) é que este incremente a súa redundancia; isto permite que un xene do par adquira unha nova función, mentres que a outra copia realiza a función orixinal.[16][17] Outros tipos de mutacións crean ocasionalmente novos xenes a partir de ADN que previamente era non codificante.[18][19]
Os cambios no número de cromosomas poden orixinar mutacións incluso máis grandes, nas que segmentos de ADN dos cromosomas rompen e recolócanse. Por exemplo, nos Homininae, fusionáronse dous cromosomas para producir o cromosoma 2 humano; esta fusión non ocorreu na liñaxe doutros simios, os cales manteñen estes cromosomas separados, polo que teñen un par de cromosomas máis que os humanos.[20] Na evolución, o papel máis importante deste tipo de rearranxos cromosómicos pode ser o de acelerar a diverxencia dunha poboación en novas especies ao faceren menos probable que as pobaociósn se hibriden, conservando así as diferenzas xenéticas entre elas.[21]
As secuencias de ADN que poden moverse polo xenoma, como os transposóns, constitúen unha fracción importante do material xenético de plantas e animais e puideron ter sido importantes na evolución dos xenomas.[22] Por exemplo, no xenoma humano existen máis dun millón de copias de secuencias Alu e estas secuencias foron agora en moitos casos recrutadas para realizar funcións como a regulación da expresión xénica.[23] Outro efecto destas secuencias de ADN móbiles é que cando se moven dentro dun xenoma, poden facer mutar ou eliminar xenes existentes e así producir diversidade xenética.[6]
As mutacións non letais acumúlanse dentro do acervo xenético e fan aumentar as variacións xenéticas.[24] A abundancia dalgúns cambios xenéticos dentro do acervo xenético pode ser reducido pola selección natural, mentres que outras mutacións "máis favorables" poden acumularse e ter como resultado cambios adaptativos.

Por exemplo, unha bolboreta pode producir descendencia con novas mutacións. A maioría destas mutacións non terán efectos; pero unha podería cambiar a cor da descendencia da bolboreta, facendo máis difícil (ou máis doado) para os predadores vela. Se este cambio de cor é vantaxoso, as posibilidades de supervivencia desta bolboreta e de que produza a súa propia descendencia serán algo mellores, e co tempo o número de bolboretas con esta mutación poden formar unha porcentaxe máis grande da poboación.
As mutacións neutras defínense como mutacións cuxos efectos non inflúen na fitness dun individuo. A súa frecuencia pode incrementarse co tempo debido á deriva xenética. Crese que a maioría das mutacións non teñen un efecto significativo sobre a fitness dun organismo.[25] Ademais, os mecanismos de reparación do ADN poden arranxar a maioría dos cambios antes de que se convertan en mutacións permanentes e moitos organismos teñen mecanismos para eliminar células somáticas que doutro modo quedarían permanentemente mutadas.
As mutacións beneficiosas poden mellorar o éxito reprodutivo.[26][27]
Causas
[editar | editar a fonte]- Artigo principal: Mutaxénese.
Catro clases de mutacións naturais son: (1) mutacións espontáneas (degradación molecular), (2) mutacións debidas ao bypass de replicación tendente ao erro de danos no ADN por causas naturais (tamén chamadas síntese translesión tendente ao erro), (3) erros introducidos durante a reparación do ADN, e (4) mutacións inducidas causadas por mutáxenos. Os científicos tamén poden introducir deliberadamente secuencias mutantes por manipulación do ADN para faceren experimentos científicos.
Un estudo de 2017 afirmou que o 66% das mutacións causantes de cancro son aleatorias, o 29% son debidas ao ambiente (a poboación estudada abranguía 69 países) e o 5% eran herdadas.[28]
Os humanos como media transmiten 60 mutacións novas aos seus fillos, pero poden transmitir máis dependendo da súa idade, e cada por cada ano de idade engádense dúas novas mutacións aos fillos.[29]
Mutación espontánea
[editar | editar a fonte]As mutacións espontáneas ocorren cunha probabilidade distinta de cero mesmo en persoas con boa saúde e células non contaminadas. Poden clasificarse de acordo ao seu cambio específico:[30]
- Tautomería.— Unha base cambia polo reposicionamento dun átomo de hidróxeno, alterando o padrón de enlaces de hidróxeno desa base, orixinando un emparellamento de bases incorrecto durante a replicación.
- Despurinación.— Perda dunha base púrica (A ou G) para formar un sitio apurínico (sitio AP).
- Desaminación.— Por hidrólise cambia unha base normal a unha base atípica que contén un grupo ceto en lugar do grupo amino orixinal. Exemplos son: C → U e A → HX (hipoxantina), que poden ser corrixidos polos mecanismos de reparación do ADN; e 5MeC (5-metilcitosina) → T, que é menos probable que sexa detectada como unha mutación porque a timina é unha base normal do ADN.
- Discordancia de febra escorregada (slipped strand mispairing), tamén chamada escorregamento de replicación.— Desnaturalización da nova febra a partir da febra molde durante a replicación, seguida da renaturalización nun punto diferente ("escorregamento" ou "slipping"). Isto pode orixinar insercións ou delecións.
Bypass da replicación tendente ao erro
[editar | editar a fonte]Hai cada vez máis probas de que a maioría das mutacións que se orixinan espontaneamente débense á repicación tendente ao erro (síntese translesión) despois dun dano no ADN na febra molde. Os danos oxidativos no ADN ocorren por causas naturais e orixínanse polo menos unhas poucas miles de veces por célula e por día en humanos, aínda que a maioría son reparadas.[31] En ratos, a maioría das mutacións son causadas por síntese translesión.[32] Igualmente, en lévedos, Kunz et al.[33] atoparon que máis do 60% das substitucións dun só par de bases espontáneas e das delecións eran causadas por síntese translesión.
Erros introducidos durante a reparación do ADN
[editar | editar a fonte]- Véxase tamén: Danos no ADN por causas naturais e Reparación do ADN.
Aínda que as roturas de dobre febra no ADN por causas naturais ocorren a frecuencias relativamente baixas no ADN, a súa reparación a miúdo causa mutacións. A unión de extremos non homólogos (NHEJ) é o mecanismo principal para a reparación de roturas de dobre febra. A unión de extremos no homólogos afecta a uns poucos nucleótidos para permitir o aliñamento algo inexacto dos dous extremos para unilos e seguidamente engadir nucleótidos para encher os ocos. Como consecuencia, este mecanismo introduce mutacións.[34]

Mutación inducida
[editar | editar a fonte]
As mutacións inducidas son alteracións no xene causadas por estar en contacto con mutáxenos e por causas ambientais.
As mutacións inducidas a nivel molecular pode ser causadas por:
- Compostos químicos.
- Hidroxilamina.
- Análogos de bases (por exemplo, a bromodesoxiuridina (BrdU)).
- Axentes alquilantes (por exemplo, N-etil-N-nitrosourea (ENU). Estes axentes poden facer mutar tanto ADN replicante coma non replicante. En contraste, un análogo de bases pode facer mutar o ADN só cando o análogo se incorpora ao ADN en replicación. Cada unha destas clases de mutáxenos químicos teñen certos efectos que despois levan á aparición de transicións, transversións ou delecións.
- Axentes que forman adutos do ADN (por exemplo, a ocratoxina A).[36]
- Axentes intercalantes (por exemplo, bromuro de etidio).
- Enlazadores cruzados do ADN.
- Danos oxidativos.
- O ácido nitroso converte os grupos amino de A e C en grupos diazo, alterando os seus padróns de enaces de hidróxeno, o que orixina un apareamento de bases incorrecto durante a replicación.
- Radiación.
- Luz ultravioleta (UV) (radiación non ionizante). Dúas bases nucleotídicas no ADN (citosina e timina) son moi vulnerables á radiación, que lles pode facer cambiar as súas propiedades. A luz UV pode inducir que bases pirimidínicas adxacentes na febra do ADN queden unidas covalentemente formando un dímero de pirimidina. A radiación UV, en particular a UVA de onda máis longa, poden causar danos oxidativos no ADN.[37]
- Radiación ionizante. A exposición á radiación ionizante, como a radiación gamma, pode ter como resultado mutacións, posiblemente causando cancro e a morte.
Aínda que antes se asumía que as mutacións ocorrían por azar ou eran inducidas por mutáxenos, descubríronse mecanismos moleculares que crean mutacións en bacterias e por toda a árbore da vida. Como dixo S. Rosenberg, "Estes mecanismos revelan unha imaxe de mutaxénese altamente regulada, regulada á alza temporalmente por respostas ao estrés e activada cando as células/organismos están mal adaptados aos seus ambientes, cando están sometidos a estreses, acelerando potencialente a adaptación." [38] Como son mecanismos mutaxénicos autoinducidos que incrementan a taxa de adaptación dos organismos, foron ás veces denominados mecanismos de mutaxénese adaptativos e inclúen a resposta SOS en bacterias,[39] a recombinación intracromosómica ectópica [40] e outros eventos cromosómicos como as duplicacións.[38]
Clasificación dos tipos de mutacións
[editar | editar a fonte]- Véxase tamén: Anormalidades cromosómicas.
Por efectos sobre a estrutura
[editar | editar a fonte]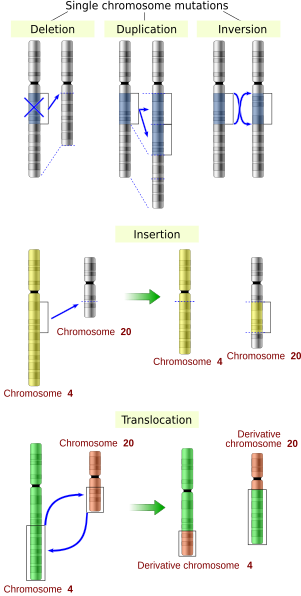
A secuencia dun xene pode ser alterada de diversas maneiras.[41] As mutacións xénicas teñen efectos diversos sobre a saúde dependendo de onde ocorren e de se alteran a función de proteínas esenciais. As mutacións na estrutura dos xenes poden ser clasificadas en varios tipos, que son:
Mutacións a grande escala
[editar | editar a fonte]As mutacións a grande escala en estruturas cromosómicas son:
- Amplificacións (ou duplicacións xénicas) ou repetición dun segmento cromosómicio ou presenza dun cacho extra dun fragmento roto dun cromosoma que pode quedar unido a un cromosoma homólogo ou non homólogo de modo que algúns xenes estean presentes en máis de dúas doses, orixinando múltiples copias de todas as rexións cromosómicas, incrementando a dosificación dos xenes localizados dentro deles.
- As delecións de grandes rexións cromosómicas, causan a perda dos xenes desas rexións.
- As mutacións cuxos efectos son xuntar cachos previamente separados do ADN, o que potencialmente pode deixar xuntos xenes separados para formar un xene de fusión funcionalmente distinto (por exemplo, o bcr-abl).
- Cambios a grande escala na estrutura dos cromosomas chamados rearranxos cromosómicos que poden causar unha diminución da fitness pero tamén a especiación en poboacións illadas endogámicas. Entre estes están:
- Translocacións cromosómicas: intercambio de fragmentos xenéticos entre cromosomas non homólogos.
- Inversións cromosómicas: reverten a orientación dun segmento cromosómico.
- Sobrecruzamento cromosómico non homólogo.
- Delecións intersticiais: unha deleción intracromosómica que elimina un segmento de ADN dun só cromosoma, o que xustapón xenes antes afastados. Por exemplo, as células illadas dun astrocitoma humano, un tipo de tumor cerebral, teñen unha deleción cromosómica que elimina secuencias entre o xene chamado "Fused in Glioblastoma" (FIG) e o do receptor da tirosina quinase (ROS), producindo unha proteína de fusión (FIG-ROS). A proteína de fusión anormal FIG-ROS ten unha actividade de quinase activa constitutivamente que causa unha transformación oncoxénica (unha transformación de células normais en células cancerosas).
- A perda de heterocigosidade: perda dun alelo, ou por unha deleción ou por un evento de recombinación, nun organismo que previamente tiña dous alelos diferentes.
Mutacións a pequena escala
[editar | editar a fonte]As mutacións a pequena escala afectan un xene nun ou uns poucos nucleótidos. Se só afectan un nucleótido chámanse mutacións puntuais. As mutacións a pequena escala son:
- As insercións engaden un ou máis nucleótidos no ADN. Son causadas normalmente por elementos transpoñibles, ou erros durante a replicación de elementos repetidos. As insercións na rexión codificante dun xene poden alterar o empalme de ARNm (mutación de sitio de empalme), ou causan un desprazamento no marco de lectura (mutación por desprazamento do marco de lectura), ambas as cales causan unha significativa alteración do produto xénico. As insercións poden ser revertidas por excisión do elemento transpoñible.
- As delecións ou deficiencias eliminan un ou máis nucleótidos do ADN. Igual que as insercións, estas mutacións poden alterar o marco de lectura do xene. En xeral, son irreversibles. Aínda que en teoría podería restaurarse exactamente a mesma secuencia por medio dunha inserción, os elementos transpoñibles que poden reverter unha deleción moi curta (por exemplo de 1 ou 2 bases) en calquera localización, teñen unha existencia altamente improbable ou non existen en absoluto.
- As mutacións por substitución, a miúdo causadas por compostos químicos ou un mal funcionamento da replicación do ADN, intercambian un só nucleótido por outro.[42] Estes cambios son clasificados como transicións ou transversións.[43] A máis común é a transición, que intercambia unha purina por unha purina (A ↔ G) ou unha pirimidina por unha pirimidina, (C ↔ T). Unha transición pode ser causada polo ácido nitroso, emparellamento incorrecto de bases ou análogos de bases mutaxénicos como o BrdU. Menos comúns son as transversións, que intercambian unha purina por unha pirimidina ou unha pirimidina por unha prina (C/T ↔ A/G). Un exemplo dunha transversión é a conversión dunha adenina (A) nunha citosina (C). As mutacións puntuais son modificacións dun só par de bases do ADN ou outros pequenos grupos de pares de bases nun punto dun xene. Unha mutación puntual pode ser revertida por outra mutación puntual, na cal o nucleótido é cambiado ao seu estado orixinal (reversión verdadeira) ou por unha reversión de segundo sitio (unha mutación complementaria noutro lugar que resulta nun xene que recupera a súa funcionalidade). Como se discute máis abaixo, as mutacións puntais que ocorren dentro de rexións codificantes de proteínas dun xene poden ser clasificadas como substitucións sinónimas ou non sinónimas, as últimas das cales á súa vez poden dividirse en mutacións con cambio de sentido ou mutacións sen sentido.
Por impacto na secuencia de proteínas
[editar | editar a fonte]- Unha mutación por desprazamento do marco de lectura é unha mutación causada pola inserción ou deleción nunha secuencia de ADN de certo número de nucleótidos que non é divisible por tres. Debido a que os codóns están formados por tripletes e que a expresión dos xenes está baseada nos codóns, a inserción ou deleción pode distorsionar o marco de lectura ou o agrupamento de codóns, o que dá lugar a unha tradución completmente diferente a partir da orixinal.[44] Canto máis ao principio da secuencia ocorre a deleción ou inserción, maior é a alteración producida na proteína. (Por exemplo, a secuencia de codóns CCU GAC UAC CUA codifica os aminoácidos prolina, ácido aspártico, tirosina e leucina, respectivamente. Se eliminamos o U do CCU, a secuencia resultante sería CCG ACU ACC UAx, que codificaría prolina, treonina, treonina e parte doutro aminoácido ou quizais dun codón de terminación (onde x significa o seguinte nucleótido).) En contraste, calquera inserción ou deleción que é divisible por tres denomínase mutación no marco ou en pauta.
- Unha mutación por substitución puntual é o resultado dun cambio nun só nucleótido e pode ser sinónima ou non sinónima.
- Unha substitución sinónima substitúe un codón por outro codón que codifica o mesmo aminoácido, así que a secuencia de aminoácidos producida non é modificada. As mutacións sinónimas ocorren debido a que o código xenético é dexenerado. Se esta mutación non ten como resultado ningún efecto fenotípico, entón denomínase mutación silenciosa, pero non todas as substitcións sinónimas son silenciosas. (Pode haber tamén mutacións silenciosas en nucleótidos fóra de rexións codificantes, como os intróns, porque a secuencia de nucleótidos exacta non é crucial como si ocorre nas rexións codificantes, pero estas nos se consideran substitcións sinónimas.)
- Unha substitución non sinónima substitúe un codón por outro codón que codifica un aminoácido diferente, así que a secuencia de aminoácidos producida é modificada. As substituciónn non sinónimas poden ser clasificadas como mutación sen sentido ou con cambio de sentido:
- Unha mutación con cambio de sentido cambia un nucleótido causando a substitución do aminoácido orixinal por un aminoácido diferente. Este á súa vez pode facer que a proteína resultante sexa non funcional. Tales mutacións son responsables de doenzas como a epidermólise ampular, anemia falciforme e esclerose laeral amiotrófica mediada por SOD1.[45] Por outra parte, se ocorre unha mutación con cambio de sentido nun codón o aminoácido orixinal é cambiado por un aminoácido diferente pero quimicamente similar, entón ás veces a proteína sofre poucos ou ningún cambios. Por exemplo, un cambio de AAA a AGA cambiará a lisina por arxinina, que é un aminoácido quimicamente similar. Neste último caso a mutación terá pouco ou ningún efecto sobre o fenotipo e, por tanto, será neutra.
- Unha mutación sen sentido é unha mutación puntual nunha secuencia de ADN que ten como resultado a aparición dun codón de finalización prematuro ou un codón sen sentido no ARNm transcrito, e posiblemente un produto proteico truncado xeralmente non funcional. Este tipo de mutación foi ligada a diferentes mutacións, como a hiperplasia adrenal conxénita. (Ver codón de finalización ou de stop.)
Por efectos sobre a función
[editar | editar a fonte]- As mutación de perda de función, tamén chamadas mutacións inactivantes, teñen como resultado que o produto xénico funciona peor ou non funciona, é dicir, está parcial ou totalmente inctivado. Cando o alelo sofre unha completa falta de función (alelo nulo) adoita denominarse mutación amorfa ou amórfica no esquema de morfos de Muller. Os fenotipos asociados con esas mutacións adoitan ser recesivos. As excepcións danse cando o organismo é haploide, ou cando a dose reducida dun produto xénico normal non é suficiente para ter un fenotipo normal (o que se chama haploinsuficiencia).
- As mutacións de ganancia de función, tamén chamadas mutcións activantes, cambian o produto xénico de modo que o seu efecto se fai máis forte (activación potenciada) ou mesmo é substituído por unha función diferente e anormal. Cando se crea o novo alelo, un heterocigoto que contén o novo alelo creado xunto co orixinal expresará o novo alelo; xeneticamente isto define as mutacións como fenotipos dominantes. Varios morfos de Muller corresponden a ganancias de función, incluíndo os hipermorfos (expresión xénica incrementada) e neomorfos (nova función).[46]
- As mutacións dominantes negativas (tamén chamadas mutacións antimórficas) teñen un produto xénico alterado que actúa antagonistamente con respecto ao alelo de tipo silvestre. Estas mutacións xeralmente resultan na alteración da función molecular (a miúdo inactivándoa) e están caracterizados por ter un fenotipo dominante ou semidominante. Nos humanos, as mutacións negativas dominantes foron implicadas no cancro (por exemplo, as mutacións nos xenes p53,[47] ATM,[48] CEBPA[49] e PPAR-gamma[50]). A síndrome de Marfan é causada por mutacións no xene FBN1, localizado no cromosoma 15, que codifica a fibrilina-1, unha glicoproteína compoñente da matriz extracelular.[51] A síndrome de Marfan é tamén un exemplo de mutación negativa dominante e haploinsuficiencia.[52][53]
- Os hipomorfos, segundo a clasificadión de Muller, caracterízanse por ter produtos xénicos alterados que actúan cunha diminución da expresión xénica do alelo silvestre. Xeralmente, as mutacións hipomórficas son recesivas, pero a haploinsuficiencia causa que algúns alelos sexan dominantes.
- Os neomorfos caracterízanse polo control da síntese de novos produtos proteicos.
- As mutacións letais son mutacións que orixinan a morte dos organismos que portan as mutacións.
- Unha mutacións retrógrada ou reversión é unha mutación puntual que restaura a secuencia orixinal e, por tanto, o fenotipo orixinal.[54]
Por efectos sobre a fitness
[editar | editar a fonte]- Véxase tamén: Fitness (bioloxía).
En xenética aplicada é usual falar de mutacións nocivas e beneficiosas.
- Unha mutación nociva ou prexudicial fai dimunuír a fitness do organismo.
- Unha mutación beneficiosa ou vantaxosa incrementa a fitness do organismo.
- Unha mutación neutra non ten efectos daniños nin beneficiosos sobre o organismo. Tales mutacións ocorren a unha velocidade constante, e son a base dos chamados reloxos moleculares. Na teoría neutralista da evolución molecular, as mutacións neutras son afectadas pola deriva xenética, na que se basea a maior parte da variabilidade a nivel molecular.
- Unha mutación case neutra é unha mutación que pode ser lixeiramente prexudicial ou vantaxosa, aínda que a maioría das mutacións case neutras son lixeiramente prexudiciais.
Distribución de efectos na fitness
[editar | editar a fonte]Intentouse inferir a distribución dos efectos sobre a fitness usando experimentos de mutaxénese e modelos teólricos aplicados a datos de secuencias moleculares. A distribución dos efectos sobre a fitness, usados para determinar a abundancia relativa de tipos diferentes de mutacións (é dicir, fortemente prexudiciais, case neutras ou vantaxosas), é relevante para moitas cuestións evolutivas, como o mantemento da variabilidade xenética,[55] a velocidade de decadencia xenómica,[56] o mantemento da reprodución sexual por cruzamento de individuos non relacionados como oposta á endogamia[57] e a evolución do sexo e a recombinación xenética.[58] A distribución de efectos sobre a fitness pode ser rastreada pola asimetría da distribución de mutacións con efectos aparentemente graves comparada coa distribución de mutacións con efectos aparentemente leves ou ausentes.[59] En resumo, a distribución dos efectos sobre a fitness xoga un importante papel na predición da dinámica evolutiva.[60][61] Utilizáronse diversas estratexias para estudar a distribución dos efectos sobre a fitness, como os métodos teórico, experimental e analítico.
- Experimentos de mutaxénese. O método directo para investigar a distribución dos efectos na fitness é inducir mutacións e despois medir os efectos sobre a fitness mutacionais, o cal xa se fixo en virus, bacterias, lévedos e Drosophila. Por exemplo, a maioría destes estudos en virus utilizaron mutaxénese dirixida a sitio para crear mutacións puntuais e medir a fitness relativa de cada mutante.[62][63][64][65] Na bacteria Escherichia coli un estudo utilizou a mutaxénese de transposón para medir directamente a fitness da inserción aleatoria dun derivado de Tn10.[66] En lévedos, desenvolveuse unha mutaxénese combinada e unha estratexia de secuenciación profunda para xerar bibliotecas sistemáticas de mutantes de alta calidade e medir a fitness con alto rendemento.[67] Porén, dado que moitas mutacións teñen efectos demasiado pequenos para ser detectados[68] e que os experimentos de mutaxénese poden detectar só mutacións de efectos moderadamente grandes, a análise de datos de secuencias de ADN pode proporcionar valiosa información sobre estas mutacións.
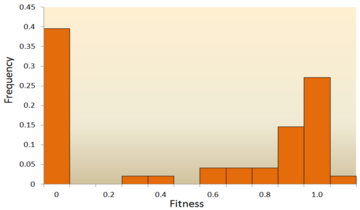
- Análise de secuencias moleculares. Co rápido desenvolvemento da tecnoloxía de secuenciación de ADN, está dispoñible unha enorme cantidade de datos de secuencias de ADN e incluso se espera máis no futuro. Desenvolvéronse varios métodos para inferir a distribución dos efectos na fitness a partir de datos de secuencias de ADN.[69][70][71][72] Examinando as diferenzas entre secuencias de ADN nunha especie ou entre especies, pódense inferir varias características da distribución de efectos na fitness para mutacións neutras, prexudiciais e vantaxosas.[24] En concreto, a estratexia da análise de secuencias de ADN permite a estimación dos efectos das mutacións con efectos moi pequenos, que apenas son detectables por medio de experimentos de mutaxénese.
Uns dos primeiros estudos teóricos da distribución de efectos na fitness fíxoos Motoo Kimura, un influente xenetista de poboacións teórico. A súa teoría neutralista da evolución molecular propón que a maioría das novas mutacións serán altamente prexudiciais, cunha pequena fracción que é neutra.[25][73] Hiroshi Akashi propuxo máis recentemente un modelo bimodal para a distribución de efectos na fitness, con modas centradas arredor de mutacións altamente prexudiciais e neutras.[74] Ambas as teorías concordan en que a gran maioría das novas mutacións son neutras ou prexudiciais e que as mutacións vantaxosas son raras, o que foi apoiado polos resultados experimentais. Un exemplo é un estudo feito sobre a distribución de efectos na fitness das mutacións aleatorias no virus da estomatite vesicular.[62] De todas as mutacións, o 39,6% eran letais, o 31,2% eran prexudiciais non letais e o 27,1% eran neutras. Outro exemplo procede de experimentos de mutaxénese de alto rendemento con lévedos.[67] Nestes experimentos mostrouse que a distribución de efectos na fitness global é bimodal, cun agrupamento de mutacións neutras, e unha ampla distribución de mutacións prexudiciais.
Aínda que relativamente poucas mutacións son vantaxosas, aquelas que si o son xogan un importante papel nos cambios evolutivos.[75] Igual que as mutacións neutras, as mutacións vantaxosas debilmente seleccionadas poden perderse debido á deriva xenética aleatoria, pero as mutacións vantaxoas fortemente seleccionadas é máis probable que sexan fixadas. Coñecer a distribución de efectos na fitness das mutacións vantaxosas poden orixinar un incremento na capacidade de predicir a dinámica evolutiva. O traballo teórico sobre esta dstribución no caso das mutacións vantaxosas realizárono John H. Gillespie[76] e H. Allen Orr.[77] Estes investigadores propuxeron que a distribución de mutacións vantaxosas debería ser exponencial baixo unha ampla gama de condicións, o cal, en xeral, foi apoiado por estudos experimentais, polo menos para as mutacións vantaxosas fortemente seleccionadas.[78][79][80]
En xeral acéptase que a maioría das mutacións son neutras ou prexudiciais, mentres que as mutacións beneficiosas son raras; porén, a proporción de tipos de mutacións varía entre diversas especies. Isto indica dous puntos importntes: primeiro, a proporción de muracións efectivamente neutras é probable que varíe entre especies, o que ten como resultado a dependencia do tamaño efectivo da poboación; segundo, os efectos medios das mutacións prexudiciais varían drasticamente entre especies.[24] Ademais, a distribución de efectos na fitness difire entre rexións codificantes e rexións non codificantes, e as de ADN non codificante contén mutacións debilmente seleccionadas.[24]
Por herdanza
[editar | editar a fonte]
En organismos multicelulares con células reprodutoras, as mutacións pode ser divididas en mutacións na liña xerminal, que poden transmitirse aos descendentes por medio do ADN das células reprodutoras, e mutacións somáticas (tamén chamadas mutacións adquiridas),[81] que afectan a células que non interveñen na reprodución e que normalmente non se transmiten á descendencia.
Unha mutación na liña xerminal dá lugar a unha mutación constitucional na descendencia, é dicir, unha mutación que está presente en todas as células. Unha mutación constitucional pode tamén producirse moi pouco despois da fecundación, ou continuar a partir dunha mutación constitucional nun proxenitor.[82]
A distinción entre mutacións na liña xerminal e mutacións somáticas é importante en animais que teñen unha liña xerminal específica para producir as células reprodutoras. Porén, non ten moito valor á hora de comprender os efectos das mutacións en plantas, que carecen dunha liña xerminal específica. A distinción é tamén borrosa en animais que se reproducen asexualmente por mecanismos como a xemación, porque as células que dan lugar ao organismo fillo tamén dan lugar á liña xerminal dese organismo.
Unha nova mutación de liña xerminal non herdada dos proxenitores denomínase mutación de novo.
Os organismos diploides (por exemplo, os humanos) conteñen dúas copias de cada xene, un alelo de orixe paterna e outro de orixe materna. Baseándose na aparición da mutación en cada cromosoma, podemos clasificar as mutacións en tres tipos.
- Unha mutación heterocigota é unha mutación nun só alelo (dos dous que hai para cada xene nos organismos diploides).
- Unha mutación homocigota é unha mutación idéntica en ambos os alelos do xene, paterno e materno.
- As mutacións de heterocigosidade composta ou dun composto xenético consiste en dúas mutacións diferentes nos alelos dese xene, unha no alelo materno e outra no paterno.[83]
Un organismo de tipo silvestre (ou tipo salvaxe) ou organismo non mutado homocigoto é o que non ten ningún dos dous alelos mutado.
Clases especiais
[editar | editar a fonte]- Unha mutación condicional é unha mutación que presenta o fentoipo de tipo silvestre (ou menos grave) en certas condicións "permisivas" do ambiente e un fenotipo mutante baixo certas condicións "restritivas". Por exemplo, unha mutación sensible á temperatura pode causar a morte celular a altas temperaturas (condición restitiva), pero podería non ter consecuencias deletéreas a unha temperatura baixa (condición permisiva).[84] Estas mutacións non son autónomas, xa que a súa manifestación depende da presenza de certas condicións, a diferenza doutras mutacións que aparecen autonomamente.[85] As condicións permisivas poden ser a temperatura,[86] certos compostos químicos,[87] a luz[87] ou mutacións noutras partes do xenoma.[85] Os mecanismos in vivo, como os interruptores trasncricionais (trasncriptional switches) poden crear mutacións condicionais. Por exemplo, a asociación do dominio de unión a esteroides pode crear un interruptor transcricional que pode cambiar a expresión dun xene baseada na presenza dun ligando esteroide.[88] As mutacións condicionais teñen aplicacións en investigación, xa que permiten controlar a expresión xénica. Isto é especialmente útil en adultos ao permitir a expresión despois dun certo período de crecemento, eliminando así os efectos prexudiciais da expresión xénica observada durante as etapas do desenvolvemento en organismos modelo.[87] Os sistemas de ADN recombinase como a Recombinación Cre-Lox usada en aociación con promotores que son activados baixo certas condicións poden xerar mutacións condicionais. A tecnoloxia da Recombinase Dual pode utilizarse para inducir múltiples mutacións condicionais para estudar as doenzas que se manifestan como resultado de mutacións simultáneas en múltiples xenes.[87] Sábese que certas inteínas se empalman só a certas temperaturas permisivas, o que causa a outras temperaturas unha síntese proteica incorrecta e, dese xeito, mutacións de perda de función.[89] As mutacións condicionais poden tamén utilizarse en estudos xenéticos asociados co envellecemento, xa que a expresión pode cambiar despois de pasado certo período de tempo na vida do organismo.[86]
- Os loci de caracteres cuantitativos para a temporización da replicación (ou rtQTL, do inglés replication timing quantitative trait loci) afectan tamén a replicación do ADN.
Nomenclatura
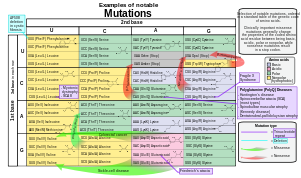
[editar | editar a fonte]Para categorizar unha mutación, debería de identificarse a secuencia "normal", que debe obterse a partir do ADN dun orgnismo "normal" ou con "saúde" (o que se opón a "mutante" ou "enfermo"); idealmente, debería estar a disposición pública para a comparación directa nucleótido a nucleótido, e ter o acordo da comunidade científica ou dun grupo de xenetistas expertos e biólogos, que teñen a responsabilidade de establecer o estándar ou a denominada secuencia "consenso". Este paso require un temendo esforzo científico. Unha vez que se coñece a secuencia consenso, as mutacións dun xenoma poden ser precisadas, descritas e clasificadas. O comité da Human Genome Variation Society (HGVS) desenvolveu a nomenclatura das variantes das secuencias humanas estándar,[90] que deberían utilizar os investigadores e os centros de diagnóstico para xerar descricións non ambiguas das mutacións. En principio, esta nomenclatura pode utilizarse támén para describir mutacións noutros organismos. A nomenclatura especifica o tipo de mutación e os cambios de bases ou aminoácidos. Exemplos de nomenclatura son:
- Substitución de nucleótidos (por exemplo, 76A>T).— O número é a posición do nucleótido desde o extremo 5'; a primeira letra representa o nucleótido de tipo silvestre ou salvaxe, e a segunda letra representa o nucleótido que substitúe o tipo silvestre. No exemplo dado, a adenina na posición 76ª foi substituída por unha timina.
- Se cómpre diferenciar entre mutacións no ADN xenómico, ADN mitocondrial e ARN, utilízase unha convención simple. Por exemplo, se a 100ª base dunha secuencia nucleotídica mutou de G a C, entón esctibiríase g.100G>C se a mutación ocorreu no ADN xenómico, ou escribiríase m.100G>C se ocorreu no ADN mitocondrial, ou r.100g>c se ocorreu no ARN. Nótese que, no caso de mutacións no ARN, os nucleótidos escríbense con minúscula.
- Substitución de aminoácidos (por exemplo, D111E).— A primeira letra é a que simboliza o aminoácido de tipo silvestre (cos símbolos dunha letra dos aminoácidos), o número é a posición do aminoácido desde o extremo N-terminal da proteína, e a segunda letra é o símbolo do aminoácido presente na mutación. As mutacións sen sentido represéntanse cun X para o segundo aminoácido (por exemplo, D111X).
- Deleción de aminoácidos (por exemplo, ΔF508).— A letra grega Δ (delta) indica que é unha deleción. A letra latina seguinte refírese ao aminoácido presente no tipo silvestre e o número é a súa posición desde o N-terminal no tipo silvestre.
Taxas de mutación
[editar | editar a fonte]- Artigo principal: Taxa de mutación.
As taxas de mutación varían substancialmente entre especies, e as forzas evolutivas que xeralmente determinan a mutación están sendo moi investigadas.
Causa de enfermidades
[editar | editar a fonte]Os cambios no ADN causados polas mutacións nunha rexión codificante do ADN poden causar erros en secuencias de proteínas que poden ter como resultado a formación de proteínas parcial ou completamente non funcionais. Cada célula, para poder funcionar correctamente, depende de que miles de proteínas funcionen debidamente nos lugares e momentos axeitados. Cando unha mutación altera unha proteína que desempeña un papel fundamental no corpo, pode causar algún trastorno médico. Algunhas mutacións alteran a secuencia de bases do ADN dun xene pero non cambia a función da proteína producida polo xene. Un estudo sobre a comparación de xenes entre diferentes especies de Drosophila suxire que se unha mutación non cambia unha proteína, a mutación moi probablemente será daniña, cunha estimación dun 70 por cento de polimorfismos de aminoácidos que teñen efectos nocivos e os restantes ou son neutros ou feblemente beneficiosos.[8] Porén, os estudos demostran que só o 7% das mutacións puntuais no ADN non codificante de lévedos son prexudiciais e o 12% no ADN codificante son prexudiciais. O resto das mutacións son neutras ou lixeiramente beneficiosas.[91]
Trastornos herdados
[editar | editar a fonte]- Véxase tamén: Trastorno xenético.
Se unha mutación está presente nunha célula xerminal, pode dar lugar a unha descendencia que porta a mutación en todas as súas células. Isto é o que ocorre nas doenzas hereditarias. En particular, se hai unha mutación nun xene de reparación do ADN nunha célula xerminal, as persoas que porten esas mutacións na liña xerminal poden ter un maior risco de padecer cancro. Un exemplo dunha destas mutacións é o albinismo, unha mutación que ocorre nos xenes OCA1 ou OCA2. Os individuos con este trastorno son máis propensos a moitos tipos de cancros e outros trastornos e teñen alteracións na visión.
Os danos no ADN poden causar un erro cando o ADN se replica, e este erro de replicación pode causar unha mutación xénica que, á súa vez, podería causar un trastorno xenético. Os danos no ADN son reparados polo sistema de reparación do ADN da célula. Cada célula ten diversas vías polas cales os encimas recoñecen e reparan danos no ADN. Como o ADN pode ser danado de moitas maneiras, o proceso de reparación do ADN é un modo importante polo cal o corpo se protexe das doenzas. Unha vez que os danos no ADN deron lugar a unha mutación, a mutación non pode ser reparada.
Papel na carcinoxénese
[editar | editar a fonte]- Véxase tamén: Carcinoxénese.
Por outra parte, unha mutación pode ocorrer nunha célula somática dun organismo. Tales mutacións estarán presentes en todos os descendentes desta célula dentro do mesmo organismo. A acumulación de certas mutacións ao longo de xeracións de células somáticas é en parte a causa da transformación maligna, desde unha célula normal a unha célla cancerosa.[92]
As células con mutacións de perda de función heterocigotas (unha copia boa do xene e unha copia mutada) poden funcionar normalmente coa copia non mutada ata que a copia boa é mutada somaticamente de forma espontánea. Este tipo de mutación ocorre a miúdo en organismos vivos, pero é difícil de medir a súa taxa. A medición desta taxa é importante para predicir a taxa á que as persoas poden desenvolver o cancro.[93]
As mutacións puntuais poden orixinarse a partir de mutacións espontáneas que ocorren durante a replicación do ADN. A taxa de mutación pode ser incrementada por mutáxenos. Os mutáxenos poden ser físicos, como a radiación dos raios UV, os raios X ou a calor extrema, ou compostos químicos (as moléculas que colocan mal as bases ou distorsionan a forma helicoidal do ADN). Os mutáxenos asociados con cancros son frecuentemente estudados para aprender sobre o cancro e a súa prevención.
Mutacións en prións
[editar | editar a fonte]Os prións son proteínas e non conteñen material xenético. Porén, a replicación dos prións está suxeita a mutacións e a selección natural igual que outras formas de replicación.[94] O xene humano PRNP codifica a importante proteína prión PrP, e pode sufrir mutacións que dan lugar a enfermidades causadas por prións.
Mutacións beneficiosas
[editar | editar a fonte]Aínda que as mutcións que causan cambios en secuencias de proteínas poden ser daniñas para un organismo, en ocasións o efecto pode ser positivo nun ambiente dado. Neste caso, a mutación pode capacitar ao organismo mutante para resistir determinados estreses ambientais mellor que os organismos de tipo silvestre, ou reproducirse máis rapidamente. Neses casos unha mutación tenderá a ser máis común nunha poboación por efecto da selección natural. Exemplos son:
Resistencia ao VIH: unha deleción específica de 32 pares de bases no CCR5 humano (CCR5-Δ32) confire resistencia ao VIH aos homocigotos e atrasa o comezo da fase de SIDA en heterocigotos.[95] Unha posible explicación da etioloxía da relativamente alta frecuencia de CCR5-Δ32 na poboación europea é que proporcionou resistencia á peste bubónica no século XIV en Europa. As persoas con esta mutación tiñan máis probabilidades de sobrevivir á infección, polo que a súa frecuencia na poboación se incrementou.[96] Esta teoría podería explicar por que esta mutación non se encontra no sur de África, que non foi afectada pola peste bubónica. Unha nova teoría suxire que a presión selectiva sobre a mutación CCR5 Delta 32 foi causada pola varíola en vez de pola peste bubónica.[97]
Resistencia á malaria: un exemplo dunha mutación nociva é a da anemia falciforme, un trastorno do sangue no cal o corpo produce un tipo anormal da proteína transportadora de oxíxeno hemoglobina dos glóbulos vermellos. Un terzo dos habitantes indíxenas da África subsahariana portan o alelo, porque, en áreas onde a malaria é común, ten un valor de supervivencia portar un só alelo falciforme (carácter de célula falciforme).[98] Aqueles que teñen só un dos dou alelos da anemia falciforme son máis resistentes á malaria, xa que o axente infecciosa da malaria, o Plasmodium queda parado nas células falciformes que infecta.
Resistencia a antibióticos: practicamente todas as bacterias desenvolven resistencia a antibióticos cando se expoñen a antibióticos. De feito, as poboacións bacterianas xa teñen esas mutacións nunha baixa frecuencia, que despois son seleccionadas baixo a presión exercida polos antibióticos.[99] Obviamente, tales mutacións son só beneficiosas para a bacteria, pero non para o que é infectado por elas.
Persistencia da lactase: a persistencia da lactase é unha mutación permitiu aos humanos expresar o encima lactase despois de ser destetados, de modo que de adultos poden dixerir o azucre lactosa do leite, o que é probablemente unha das mutacións máis beneficiosas na evolución humana recente.[100]
Historia
[editar | editar a fonte]
O mutacionismo é unha das varias alternativas á evolución por selección natural que existiron antes e despois da publicación en 1859 do libro de Charles Darwin A orixe das especies. Nesta teoría, a mutación é a fonte de novidades xenéticas, creando novas formas e especies, en ocasións instantaneamente,[101] nun salto súbito.[102] As mutacións eran consideradas como impulsoras da evolución, que estaba limitada pola maior ou menor aparición das mutacións.
Antes de Darwin, os biólogos xeralmente crían no saltacionismo, a posibilidade de grandes saltos evolutivos, incluíndo a especiación inmediata. Por exemplo, en 1822 Étienne Geoffroy Saint-Hilaire argumentou que as especies podían formarse por transformacións repentinas, o que sería posteriormente chamado macromutación.[103] Darwin opúxose ao saltacionismo, insistindo no gradualismo na evolución igual que na en xeoloxía. En 1864, Albert von Kölliker fixo revivir a teoría de Geoffroy.[104] En 1901 o xenetista Hugo de Vries deulle o nome de "mutación" ás novas formas similares que se orixinaban subitamente nos seus experimentos sobre a planta Oenothera lamarckiana, e na primeira década do século XX, o mutacionismo, ou como o chamou de Vries mutationstheorie,[101][105] converteuse nun rival do darwinismo, que foi apoiado durante certo tempo por xenetistas como William Bateson,[106] Thomas Hunt Morgan e Reginald Punnett.[101][107]
A comprensión do mutacionismo enturbouse pola interpretación que fixeron dos primeiros mutacionistas os científicos que apoiaban a síntese moderna da evolución, dicindo que os mutacionistas se opoñían á evolución darwinista, así como os seus rivais da escola biométrica, que argumentaban que a selección operaba sobre a variación continua. Nesta interpretación, os mutacionistas foron derrotados por unha síntese da xenética e a selección natural que se supoñía empezara máis tarde, arredor de 1918, cos traballos do matemático Ronald Fisher.[108][109][110][111] Porén, o aliñamento da xenética mendeliana e a selección natural empezou xa antes, en 1902, cun artigo de Udny Yule,[112] e acumulouse cos traballos teóricos e experimentais realizados en Europa e América. Malia esta controversia, os primeiros mutacionistas xa aceptaran en 1918 a selección natural e explicaban a variación continua como resultado de que moitos xenes actuaban sobre as mesmas características, como a altura.[109][110]
O mutacionismo, xunto con outras alternativas ao darwinismo como o lamarckismo e a ortoxénese, foi descartado pola maioría dos biólogos, xa que consideraban que a xenética mendeliana e a selección natural podían facilmente funcionar xuntas; a mutación pasou a adoptar o papel de fonte da variabilidade xenética esencial sobre a que actuaba a selección natural. Porén, o mutacionismo non se esvaeu totalmente. En 1940, Richard Goldschmidt argumentou outra vez sobre a especiación nun só paso por unha macromutación, describindo os organismos que así se orixinaban como "monstros esperanzados", gañándose un rexeitamento xeral.[113][114] En 1987, Masatoshi Nei argumentou controvertidamente que a evolución estivo a miúdo limitada á mutación.[115] Biólogos modernos como Douglas J. Futuyma concluíron que esencialmente todas as afirmacións de que a evolución é impulsada por grandes mutacións poden ser explicadas pola evolución darwinista.[116]
Notas
[editar | editar a fonte]- ↑ "mutation | Learn Science at Scitable". www.nature.com (en inglés). Consultado o 2018-09-24.
- ↑ Sharma S, Javadekar SM, Pandey M, Srivastava M, Kumari R, Raghavan SC (marzo de 2015). "Homology and enzymatic requirements of microhomology-dependent alternative end joining". Cell Death & Disease 6 (3): e1697. PMC 4385936. PMID 25789972. doi:10.1038/cddis.2015.58.
- ↑ Chen J, Miller BF, Furano AV (abril de 2014). "Repair of naturally occurring mismatches can induce mutations in flanking DNA". eLife 3: e02001. PMC 3999860. PMID 24843013. doi:10.7554/elife.02001.
- ↑ Rodgers K, McVey M (xaneiro de 2016). "Error-Prone Repair of DNA Double-Strand Breaks". Journal of Cellular Physiology 231 (1): 15–24. PMC 4586358. PMID 26033759. doi:10.1002/jcp.25053.
- ↑ 5,0 5,1 Bertram JS (decembro de 2000). "The molecular biology of cancer". Molecular Aspects of Medicine 21 (6): 167–223. PMID 11173079. doi:10.1016/S0098-2997(00)00007-8.
- ↑ 6,0 6,1 Aminetzach YT, Macpherson JM, Petrov DA (xullo de 2005). "Pesticide resistance via transposition-mediated adaptive gene truncation in Drosophila". Science 309 (5735): 764–7. Bibcode:2005Sci...309..764A. PMID 16051794. doi:10.1126/science.1112699.
- ↑ Burrus V, Waldor MK (xuño de 2004). "Shaping bacterial genomes with integrative and conjugative elements". Research in Microbiology 155 (5): 376–86. PMID 15207870. doi:10.1016/j.resmic.2004.01.012.
- ↑ 8,0 8,1 Sawyer SA, Parsch J, Zhang Z, Hartl DL (abril de 2007). "Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila". Proceedings of the National Academy of Sciences of the United States of America 104 (16): 6504–10. Bibcode:2007PNAS..104.6504S. PMC 1871816. PMID 17409186. doi:10.1073/pnas.0701572104.
- ↑ Hastings PJ, Lupski JR, Rosenberg SM, Ira G (agosto de 2009). "Mechanisms of change in gene copy number". Nature Reviews. Genetics 10 (8): 551–64. PMC 2864001. PMID 19597530. doi:10.1038/nrg2593.
- ↑ Carroll SB, Grenier JK, Weatherbee SD (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design (2nd ed.). Malden, MA: Blackwell Publishing. ISBN 978-1-4051-1950-4. LCCN 2003027991. OCLC 53972564.
- ↑ Harrison PM, Gerstein M (maio de 2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". Journal of Molecular Biology 318 (5): 1155–74. PMID 12083509. doi:10.1016/S0022-2836(02)00109-2.
- ↑ Orengo CA, Thornton JM (xullo de 2005). "Protein families and their evolution-a structural perspective". Annual Review of Biochemistry 74: 867–900. PMID 15954844. doi:10.1146/annurev.biochem.74.082803.133029.
- ↑ Long M, Betrán E, Thornton K, Wang W (novembro de 2003). "The origin of new genes: glimpses from the young and old". Nature Reviews. Genetics 4 (11): 865–75. PMID 14634634. doi:10.1038/nrg1204.
- ↑ Wang M, Caetano-Anollés G (xaneiro de 2009). "The evolutionary mechanics of domain organization in proteomes and the rise of modularity in the protein world". Structure 17 (1): 66–78. PMID 19141283. doi:10.1016/j.str.2008.11.008.
- ↑ Bowmaker JK (maio de 1998). "Evolution of colour vision in vertebrates". Eye 12 (Pt 3b): 541–7. PMID 9775215. doi:10.1038/eye.1998.143.
- ↑ Gregory TR, Hebert PD (abril de 1999). "The modulation of DNA content: proximate causes and ultimate consequences". Genome Research 9 (4): 317–24. PMID 10207154. doi:10.1101/gr.9.4.317 (inactivo 2020-03-24).
- ↑ Hurles M (xullo de 2004). "Gene duplication: the genomic trade in spare parts". PLOS Biology 2 (7): E206. PMC 449868. PMID 15252449. doi:10.1371/journal.pbio.0020206.
- ↑ Liu N, Okamura K, Tyler DM, Phillips MD, Chung WJ, Lai EC (outubro de 2008). "The evolution and functional diversification of animal microRNA genes". Cell Research 18 (10): 985–96. PMC 2712117. PMID 18711447. doi:10.1038/cr.2008.278.
- ↑ Siepel A (outubro de 2009). "Darwinian alchemy: Human genes from noncoding DNA". Genome Research 19 (10): 1693–5. PMC 2765273. PMID 19797681. doi:10.1101/gr.098376.109.
- ↑ Zhang J, Wang X, Podlaha O (maio de 2004). "Testing the chromosomal speciation hypothesis for humans and chimpanzees". Genome Research 14 (5): 845–51. PMC 479111. PMID 15123584. doi:10.1101/gr.1891104.
- ↑ Ayala FJ, Coluzzi M (maio de 2005). "Chromosome speciation: humans, Drosophila, and mosquitoes". Proceedings of the National Academy of Sciences of the United States of America. 102 Suppl 1 (Suppl 1): 6535–42. Bibcode:2005PNAS..102.6535A. PMC 1131864. PMID 15851677. doi:10.1073/pnas.0501847102.
- ↑ Hurst GD, Werren JH (agosto de 2001). "The role of selfish genetic elements in eukaryotic evolution". Nature Reviews Genetics 2 (8): 597–606. PMID 11483984. doi:10.1038/35084545.
- ↑ Häsler J, Strub K (novembro de 2006). "Alu elements as regulators of gene expression". Nucleic Acids Research 34 (19): 5491–7. PMC 1636486. PMID 17020921. doi:10.1093/nar/gkl706.
- ↑ 24,0 24,1 24,2 24,3 Eyre-Walker A, Keightley PD (agosto de 2007). "The distribution of fitness effects of new mutations" (PDF). Nature Reviews Genetics 8 (8): 610–8. PMID 17637733. doi:10.1038/nrg2146. Arquivado dende o orixinal (PDF) o 4 de marzo de 2016.
- ↑ 25,0 25,1 Kimura M (1983). The Neutral Theory of Molecular Evolution. Cambridge, UK; New York: Cambridge University Press. ISBN 978-0-521-23109-1. LCCN 82022225. OCLC 9081989.
- ↑ Dover GA, Darwin C (2000). Dear Mr. Darwin: Letters on the Evolution of Life and Human Nature (en inglés). University of California Press. ISBN 9780520227903.
- ↑ Tibayrenc, Michel (2017-01-12). Genetics and Evolution of Infectious Diseases (en inglés). Elsevier. ISBN 9780128001530.
- ↑ "Cancer Is Partly Caused By Bad Luck, Study Finds". Arquivado dende o orixinal o 13 de xullo de 2017.
- ↑ Jha, Alok (22 de agosto de 2012). "Older fathers pass on more genetic mutations, study shows". the Guardian.
- ↑ Montelone BA (1998). "Mutation, Mutagens, and DNA Repair". www-personal.ksu.edu. Arquivado dende o orixinal o 26 de setembro de 2015. Consultado o 2015-10-02.
- ↑ Saul RL, Ames BN. Background levels of DNA damage in the population. Basic Life Science. 1986;38:529-35. PMID 3741344 . DOI 10.1007/978-1-4615-9462-8_55 . Cita: "Baseándose nas medidas de produtos de oxidación do ADN na urina humana, estimamos que a taxa total de formación de todos os produtos de danos oxidativos no ADN no home é da orde duns poucos miles de eventos por célula e por día"
- ↑ Stuart GR, Oda Y, de Boer JG, Glickman BW (marzo de 2000). "Mutation frequency and specificity with age in liver, bladder and brain of lacI transgenic mice". Genetics 154 (3): 1291–300. PMC 1460990. PMID 10757770.
- ↑ Kunz BA, Ramachandran K, Vonarx EJ (abril de 1998). "DNA sequence analysis of spontaneous mutagenesis in Saccharomyces cerevisiae". Genetics 148 (4): 1491–505. PMC 1460101. PMID 9560369.
- ↑ Lieber MR (xullo de 2010). "The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway". Annual Review of Biochemistry 79: 181–211. PMC 3079308. PMID 20192759. doi:10.1146/annurev.biochem.052308.093131.
- ↑ Creado a partir de PDB 1JDG Arquivado 31 de decembro de 2015 en Wayback Machine.
- ↑ Pfohl-Leszkowicz A, Manderville RA (aneiro de 2007). "Ochratoxin A: An overview on toxicity and carcinogenicity in animals and humans". Molecular Nutrition & Food Research 51 (1): 61–99. PMID 17195275. doi:10.1002/mnfr.200600137.
- ↑ Kozmin S, Slezak G, Reynaud-Angelin A, Elie C, de Rycke Y, Boiteux S, Sage E (setembro de 2005). "UVA radiation is highly mutagenic in cells that are unable to repair 7,8-dihydro-8-oxoguanine in Saccharomyces cerevisiae". Proceedings of the National Academy of Sciences of the United States of America 102 (38): 13538–43. Bibcode:2005PNAS..10213538K. PMC 1224634. PMID 16157879. doi:10.1073/pnas.0504497102.
- ↑ 38,0 38,1 Fitzgerald DM, Rosenberg SM (abril de 2019). "What is mutation? A chapter in the series: How microbes "jeopardize" the modern synthesis". PLOS Genetics 15 (4): e1007995. PMC 6443146. PMID 30933985. doi:10.1371/journal.pgen.1007995.
- ↑ Galhardo RS, Hastings PJ, Rosenberg SM (2007-01-01). "Mutation as a stress response and the regulation of evolvability". Critical Reviews in Biochemistry and Molecular Biology 42 (5): 399–435. PMC 3319127. PMID 17917874. doi:10.1080/10409230701648502.
- ↑ Quinto-Alemany D, Canerina-Amaro A, Hernández-Abad LG, Machín F, Romesberg FE, Gil-Lamaignere C (2012-07-31). Sturtevant J, ed. "Yeasts acquire resistance secondary to antifungal drug treatment by adaptive mutagenesis". PLOS One 7 (7): e42279. Bibcode:2012PLoSO...742279Q. PMC 3409178. PMID 22860105. doi:10.1371/journal.pone.0042279.
- ↑ Rahman, Nazneen. "The clinical impact of DNA sequence changes". Transforming Genetic Medicine Initiative. Arquivado dende o orixinal o 04 de agosto de 2017. Consultado o 27 de xuño de 2017.
- ↑ Freese E (abril de 1959). "The Difference Between Spontaneous and Base-Analogue Induced Mutations of Phage T4". Proceedings of the National Academy of Sciences of the United States of America 45 (4): 622–33. Bibcode:1959PNAS...45..622F. PMC 222607. PMID 16590424. doi:10.1073/pnas.45.4.622.
- ↑ Freese E (xuño de 1959). "The specific mutagenic effect of base analogues on Phage T4". Journal of Molecular Biology 1 (2): 87–105. doi:10.1016/S0022-2836(59)80038-3.
- ↑ Hogan, C. Michael (12 de outubro de 2010). "Mutation". En Monosson, Emily. Encyclopedia of Earth. Washington, D.C.: Environmental Information Coalition, National Council for Science and the Environment. OCLC 72808636. Arquivado dende o orixinal o 14 de novembro de 2015. Consultado o 2015-10-08.
- ↑ Boillée S, Vande Velde C, Cleveland DW (outubro de 2006). "ALS: a disease of motor neurons and their nonneuronal neighbors". Neuron 52 (1): 39–59. PMID 17015226. doi:10.1016/j.neuron.2006.09.018.
- ↑ Steenhuysen, Julie (2017-12-19). "U.S. Lifts Funding Ban on Studies That Enhance Dangerous Germs". U.S. News & World Report. Consultado o 2018-01-15.
- ↑ Goh AM, Coffill CR, Lane DP (xaneiro de 2011). "The role of mutant p53 in human cancer". The Journal of Pathology 223 (2): 116–26. PMID 21125670. doi:10.1002/path.2784.
- ↑ Chenevix-Trench G, Spurdle AB, Gatei M, Kelly H, Marsh A, Chen X, Donn K, Cummings M, Nyholt D, Jenkins MA, Scott C, Pupo GM, Dörk T, Bendix R, Kirk J, Tucker K, McCredie MR, Hopper JL, Sambrook J, Mann GJ, Khanna KK (febreiro de 2002). "Dominant negative ATM mutations in breast cancer families". Journal of the National Cancer Institute 94 (3): 205–15. PMID 11830610. doi:10.1093/jnci/94.3.205.
- ↑ Paz-Priel I, Friedman A (2011). "C/EBPα dysregulation in AML and ALL". Critical Reviews in Oncogenesis 16 (1–2): 93–102. PMC 3243939. PMID 22150310. doi:10.1615/critrevoncog.v16.i1-2.90.
- ↑ Capaccio D, Ciccodicola A, Sabatino L, Casamassimi A, Pancione M, Fucci A, Febbraro A, Merlino A, Graziano G, Colantuoni V (xuño de 2010). "A novel germline mutation in peroxisome proliferator-activated receptor gamma gene associated with large intestine polyp formation and dyslipidemia". Biochimica et Biophysica Acta 1802 (6): 572–81. PMID 20123124. doi:10.1016/j.bbadis.2010.01.012.
- ↑ McKusick VA (xullo de 1991). "The defect in Marfan syndrome". Nature 352 (6333): 279–81. Bibcode:1991Natur.352..279M. PMID 1852198. doi:10.1038/352279a0.
- ↑ Judge DP, Biery NJ, Keene DR, Geubtner J, Myers L, Huso DL, Sakai LY, Dietz HC (xullo de 2004). "Evidence for a critical contribution of haploinsufficiency in the complex pathogenesis of Marfan syndrome". The Journal of Clinical Investigation 114 (2): 172–81. PMC 449744. PMID 15254584. doi:10.1172/JCI20641.
- ↑ Judge DP, Dietz HC (decembro de 2005). "Marfan's syndrome". Lancet 366 (9501): 1965–76. PMC 1513064. PMID 16325700. doi:10.1016/S0140-6736(05)67789-6.
- ↑ Ellis NA, Ciocci S, German J (febreiro de 2001). "Back mutation can produce phenotype reversion in Bloom syndrome somatic cells". Human Genetics 108 (2): 167–73. PMID 11281456. doi:10.1007/s004390000447.
- ↑ Charlesworth D, Charlesworth B, Morgan MT (decembro de 1995). "The pattern of neutral molecular variation under the background selection model". Genetics 141 (4): 1619–32. PMC 1206892. PMID 8601499.
- ↑ Loewe L (abril de 2006). "Quantifying the genomic decay paradox due to Muller's ratchet in human mitochondrial DNA". Genetical Research 87 (2): 133–59. PMID 16709275. doi:10.1017/S0016672306008123.
- ↑ Bernstein H, Hopf FA, Michod RE (1987). The molecular basis of the evolution of sex. Advances in Genetics 24. pp. 323–70. ISBN 9780120176243. PMID 3324702. doi:10.1016/s0065-2660(08)60012-7.
- ↑ Peck JR, Barreau G, Heath SC (abril de 1997). "Imperfect genes, Fisherian mutation and the evolution of sex". Genetics 145 (4): 1171–99. PMC 1207886. PMID 9093868.
- ↑ Simcikova D, Heneberg P (decembro de 2019). "Refinement of evolutionary medicine predictions based on clinical evidence for the manifestations of Mendelian diseases". Scientific Reports 9 (1): 18577. Bibcode:2019NatSR...918577S. PMC 6901466. PMID 31819097. doi:10.1038/s41598-019-54976-4.
- ↑ Keightley PD, Lynch M (marzo de 2003). "Toward a realistic model of mutations affecting fitness". Evolution; International Journal of Organic Evolution 57 (3): 683–5; discussion 686–9. JSTOR 3094781. PMID 12703958. doi:10.1554/0014-3820(2003)057[0683:tarmom]2.0.co;2.
- ↑ Barton NH, Keightley PD (xaneiro de 2002). "Understanding quantitative genetic variation". Nature Reviews Genetics 3 (1): 11–21. PMID 11823787. doi:10.1038/nrg700.
- ↑ 62,0 62,1 62,2 Sanjuán R, Moya A, Elena SF (xuño de 2004). "The distribution of fitness effects caused by single-nucleotide substitutions in an RNA virus". Proceedings of the National Academy of Sciences of the United States of America 101 (22): 8396–401. Bibcode:2004PNAS..101.8396S. PMC 420405. PMID 15159545. doi:10.1073/pnas.0400146101.
- ↑ Carrasco P, de la Iglesia F, Elena SF (decembro de 2007). "Distribution of fitness and virulence effects caused by single-nucleotide substitutions in Tobacco Etch virus". Journal of Virology 81 (23): 12979–84. PMC 2169111. PMID 17898073. doi:10.1128/JVI.00524-07.
- ↑ Sanjuán R (xuño de 2010). "Mutational fitness effects in RNA and single-stranded DNA viruses: common patterns revealed by site-directed mutagenesis studies". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 365 (1548): 1975–82. PMC 2880115. PMID 20478892. doi:10.1098/rstb.2010.0063.
- ↑ Peris JB, Davis P, Cuevas JM, Nebot MR, Sanjuán R (xuño de 2010). "Distribution of fitness effects caused by single-nucleotide substitutions in bacteriophage f1". Genetics 185 (2): 603–9. PMC 2881140. PMID 20382832. doi:10.1534/genetics.110.115162.
- ↑ Elena SF, Ekunwe L, Hajela N, Oden SA, Lenski RE (marzo de 1998). "Distribution of fitness effects caused by random insertion mutations in Escherichia coli". Genetica. 102–103 (1–6): 349–58. PMID 9720287. doi:10.1023/A:1017031008316.
- ↑ 67,0 67,1 Hietpas RT, Jensen JD, Bolon DN (maio de 2011). "Experimental illumination of a fitness landscape". Proceedings of the National Academy of Sciences of the United States of America 108 (19): 7896–901. Bibcode:2011PNAS..108.7896H. PMC 3093508. PMID 21464309. doi:10.1073/pnas.1016024108.
- ↑ Davies EK, Peters AD, Keightley PD (setembro de 1999). "High frequency of cryptic deleterious mutations in Caenorhabditis elegans". Science 285 (5434): 1748–51. PMID 10481013. doi:10.1126/science.285.5434.1748.
- ↑ Loewe L, Charlesworth B (setembro de 2006). "Inferring the distribution of mutational effects on fitness in Drosophila". Biology Letters 2 (3): 426–30. PMC 1686194. PMID 17148422. doi:10.1098/rsbl.2006.0481.
- ↑ Eyre-Walker A, Woolfit M, Phelps T (xuño de 2006). "The distribution of fitness effects of new deleterious amino acid mutations in humans". Genetics 173 (2): 891–900. PMC 1526495. PMID 16547091. doi:10.1534/genetics.106.057570.
- ↑ Sawyer SA, Kulathinal RJ, Bustamante CD, Hartl DL (agosto de 2003). "Bayesian analysis suggests that most amino acid replacements in Drosophila are driven by positive selection". Journal of Molecular Evolution. 57 Suppl 1 (1): S154–64. Bibcode:2003JMolE..57S.154S. PMID 15008412. doi:10.1007/s00239-003-0022-3.
- ↑ Piganeau G, Eyre-Walker A (setembro de 2003). "Estimating the distribution of fitness effects from DNA sequence data: implications for the molecular clock". Proceedings of the National Academy of Sciences of the United States of America 100 (18): 10335–40. Bibcode:2003PNAS..10010335P. PMC 193562. PMID 12925735. doi:10.1073/pnas.1833064100.
- ↑ Kimura M (febreiro de 1968). "Evolutionary rate at the molecular level". Nature 217 (5129): 624–6. Bibcode:1968Natur.217..624K. PMID 5637732. doi:10.1038/217624a0.
- ↑ Akashi H (setembro de 1999). "Within- and between-species DNA sequence variation and the 'footprint' of natural selection". Gene 238 (1): 39–51. PMID 10570982. doi:10.1016/S0378-1119(99)00294-2.
- ↑ Eyre-Walker A (outubro de 2006). "The genomic rate of adaptive evolution". Trends in Ecology & Evolution 21 (10): 569–75. PMID 16820244. doi:10.1016/j.tree.2006.06.015.
- ↑ Gillespie JH (setembro de 1984). "Molecular Evolution Over the Mutational Landscape". Evolution 38 (5): 1116–1129. JSTOR 2408444. PMID 28555784. doi:10.2307/2408444.
- ↑ Orr HA (abril de 2003). "The distribution of fitness effects among beneficial mutations". Genetics 163 (4): 1519–26. PMC 1462510. PMID 12702694.
- ↑ Kassen R, Bataillon T (abril de 2006). "Distribution of fitness effects among beneficial mutations before selection in experimental populations of bacteria". Nature Genetics 38 (4): 484–8. PMID 16550173. doi:10.1038/ng1751.
- ↑ Rokyta DR, Joyce P, Caudle SB, Wichman HA (abril de 2005). "An empirical test of the mutational landscape model of adaptation using a single-stranded DNA virus". Nature Genetics 37 (4): 441–4. PMID 15778707. doi:10.1038/ng1535.
- ↑ Imhof M, Schlotterer C (xaneiro de 2001). "Fitness effects of advantageous mutations in evolving Escherichia coli populations". Proceedings of the National Academy of Sciences of the United States of America 98 (3): 1113–7. Bibcode:2001PNAS...98.1113I. PMC 14717. PMID 11158603. doi:10.1073/pnas.98.3.1113.
- ↑ "Somatic cell genetic mutation". Genome Dictionary. Athens, Greece: Information Technology Associates. 30 de xuño de 2007. Arquivado dende o orixinal o 24 de febreiro de 2010. Consultado o 2010-06-06.
- ↑ "RB1 Genetics". Daisy's Eye Cancer Fund. Oxford, UK. Arquivado dende o orixinal o 26 de novembro de 2011. Consultado o 2015-10-09.
- ↑ "Compound heterozygote". MedTerms. New York: WebMD. 14 de xuño de 2012. Arquivado dende o orixinal o 4 de marzo de 2016. Consultado o 2015-10-09.
- ↑ Alberts (2014). Molecular Biology of the Cell (6 ed.). Garland Science. p. 487. ISBN 9780815344322.
- ↑ 85,0 85,1 Chadov BF, Fedorova NB, Chadova EV (2015-07-01). "Conditional mutations in Drosophila melanogaster: On the occasion of the 150th anniversary of G. Mendel's report in Brünn". Mutation Research/Reviews in Mutation Research 765: 40–55. PMID 26281767. doi:10.1016/j.mrrev.2015.06.001.
- ↑ 86,0 86,1 Landis G, Bhole D, Lu L, Tower J (xullo de 2001). "High-frequency generation of conditional mutations affecting Drosophila melanogaster development and life span". Genetics 158 (3): 1167–76. PMC 1461716. PMID 11454765. Arquivado dende o orixinal o 22 de marzo de 2017.
- ↑ 87,0 87,1 87,2 87,3 Gierut JJ, Jacks TE, Haigis KM (abril de 2014). "Strategies to achieve conditional gene mutation in mice". Cold Spring Harbor Protocols 2014 (4): 339–49. PMC 4142476. PMID 24692485. doi:10.1101/pdb.top069807.
- ↑ Spencer DM (maio de 1996). "Creating conditional mutations in mammals". Trends in Genetics 12 (5): 181–7. PMID 8984733. doi:10.1016/0168-9525(96)10013-5.
- ↑ Tan G, Chen M, Foote C, Tan C (setembro de 2009). "Temperature-sensitive mutations made easy: generating conditional mutations by using temperature-sensitive inteins that function within different temperature ranges". Genetics 183 (1): 13–22. PMC 2746138. PMID 19596904. doi:10.1534/genetics.109.104794.
- ↑ den Dunnen JT, Antonarakis SE (xaneiro de 2000). "Mutation nomenclature extensions and suggestions to describe complex mutations: a discussion". Human Mutation 15 (1): 7–12. PMID 10612815. doi:10.1002/(SICI)1098-1004(200001)15:1<7::AID-HUMU4>3.0.CO;2-N.
- ↑ Doniger SW, Kim HS, Swain D, Corcuera D, Williams M, Yang SP, Fay JC (agosto de 2008). Pritchard JK, ed. "A catalog of neutral and deleterious polymorphism in yeast". PLOS Genetics 4 (8): e1000183. PMC 2515631. PMID 18769710. doi:10.1371/journal.pgen.1000183.
- ↑ Ionov Y, Peinado MA, Malkhosyan S, Shibata D, Perucho M (xuño de 1993). "Ubiquitous somatic mutations in simple repeated sequences reveal a new mechanism for colonic carcinogenesis". Nature 363 (6429): 558–61. Bibcode:1993Natur.363..558I. PMID 8505985. doi:10.1038/363558a0.
- ↑ Araten DJ, Golde DW, Zhang RH, Thaler HT, Gargiulo L, Notaro R, Luzzatto L (setembro de 2005). "A quantitative measurement of the human somatic mutation rate". Cancer Research 65 (18): 8111–7. PMID 16166284. doi:10.1158/0008-5472.CAN-04-1198.
- ↑ "'Lifeless' prion proteins are 'capable of evolution'". Health. BBC News Online (London). 1 de xaneiro de 2010. Arquivado dende o orixinal o 25 de setembro de 2015. Consultado o 2015-10-10.
- ↑ Sullivan AD, Wigginton J, Kirschner D (agosto de 2001). "The coreceptor mutation CCR5Delta32 influences the dynamics of HIV epidemics and is selected for by HIV". Proceedings of the National Academy of Sciences of the United States of America 98 (18): 10214–9. Bibcode:2001PNAS...9810214S. PMC 56941. PMID 11517319. doi:10.1073/pnas.181325198.
- ↑ "Mystery of the Black Death". Secrets of the Dead. Tempada 3. Episodio 2. 30 de outubro de 2002. PBS. Arquivado dende o orixinal o 12 de outubro de 2015. Consultado o 2015-10-10.
- ↑ Galvani AP, Slatkin M (decembro de 2003). "Evaluating plague and smallpox as historical selective pressures for the CCR5-Delta 32 HIV-resistance allele". Proceedings of the National Academy of Sciences of the United States of America 100 (25): 15276–9. Bibcode:2003PNAS..10015276G. PMC 299980. PMID 14645720. doi:10.1073/pnas.2435085100.
- ↑ Konotey-Ahulu, Felix. "Frequently Asked Questions [FAQ's]". sicklecell.md. Arquivado dende o orixinal o 30 de abril de 2011.
- ↑ Hughes D, Andersson DI (setembro de 2017). "Evolutionary Trajectories to Antibiotic Resistance". Annual Review of Microbiology 71: 579–596. PMID 28697667. doi:10.1146/annurev-micro-090816-093813.
- ↑ Ségurel L, Bon C (agosto de 2017). "On the Evolution of Lactase Persistence in Humans". Annual Review of Genomics and Human Genetics 18: 297–319. PMID 28426286. doi:10.1146/annurev-genom-091416-035340.
- ↑ 101,0 101,1 101,2 Bowler, Peter J. (1992) [1983]. The Eclipse of Darwinism. p. 198.
- ↑ Smocovitis, Vassiliki Betty (1996). Unifying Biology: The Evolutionary Synthesis and Evolutionary Biology. Journal of the History of Biology 25 (Princeton, NJ: Princeton University Press). p. 56. ISBN 978-0-691-03343-3. LCCN 96005605. OCLC 34411399. PMID 11623198. doi:10.1007/bf01947504.
- ↑ Hallgrímsson, Benedikt; Hall, Brian K. (2011). Variation: A Central Concept in Biology. Academic Press. p. 18.
- ↑ Sewall Wright. (1984). Evolution and the Genetics of Populations: Genetics and Biometric Foundations Volume 1. University of Chicago Press. p. 10
- ↑ De Vries, Hugo (1905). Species and Varieties: Their Origin by Mutation.
- ↑ Bateson, William (1894). Materials for the Study of Variation, Treated with Especial Regard to Discontinuity in the Origin of Species.
- ↑ Punnett, Reginald C. (1915). Mimicry in Butterflies. Cambridge University Press.
- ↑ Mayr, Ernst (2007). What Makes Biology Unique?: Considerations on the Autonomy of a Scientific Discipline. Cambridge University Press.
- ↑ 109,0 109,1 Provine, W. B. (2001). The Origins of Theoretical Population Genetics, with a new afterword. University of Chicago Press, Chicago. pp. 56–107.
- ↑ 110,0 110,1 Stoltzfus, A.; Cable, K. (2014). "Mendelian-Mutationism: The Forgotten Evolutionary Synthesis". J Hist Biol 47 (4): 501–546. PMID 24811736. doi:10.1007/s10739-014-9383-2.
- ↑ Hull, D. L. (1985). "Darwinism as an historical entity: A historiographic proposal". En Kohn, D. The Darwinian Heritage. Princeton University Press. pp. 773–812.
- ↑ Yule, G. Udny (1902). "Mendel's Laws and their probable relations to inter-racial heredity". New Phytologist 1 (10): 226–227. doi:10.1111/j.1469-8137.1902.tb07336.x.
- ↑ Gould, S. J. (1982). "The uses of heresey; an introduction to Richard Goldschmidt's The Material Basis of Evolution." pp. xiii-xlii. Yale University Press.
- ↑ Ruse, Michael (1996). Monad to man: the Concept of Progress in Evolutionary Biology. Harvard University Press. pp. 412–413. ISBN 978-0-674-03248-4.
- ↑ A. Stoltzfus (2014). "In search of mutation-driven evolution". Evolution & Development 16: 57–59. doi:10.1111/ede.12062.
- ↑ Futuyma, Douglas J. (2015). Serrelli, E.; Gontier, N., eds. Can Modern Evolutionary Theory Explain Macroevolution? (PDF). Macroevolution (Springer). pp. 29–85. Arquivado dende o orixinal (PDF) o 23 de febreiro de 2020. Consultado o 22 de abril de 2020.
Véxase tamén
[editar | editar a fonte]| Wikimedia Commons ten máis contidos multimedia na categoría: Mutación |
Outros artigos
[editar | editar a fonte]- Paramutación
- Aneuploidía
- Antioxidante
- Deleción
- Ecoxenética
- Embrioloxía
- Homeobox
- Poliploidía
- Translocación robertsoniana
- Mutaxénese etiquetada con sinatura
- Hipermutación somática
- Expansión de repeticións de trinucleótidos
Ligazóns exrternas
[editar | editar a fonte]- Jones S, Woolfson A, Partridge L (6 de decembro de 2007). "Genetic Mutation". In Our Time. BBC Radio 4. Consultado o 2015-10-18.
- Liou, Stephanie (5 de febreiro de 2011). "All About Mutations". HOPES. Huntington's Disease Outreach Project for Education at Stanford. Consultado o 2015-10-18.
- "Locus Specific Mutation Databases". Leiden, the Netherlands: Leiden University Medical Center. Consultado o 2015-10-18.
- "Welcome to the Mutalyzer website". Leiden, the Netherlands: Leiden University Medical Center. Consultado o 2015-10-18. — A páxina web de Mutalyzer.
