Vibrio cholerae
| Vibrio cholerae | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
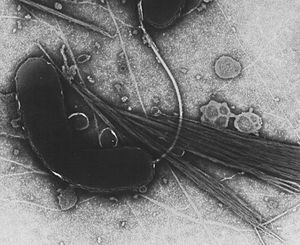 Imaxe de microscopio electrónico de transmisión de V. cholerae | |||||||||||||||
| Clasificación científica | |||||||||||||||
| |||||||||||||||
| Nome binomial | |||||||||||||||
| 'Vibrio cholerae' Pacini 1854 | |||||||||||||||
Vibrio cholerae é unha especie de bacterias gramnegativas, con forma de coma. Algunhas cepas de V. cholerae son patóxenas e causan o cólera. V. cholerae é un organismos anaerobio facultativo e ten un flaxelo nun polo da célula. V. cholerae foi illado por primeira vez en pacientes con cólera polo anatomista italiano Filippo Pacini en 1854, pero este descubrimento non foi coñecido amplamente ata que Robert Koch, 30 anos despois, demostrou que era o axente causante do cólera.[1][2]
Patoxénese[editar | editar a fonte]
Os xenes de patoxenicidade de V. cholerae codifican as proteínas que directa ou indirectamente están implicadas na virulencia da bacteria. Durante a infección, V. cholerae segrega a toxina colérica, unha proteína que causa unha diarrea acuosa profusa. A colonización do intestino delgado tamén require o pilus (TCP) corregulado pola toxina, un delgado e flexible filamento que colga da superficie da célula bacteriana.
A toxina colérica consta de seis subunidades de dous tipos: 1 subunidade A e 5 copias da subunidade B. A toxina únese polas súas subunidades B aos gangliósidos GM1 da superficie da célula, e seguidamente é endocitada, e despois de pasar por varios orgánulos, actívase e acaba afectando indirectamente ás canles de cloro dos enterocitos, o que orixina a diarrea.
Cultivo[editar | editar a fonte]
Cultívase xeralmente en ágar TCBS a partir de mostras fecais, e orixina colonias amarelas doadamente visibles sobre o fondo verde escuro do ágar. O ágar TCBS contén tiosulfato, citrato, sales biliares e sacarosa. Fanse probas bioquímicas para a oxidase (positiva) entre outras.
Xenoma[editar | editar a fonte]
V. cholerae ten dous cromosomas circulares, que en total constitúen un xenoma de 4 millóns de pares de bases e 3.885 xenes preditos.[3] Os xenes da toxína do cólera pertencen en realidade ao bacteriófago temperado CTXfi (CTXφ), e están integrados no xenoma de V. cholerae. O fago CTXφ pode transmitir os xenes da toxina colérica dunha cepa de V. cholerae a outra, o que é unha forma de transferencia horizontal de xenes. Os xenes do pilus corregulado pola toxina están codificados na illa de patoxenicidade VPI, pertencente a outro fago, o VPIφ.
Bacteriófago CTXφ[editar | editar a fonte]
O fago CTXφ é un fago filamentoso que contén os xenes da toxina do cólera. As partículas infecciosas de CTXφ prodúcense cando V. cholerae infecta aos humanos. As partículas do fago fillas saen da célula bacteriana sen que se produza lise. Cando o CTXφ infecta as células de V. cholerae, intégrase en sitios específicos de cada cromosoma. Estes sitios xeralmente conteñen conxuntos en tándem de profagos CTXφ integrados. Ademais dos xenes ctxA e ctxB que codifican a toxina colérica, o CTXφ contén oito xenes implicados na reprodución do fago, empaquetado, secreción, integración, e regulación. O xenoma do CTXφ ten unha lonxitude de 6,9 kb.[4]
Illa de patoxenicidade VPI[editar | editar a fonte]
A illa de patoxenicidade VPI contén xenes implicados na creación dos pilus TCP corregulados pola toxina. Trátase dun longo elemento xenético (de ~40 kb) flanqueado por dúas rexións repetitivas (sitios de tipo att), que lembran en estrutura aos transposóns. Parece ser que procede do fago VPIfi (ou VPIφ). A illa de patoxenicidade VPI contén dous clusters de xenes, chamados TCP e ACF. Na illa de patoxenicidade VPI identificáronse vinte xenes, dos cales algúns están fóra dos clusters, como os xenes tagA, tagB e aldA.[5] O cluster ACF está composto por 4 xenes: acfABC (son tres) e tagE, que codifican un suposto factor de colonización accesorio activado por toxR. O cluster TCP está composto por 15 xenes: tcpABCDEFHIJPQRST (son 14) e o xene regulatorio toxT. A subunidade TcpA do pilus de tipo IV corregulado pola toxina é de feito a proteína da cuberta do fago VPIfi, que funciona como receptor para o outro fago, o CTXfi e como factor de colonización.
Ecoloxía e epidemioloxía[editar | editar a fonte]
Os principais reservorios de V. cholerae son as persoas e os medios acuáticos como as augas salobres e os estuarios, a miúdo en asociación con copépodos ou outros membros do zooplancto, mariscos e plantas acuáticas.
As infeccións de cólera adquírense xeralmente por beber auga na cal se encontra V. cholerae de forma natural ou na cal foi introducida polas feces dunha persoa infectada. Outros vehículos comúns son os peixes e mariscos contaminados ou cereais cocidos sobrantes que non foron debidamente requentados. A transmisión de persoa a persoa, mesmo no persoal sanitario en estreito contacto cos doentes nas epidemias, está documentada moi raramente.
Diversidade e evolución[editar | editar a fonte]
Causan estalidos de cólera dous serogrupos de V. cholerae, o O1 e o O139. O serogrupo 01 causa a maioría das epidemias, mentres que o O139 (identificado por primeira vez en Bangladesh en 1992) está confinado no sueste asiático. Moitos outros serogrupos de V. cholerae, con ou sen o xene da toxina do cólera (e tamén as cepas non toxixénicas dos serogrupos O1 e O139), poden causar unha enfermidade de tipo cólera, pero só as cepas toxixénicas dos serogrupos O1 e O139 poden causar epidemias estendidas de importancia.
O V. cholerae O1 ten dous biotipos, o clásico e o El Tor, e cada un deles ten dous serotipos, o Inaba e o Ogawa. Os síntomas da infección que causan son indistinguibles, aínda que é maior o número de persoas infectadas polo biotipo El Tor que permanecen asintomáticas ou presentan só unha enfermidade leve. Nos últimos anos, as infeccións co biotipo clásico O1 fixéronse raras e están limitadas a partes de Bangladesh e a India.[6] Recentemente, detectáronse novas cepas variantes en varias partes de Asia e África. As observacións suxiren que estas cepas causan un cólera máis grave e cunha taxa máis alta de casos mortais.
Galería[editar | editar a fonte]
-
Vibrio cholerae
-
Esquema da bacteria V. cholerae
Notas[editar | editar a fonte]
- ↑ Bentivoglio, M; Pacini, P (1995). "Filippo Pacini: A determined observer". Brain Research Bulletin 38 (2): 161–5. PMID 7583342. doi:10.1016/0361-9230(95)00083-Q.
- ↑ Howard-Jones, N (1984). "Robert Koch and the cholera vibrio: a centenary". BMJ 288 (6414): 379–81. PMC 1444283. PMID 6419937. doi:10.1136/bmj.288.6414.379.
- ↑ Fraser, Claire M.; Heidelberg, John F.; Eisen, Jonathan A.; Nelson, William C.; Clayton, Rebecca A.; Gwinn, Michelle L.; Dodson, Robert J.; Haft, Daniel H.; Hickey, Erin K. (2000). "DNA sequence of both chromosomes of the cholera pathogen Vibrio cholerae". Nature 406 (6795): 477–83. PMID 10952301. doi:10.1038/35020000.
- ↑ McLeod, S. M.; Kimsey, H. H.; Davis, B. M.; Waldor, M. K. (2005). "CTXφ and Vibrio cholerae: exploring a newly recognized type of phage-host cell relationship". Molecular Microbiology 57: 347–356. PMID 15978069. doi:10.1111/j.1365-2958.04676.x.
- ↑ Karaolis, David K. R.; Somara, Sita; Maneval, David R.; Johnson, Judith A.; Kaper, James B. (1999). "A bacteriophage encoding a pathogenicity island, a type-IV pilus and a phage receptor in cholera bacteria". Nature 399 (6734): 375–9. PMID 10360577. doi:10.1038/20715.
- ↑ Siddique, A.K.; Baqui, A.H.; Eusof, A.; Haider, K.; Hossain, M.A.; Bashir, I.; Zaman, K. (1991). "Survival of classic cholera in Bangladesh". The Lancet 337 (8750): 1125–1127. doi:10.1016/0140-6736(91)92789-5.


