Plásmido
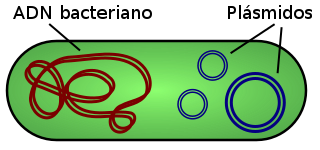
Un plásmido[1][2][3] é unha pequena molécula de ADN que se encontra nunha célula que está separada fisicamente do ADN cromosómico e pode replicarse independentemente. Normalmente trátase de pequenas moléculas de ADN circular bicatenario que se encontran en bacterias, pero ás veces están presentes tamén en arqueas e eucariontes. Na natureza, os plásmidos adoitan portar xenes que poden beneficiar a supervivencia do organismo, por exemplo danlles resistencia aos antibióticos. Aínda que os cromosomas son grandes e conteñen toda a información xenética esencial para vivir en condicións normais, os plásmidos xeralmente son moi pequenos e conteñen só xenes adicionais que poden ser útiles para o organismo en certas situacións ou condicións particulares. Os plásmidos artificiais utilízanse amplamente como vectores en clonación molecular, xa que serven para dirixir a replicación de secuencias de ADN recombinante dentro dos organismos hóspedes. No laboratorio, os plásmidos poden introducirse nunha célula polo mecanismo da transformación.
Os plásmidos considéranse replicóns, unidades de ADN con capacidade de replicación autónoma dentro dun hóspede axeitado. Porén, os plásmidos, igual que os virus, non son xeralmente clasificados como seres vivos.[4] Os plásmidos transmítense desde unha bacteria a outra (incluso doutra especie) principalmente por conxugación.[5] Esta transferencia hóspede a hóspede dun material xenético é un mecanismo de transferencia horizontal de xenes, e os plásmidos son considerados parte do mobiloma. A diferenza dos virus (que encapsulan o seu material xenético nunha cuberta proteica protectora chamada cápside), os plásmidos son ADN "espido" e non codifican os xenes necesarios para encapsular o material xenético para transferilo así a un novo hóspede. Porén, algunhas clases de plásmidos codifican os pili "sexuais" ou da conxugación necearios para a súa propia transferencia. O tamaño do plásmido varía de 1 a uns 200 kbp,[6] e o número de plásmidos idénticos nunha soa célula pode estar entre un e miles baixo certas circunstancias.
As relacións entre microbios e o ADN do plásmido non é nin parasita nin mutualista, porque ambos os casos implicarían a presenza dunha especie independente nun estado prexudicial ou comensal co organismo hóspede. En vez diso,, os plásmidos proporcionan un mecanismo para a transferencia horizontal de xenes dentro dunha poboación de microbios e proporcionan tipicamente unha vantaxe selectiva baixo un estado ambiental dado. Os plásmidos poden portar xenes que proporcionan resistencia a antibióticos presentes de forma natural nun nicho ecolóxico competitivo, ou a proteína producida pode actuar como toxina en circunstancias similares, ou permitir que o organismo utilice un composto orgánico determinado que sería vantaxoso cando escaseasen os nutrientes.[7]
Historia
[editar | editar a fonte]O termo plásmido foi introducido en 1952 polo biólogo molecular norteamericano Joshua Lederberg para referirse a "calquera determinante hereditario extracromosómico."[8] O uso inicial do termo incluía calquera material xenético que exista extracromosomicamente para polo menos parte do seu ciclo de replicación, pero como esa descrición inclúía os virus bacterianos, a noción de plásmido foi sendo refinada co tempo para comprender os elementos xenéticos que se reproducen autonomamente.[9] Posteriormente, en 1968, decidiuse que o termo plásmido debería adoptarse como termo para os elmentos xeneticos extracromosómicos,[10] e para distinguilos dos virus, a definición foi restrinxida a elementos xenéticos que existen exclusivamente ou predominantemente fóra do cromosoma e poden replicarse autonomamente.[9]
Propiedades e características
[editar | editar a fonte]
Para que os plásmidos se repliquen independentemente dentro dunha célula, deben posuír un tramo de ADN que poida actuar como orixe de replicación. A unidade autorreplicante, neste caso o plásmido, chámase replicón. Un replicón bacteriano típico pode consistir en varios elementos, como o xene para a proteína da iniciación da replicación específica do plásmido (Rep), unidades repetidas chamadas iteróns, caixas de DnaA e unha rexión adxacente rica en AT.[9] Os plásmidos máis pequenos fan uso dos encimas replicativos do hóspede para facer copias de si mesmos, mentres que os plásmidos máis grandes poden portar xenes específicos para a súa replicación. Uns poucos tipos de plásmidos poden tamén inserirse no cromosoma do hóspede, e estes plásmidos integrativos son ás veces denominados episomas en procariotas.[11]
Os plásmidos case sempre levan polo menos un xene. Moitos dos xenes que levan os plásmidos son beneficiosos para as células hóspedes, por exemplo: permiten que a célula hóspede sobreviva nun ambiente que doutro modo sería letal ou restritivo para o crecemento. Algúns destes xenes codifican trazos para dar resistencia a antibióticos ou resistencia a metais pesados, mentres que outros poden producir factores de virulencia que permiten que unha bacteria colonice un hóspede e supere as súas defensas, ou teñen funcións metabólicas específicas que permiten que a bacteria utilice un nutriente en particular, incluíndo a capacidade de degradar compostos orgánicos duradeiros ou tóxicos.[9] Os plásmidos poden tamén proporcionar ás bacterias a capacidade de fixar nitróxeno. Algúns plásmidos, porén, non teñen un efecto observable sobre o fenotipo da célula hóspede ou o seu beneficio para a célula hóspede non se pode determinar, e estes plásmidos denomínanse plásmidos crípticos.[12]
Os plásmidos que aparecen na natureza varían moito nas súas propiedades físicas. O seu tamaño pode ir de miniplásmidos moi pequenos de menos de 1 quilopares de bases (Kbp), a megaplásmidos moi grandes de varios pares de megabases (Mbp). No extremo superior do rango de tamaños, pouca diferenza hai entre un megaplásmido e un minicromosoma. Os plásmidos son xeralmente circulares, pero tamén se coñecen exemplos de plásmidos lineares. Estes plásmidos lineares requiren mecanismos especializados para replicar os seus extremos.[9]
Os plásmidos poden estar presentes nunha célula individual en diverso número, desde só un a varios centos. A cantidade normal de copias de plásmidos que se poden encontrar nunha soa célula denomínase número de copias e está determinado por como se regula a iniciación da replicación e o tamaño da molécula. Os plásmidos máis grandes adoitan ter un menor número de copias.[11] Os plásmidos con baixo número de copias que existen en só unha ou unhas poucas copias en cada bacteria están, durante a división celular, en perigo de perderse nunha das bacterias formadas. Ditos plásmidos de copia única teñen sistemas que intentan distribuír activamente unha copia a ambas as células fillas. Estes sistemas, entre os que están o sistema parABS e o sistema parMRC, denomínanse a miúdo sistemas de partición ou funcións de partición dun plásmido.
Clasificacións e tipos
[editar | editar a fonte]


Os plásmidos poden clasificarse de diversos xeitos. Poden clasificarse pola súa capacidade de favorecer a conxugación en dous grandes grupos: plásmidos conxugativos e non conxugativos. Os plásmidos conxugativos conteñen un conxunto de xenes de transferencia ou tra, que promoven a conxugación sexual entre diferentes células.[11] No complexo proceso da conxugación bacteriana, os plásmidos poden transferirse dunha bacteria a outra coa intervención de pili sexuais codificados por algúns dos xenes tra (ver figura).[13] Os plásmidos non conxugativos non poden iniciar a conxugación, polo que só poden ser transferidos coa axuda de plásmidos conxugativos. Unha clase intermedia de plásmidos son mobilizables e portan só un conxunto de xenes que cómpren para a transferencia. Poden parasitar un plásmido conxugativo, e só se transfiren a alta frecuencia na súa presenza.
Os plásmidos poden tamén ser clasificados en grupos de incompatibilidade. Un microbio pode albergar diferentes tipos de plásmidos, pero só poden existir nunha célula bacteriana plásmidos de diferentes tipos se son compatibles. Se dous plásmidos non son compatibles, un deles vaise perder rapidamente da célula. Diferentes plásmidos poden, por tanto, ser asignados a distintos grupos de incompatibilidade dependendo de se poden coexistir na célula ou non. Os plásmidos incompatibles (que pertencen ao mesmo grupo de incompatibilidade) normalmente comparten o mesmo tipo de mecanismos de replicación ou partición e dese modo non poden estar xuntos nunha célula.[14][15]
Outra maneira de clasificar os plásmidos é pola súa función. Con este criterio establécense cinco clases de plásmidos:
- Plásmidos de fertilidade ou plásmidos (ou factores) F, que conteñen xenes tra. Poden realizar a conxugación e teñen como resultado a expresión de pili sexuais. Os plásmidos de fertilidade desempeñan un importante papel na conxugación de E. coli. Ademais de que foi o primeiro deste tipo en ser descrito ten unha lonxitude aproximada de 10 Kb, contén xenes responsables da unión á célula, e da transferencia do plásmido situado entre cepas bacterianas específicas no proceso de conxugación. Gran parte do conxunto da información para a transferencia de plásmidos está situada no operón tra, o cal contén menos de 28 xenes. Estes xenes dirixen a formación de pili sexuais que unen unha célula doadora a unha receptora, outros xenes, ao contrario, colaboran na transferencia de ADN. Tamén conteñen segmentos denominados secuencias de inserción, colaboran na inserción do plásmido no cromosoma e na célula do hóspede, polo que poden existir fóra do cromosoma bacteriano ou estar integrados nel.
- Plásmidos de resistencia (ou plásmidos R), que conteñen xenes que proporcionan resistencia contra antibióticos ou velenos. Coñecidos historicamente como factores R, antes de que se comprendese ben a natureza dos plásmidos. Conteñen xenes que codifican encimas capaces de destruír ou modificar antibióticos, normalmente non están integrados no cromosoma da bacteria que os contén, e encontráronse nestes plásmidos xenes que codifican a resistencia a antibióticos como a ampicilina, o cloranfenicol e a kanamicina, entre outros. Algúns plásmidos R conteñen un só xene de resistencia; outros chegan a ter ata oito. Con frecuencia os genes de resistencia encóntranse nun elemento de transposición de forma que nas cepas bacterianas se poden desenvolver con rapidez plásmidos que codifican resistencias múltiples. Como moitos plásmidos de resistencia son á súa vez plásmidos de conxugación poden propagarse por toda unha poboación aínda que con menor rapidez que os plásmidos de fertilidade. Con frecuencia, os plásmidos ou factores R non conxugativos, pasan dunha bacteria a outra durante a conxugación promovida por outro plásmido; con este método toda unha poboación pode facerse resistente aos antibióticos. De feito o que algúns destes plásmidos se poidan transferir doadamente entre especies, promove aínda máis a propagación de resistencias. Cando o hóspede consome grandes cantidades de antibióticos selecciónanse bacterias con factores R e fanse máis frecuentes; os plásmidos R poden entón ser transferidos a xéneros máis patóxenos como Salmonella, entre outros, e producir maiores problemas de saúde pública.
- Plásmidos Col, que conteñen xenes que codifican bacteriocinas, que son proteínas que poden matar outras bacterias. Proporcionan unha vantaxe competitiva no mundo dos microbios, as bacteriocinas son proteínas que destrúen outras bacterias, poden actuar soamente contra cepas estreitamente relacionadas, ou en ocasións destrúen as células xerando poros na membrana plasmática ou dexenerando a parede celular, provocando deste modo que se incremente a súa permeabilidade, outro proceso para destruír a célula é degradando o ADN e ARN ou atacando o peptidoglicano. Os plásmidos col17 conteñen xenes para a síntese de bacteriocinas coñecidas como colicinas que están dirixidas contra E. coli. Existen plásmidos con características parecidas, as cales conteñen xenes que codifican bacteriocinas dirixidas contra outras especies; por exemplo producen cloacinas que destrúen especies de Enterobacter; o hóspede non se ve afectado polas bacteriocinas que produce. Algúns plásmidos Col son conxugativos e conteñen xenes de resistencia.
- Plásmidos degradativos, que poden facer a dixestión de substancias pouco comúns, por exemplo, o tolueno e o ácido salicílico.
- Plásmidos de virulencia, que converten a bacteria en patóxena.
- Plásmidos metabólicos, que posúen xenes para que algunhas cepas de Rhizobium induzan a nodulación nas legumes e leven a cabo a fixación do nitróxeno.
Os plásmidos poden pertencer a máis dun destes grupos funcionais.
Vectores
[editar | editar a fonte]- Artigo principal: Vector (bioloxía molecular).
Poden utilizarse plásmidos construídos artificialmente como vectores en enxeñaría xenética. Estes plásmidos serven como ferramentas importantes en laboatorios de xenética e biotecnoloxía, nos que se usan habitualmente para clonar e amplificar (facer moitas copias) ou expresar xenes determinados.[16] Disponse comercialmente dunha gran variedade de plásmidos para esas tarefas. O xene que vai ser replicado é normalmente inserido nun plásmido que tipicamente contén diversas características axeitadas ao seu uso. Estes inclúen un xene que confire resistencia a determinados antibióticos (a ampicilina é o usado con máis frecuencia para cepas bacterianas), unha orixe de replicación para permitir que a bacteria replique o ADN do plásmido, e un sitio axeitado para a clonación (é dicir, un sitio de clonación múltiple).
Clonación
[editar | editar a fonte]- Artigo principal: Vector de clonación.
Os plásmidos son os vectores de clonación bacteriana máis comunmente utilizados.[17] Estes vectores de clonación conteñen un sitio que permite inserir os fragmentos de ADN, por exemplo un sitio de clonación múltiple ou un polivinculador (polilinker ) que ten varios sitio de restrición comunmente usados ao cal se poden ligar os fragmentos de ADN. Unha vez que se insire o xene de interese, os plásmidos son introducidos en bacterias por un proceso chamado transformación. Estes plásmidos conteñen un marcador seleccionable, xeralmente un xene de resistencia a antibióticos, que lle dá á bacteria a capacidade de sobrevivir e proliferar nun medio de crecemento selectivo que conteña un determinado antibiótico. As células despois da transformación son expostas a un medio selectivo e só poden sobrevivir as células que conteñan os plásmidos. Deste modo, os antibióticos actúan como un filtro para seleccionar só as bacterias que conteñen o ADN do plásmido. O vector pode tamén conter outros xenes marcadores ou xenes reporteiros para facilitar a selección de plásmidos cunha inserción clonada. As bacterias que conteñen o plásmido poden despois crecer en grandes cantidades, ser colleitadas e o plásmido de interese pode entón illarse usando varios métodos de preparación de plásmidos.
Un vector de clonación de plásmidos utilízase normalmente para clonar fragmentos de ADN de ata 15 kbp.[18] Para clonar fragmentos máis longos de ADN, utilízanse o fago lambda con xenes lisoxénicos eliminados, un cósmido, un cromosoma artificial bacteriano ou un cromosoma artificial de lévedo.
Produción de proteínas
[editar | editar a fonte]- Artigo principal: Vector de expresión.
Outro importante uso dos plásmidos é para sintetizar unha proteína en grandes cantidades. Neste caso, cultívase a bacteria que contén un plásmido que alberga o xene de interese. Igual que a bacteria produce a proteína que lle confire resistencia ao antibiótico, pode tamén ser inducida a producir grandes cantidades doutra proteína a partir do xene inserido. Isto é un modo barato e doado de producir masivamente unha proteína que o xene codifica, por exemplo a insulina.
Terapia xénica
[editar | editar a fonte]- Artigo principal: Vectores en terapia xénica.
Os plásmidos poden tamén usarse para a transferencia de xenes en células humanas como tratamento potencial en terapia xénica para que poidan expresar a proteína que falta nas células. Algunhas estratexias de terapia xénica requiren a inserción de xenes terapéuticos en sitios diana cromosómicos preseleccionados do xenoma humano. Os vectores pláasmidos son unha das moitas aproximacións que se poden utilizar para ese propósito. As nucleases de dedo de zinc (ZFN) ofrecen un modo de causar unha rotura de dobre febra nun sitio específico do ADN do xenoma e causar a recombinación homóloga. Os plásmidos que codifican ZFN poden axudar a entregar un xene terapéutico nun sitio específico, para que se evite o dano celular ou as mutacións que poderían causar cancro ou unha resposta inmune.[19]
Modelos de enfermidades
[editar | editar a fonte]Os plásmidos foron historicamente usados para modificar por enxeñaría xenética células nai embrionarias de ratas para crear modelos de enfemidades xenéticas en ratas. A eficiencia limitada das técnicas baseadas en plásmidos descartaron o seu uso na creación de modelos celulares humanos máis exactos. Porén, os desenvolvementos en técnicas de recombinación en virus adeno-asocidos e as nucleases de dedo de zinc, permitiron a creación dunha nova xeración de modelos de enfermidades humanas isoxénicos.
Episomas
[editar | editar a fonte]O termo episoma[20] foi introducido por François Jacob e Élie Wollman en 1958 para referirse ao material xenético extracromosómico que pode replicarse autonomamente ou ser integrado no cromosoma.[21][22] Porén, desde a introdución do termo, o seu uso foi mudando a medida que plásmido se converteu no temo preferido para o ADN extracromosómico replicante autónomo. Nun simposio de 1968 en Londres algúns participantes suxeriron que o termo episoma fose abandonado, aínda que outros continuaron a usalo cun cambio no significado.[23][24]
Hoxe algúns autores usan episoma no contexto dos procariotas para referirse a un plásmido que pode integrarse no cromosoma. Os plásmidos integrativos poden replicarse e manterse establemente nunha célula ao longo de moitas xeracións, pero sempre nalgunha etapa existen como moléculas de plásmido independentes.[25] No contexto dos eucariotas, o termo episomas utilízase para significar unha molécula de ADN circular pechado extracromosómico non integrado que pode replicarse no núcleo.[26][27] Os virus son os exemplos máis comúns disto, como os herpesvirus, adenovirus e poliomavirus, pero algúns son pásmidos. Outros exemplos inclúen fragmentos cromosómicos aberrantes, como os cromosomas diminutos dobres, que se poden orixinar durante as amplificacións artificiais de xenes ou en procesos patolóxicos (por exemplo, a transformación das células cancerosas). Os episomas en eucariotas compórtanse de maneira similar aos plásmidos de procariotas no sentido de que o ADN é mantido establemente e replicado dentro da célula hóspede. Pode haber tamén episomas virais citoplásmicos (como nas infeccións de poxvirus). Algúns episomas, como os herpesvirus, replícanse polo mecanismo do círculo rodante, similar ao dos virus bacteriófagos. Outros replícanse por un mecanismo de replicación bidireccional (plásmidos do tipo theta). En ambos os casos, os episomas permanecen fisicamente separados dos cromosomas da célula hóspede. Varios virus do cancro, como o virus de Epstein-Barr e herpesvirus asociados ao sarcoma de Kaposi, mantéñense latentes, como episomas cromosomicamente diferenciados en células cancerosas, onde os virus expresan oncoxenes que promoven a proliferación da célula cancerosa. En cancros, estes episomas replícanse pasivamente xunto cos cromosomas do hóspede cando se divide a célula. Cando estes episomas virais inician a replicación lítica para xerar múltiples partículas víricas, en xeral activan os mecanismos de defensa da inmunidade innata celular que matan a célula hóspede.
Mantemento dos plásmidos
[editar | editar a fonte]- Artigo principal: Módulo de adicción.
Algúns plásmidos ou hóspedes microbianos inclúen un sistema de adicción ou sistema de matanza postsegregacional (PSK), como o hok/sok sistema (matador do hóspede/suppresor da matanza) do plásmido R1 en Escherichia coli.[28] Esta variante produce tanto un veleno de vida longa coma un antídoto de vida curta. Na literatura describíronse varios tipos de sistemas de adicción de plásmidos (toxina/ antitoxina, baseados no metabolismo, sistemas ORT)[29] e usáronse en aplicacións biotécnicas (fermentación) ou biomédicas (terapia de vacinas). As células fillas que reteñen unha copia dun plásmido sobrevivirán, mentres que a célula filla que non consegue herdar o plásmido morre ou sofre dunha velocidade de crecemento reducida debido ao veleno de longa duración da célula parental. Finalmente, a produtividade global podería verse potenciada.
A diferenza do anterior, virtualmente todos os plásmidos usados biotecnoloxicamente (como pUC18, pBR322 e vectores derivados) non conteñen sistemas de adicción toxina-antitoxina e así deben manterse baixo a presión de antibióticos para evitar a perda de plásmidos.
Plásmidos de lévedos
[editar | editar a fonte]Os lévedos albergan de forma natural varios plásmidos. Notables entre eles son os plásmidos de 2 µm (pequenos plásmidos circulares usados a miúdo en enxeñaría xenética de lévedos) e os plásmidos lineares pGKL de Kluyveromyces lactis, que son responsables dos fenotipos matadores.[30]
Outros tipos de plásmidos están a miúdo relacionados con vectores de clonación de lévedos entre os que están:
- Plásmido integrador de lévedo (YIp), vectores de lévedo que dependen da integración no cromosoma hóspede para a supervivencia e a replicación, e utilízanse usualmente cando se estuda a funcionalidade dun xene solo ou cando o xene é tóxico. Tamén conectados co xene URA3, que codifica un encima relacionado coa biosíntese de nucleótidos de pirimidina (T, C);
- Plásmido replicativo de lévedos (YRp), que transporta unha secuencia de ADN cromosómico que inclúe unha orixe de replicación. Estes plásmidos son menos estables, dado que poden acabar perdéndose durante a xemación.
Extracción do ADN de plásmidos
[editar | editar a fonte]Os plásmidos utilízanse a miúdo para purificar unha secuencia específica, xa que poden facilmente ser purificados do resto do xenoma. Para o seu uso como vectores e para a clonación molecular, os plásmidos adoitan ter que ser illados.
Hai varios métodos de illar o ADN de plásmidos das bacterias, cuxos arquetipos son o miniprep e o maxiprep/bulkprep.[16] O primeiro pode usarse para descubrir rapidamente se o plásmido é correcto en calquera dos varios clons bacterianos. O rendemento é unha pequena cantidade de ADN de plásmidos impuro, o cal é suficiente para a análise por dixestión de restrición e por algunhas técnicas de clonación.
No último, cultívanse volumes moito maiores de suspensión bacteriana a partir do cal se pode realizar unha maxi-prep. En esencia, esta é unha miniprep ampliada seguida dunha purificación adicional. Isto ten como resultado cantidades relativamente grandes (varios centos de microgramos) de ADN de plásmido moi puro.
En tempos recentes, creáronse moitos kits comerciais para realizar a extracción do plásmido a varias escalas, purezas e niveis de automatización. Os servizos comerciais poden preparar ADN de plásmido a prezos por debaixo de 300 dólares o miligramo en cantidades de miligramos e de 15 dólares o miligramo en cantidades de gramos (en 2007).
Conformacións
[editar | editar a fonte]O ADN de plásmidos pode aparecer en cinco conformacións posibles, as cales (para un tamaño dado) avanzan a diferentes velocidades no xel durante a electroforese. As conformacións están listadas máis abaixo segundo a súa velocidade electroforética (velocidade para unha voltaxe determinada aplicada), de máis lenta a máis rápida:
- ADN circular aberto con amosega, que ten unha febra cortada.
- ADN circular relaxado, no que o ADN está completamente intacto con ambas as febras sen cortar, pero foi encimaticamente relaxado (eliminados os superenrolamentos). Isto pode ser modelado ou comparado con deixar que un cable eléctrico enrolado se desenrole e relaxe e despois conectándoo consigo mesmo.
- ADN linear, que ten extremos libres, sexa porque ambas as febras foron cortadas ou porque o ADN era linear in vivo. Isto pode ser comparado cun cable eléctrico que non está conectada consigo mesmo.
- ADN superenrolado (ou circular pechado covalentemente), que está completamente intacto con ambas as febras sen cortar e cun enlolamento integral, que orixina unha forma compacta. Isto pode ser comparado con enrolar un cable eléctrico e despois conectalo consigo mesmo.
- ADN superenrolado desnaturalizado é como o ADN superenrolado, pero ten rexións desemparelladas que o fan lixeiramene menos compacto; isto pode causar unha excesiva alcalinidade durante a preparación do plásmido.
A velocidade de migración para pequenos fragmentos lineares é directamente proporcional á voltaxe aplicada a baixas voltaxes. A voltaxes maiores, os fragmentos máis grandes migran a velocidades diferentes aínda que se incrementan constantemente. Así, a resolución dun xel diminúe co incremento da voltaxe.
A unha voltaxe baixa especificada, a velocidade de migración de fragmentos de ADN lineares pequenos está en función da súa lonxitude. Os fragmentos lineares máis grandes (dunhas 20 kb) migran a unha certa velocidade fixa sen importar a súa lonxitude. Isto é así porque as moléculas avanzan coa masa da molécla seguindo o extremo en cabeza a través da matriz do xel. As dixestións de restrición son utilizadas frecuentemente para analizar plásmidos purificados. Estes encimas rompen especificamente o ADN en certas secuencias curtas. Os fragmentos lineares resultantes forman 'bandas' ao facer unha elecrroforese en xel. É posible purificar certos fragmentos cortando as bandas e sacándoas do xel e disolvendo o xel para que se liberen os fragmentos de ADN.
Debido á súa conformación apertada, o ADN sperenrolado migra máis rápido a través do xel que o linear ou circular aberto.
Software para bioinformática e deseños
[editar | editar a fonte]O uso de plásmidos como técnica de bioloxía molecular faise utilizando software de bioinformática. Estes programas rexistran a secuencia de ADN de vectores plásmidos, axudan a predicir sitios de corte de encimas de restrición, e a planificar manipulacións. Exemplos de paquetes de software que manexan mapas de plásmidos son ApE, Clone Manager, GeneConstructionKit, Geneious, Genome Compiler, LabGenius, Lasergene, MacVector, pDraw32, Serial Cloner, VectorFriends, Vector NTI e WebDSV. Estes softwares axudan a conducir experimentos completos no computador (in silico) antes de facelo con experimentos en húmido.[31]
Coleccións de plásmidos
[editar | editar a fonte]Ao longo dos anos creáronse moitos plásmidos e os investigadores depositáronos en bases de datos de plásmidos como as organizacións sen ánimo de lucro Addgene e BCCM/LMBP. Pódense buscar e pedir plásmidos desas bases de datos para continuar as investigacións. Os investigadores cargaron tamén secuencias de plásmidos na base de datos do NCBI. Usando a base de datos do NCBI, poden buscarse secuencias de plásmidos específicos.
Notas
[editar | editar a fonte]- ↑ Coordinadores: Jaime Gómez Márquez, Ana Mª Viñas Díaz e Manuel González González. Redactores: David Villar Docampo e Luís Vale Ferreira. Revisores lingüísticos: Víctor Fresco e Mª Liliana Martínez Calvo. (2010). Dicionario de bioloxía galego-castelán-inglés. (PDF). Xunta de Galicia. p. 137. ISBN 978-84-453-4973-1.
- ↑ Dicionario Digalego plásmido
- ↑ * Real Academia de Medicina e Cirurxía de Galicia- Diccionario Galego de termos médicos-2001.
- ↑ Sinkovics, J; Harvath J; Horak A. (1998). "The Origin and evolution of viruses (a review)". Acta Microbiologica et Immunologica Hungarica 45 (3–4): 349–90. PMID 9873943.
- ↑ Smillie, Chris; Garcillán-Barcia, M. Pilar; Francia, M. Victoria; Rocha, Eduardo P. C.; Cruz, Fernando de la (1 de setembro de 2010). "Mobility of Plasmids". Microbiology and Molecular Biology Reviews (en inglés) 74 (3): 434–452. ISSN 1092-2172. PMC 2937521. doi:10.1128/MMBR.00020-10.
- ↑ Thomas, Christopher M; Summers, David (2008). "Bacterial Plasmids". Encyclopedia of Life Sciences. ISBN 0470016175. doi:10.1002/9780470015902.a0000468.pub2.
- ↑ Wolfgang Schumann (2008). "Chapter 1 - Escherichia coli Cloning and Expression Vectors". En Georg Lipps. Plasmids: Current Research and Future Trends. Caister Academic Press. pp. 1–2. ISBN 978-1-904455-35-6.
- ↑ Lederberg J (1952). "Cell genetics and hereditary symbiosis". Physiol. Rev. 32 (4): 403–430. PMID 13003535.
- ↑ 9,0 9,1 9,2 9,3 9,4 Finbarr Hayes (2003). "Chapter 1 - The Function and Organization of Plasmids". En Nicola Casali, Andrew Presto. E. Coli Plasmid Vectors: Methods and Applications. Methods in Molecular Biology 235. Humana Press. pp. 1–5. ISBN 978-1-58829-151-6.
- ↑ Stanley Falkow. "Microbial Genomics: Standing on the Shoulders of Giants". Microbiology Society.
- ↑ 11,0 11,1 11,2 T. A. Brown (2010). "Chapter 2 - Vectors for Gene Cloning: Plasmids and Bacteriophages". Gene Cloning and DNA Analysis: An Introduction (6th ed.). Wiley-Blackwell. ISBN 978-1405181730.
- ↑ David Summers (1996). "Chapter 1 - The Function and Organization of Plasmids". The Biology of Plasmids. Wiley-Blackwell; First Edition. pp. 21–22. ISBN 978-0632034369.
- ↑ David P. Clark; Nanette Jean Pazdernik (2012). Molecular Biology (2nd ed.). Academic Cell. p. 795. ISBN 978-0123785947.
- ↑ Margaret C. M. Smith and R. Elizabeth Sockett, ed. (1999). Genetic Methods for Diverse Prokaryotes. Methods in Microbiology, vol. 29. Academic Press. pp. 75–77. ISBN 0-12-652340-1.
- ↑ Morgan, Kendall. "Plasmids 101: Origin of Replication". addgene.org.
- ↑ 16,0 16,1 Russell, David W.; Sambrook, Joseph (2001). Molecular cloning: a laboratory manual. Cold Spring Harbor, N.Y: Cold Spring Harbor Laboratory.
- ↑ Uldis N. Streips, Ronald E. Yasbin, ed. (2002). Modern Microbial Genetics (2nd ed.). Wiley-Blackwell. p. 248. ISBN 978-0471386650.
- ↑ Andrew Preston (2003). "Chapter 2 - Choosing a Cloning Vector". En Nicola Casali, Andrew Preston. E. Coli Plasmid Vectors: Methods and Applications. Methods in Molecular Biology, Vol. 235. Humana Press. pp. 19–26. ISBN 978-1-58829-151-6.
- ↑ Kandavelou K, Chandrasegaran S (2008). "Plasmids for Gene Therapy". Plasmids: Current Research and Future Trends. Caister Academic Press. ISBN 978-1-904455-35-6.
- ↑ Dicionario Digalego episoma Arquivado 15 de decembro de 2018 en Wayback Machine.
- ↑ Morange M (2009). "What history tells us XIX. The notion of the episome" (PDF). Journal of Biosciences 34 (6): 845–8. PMID 20093737. doi:10.1007/s12038-009-0098-z. (require subscrición (?)).
- ↑ Jacob F & Wollman EL (1958). "Les épisomes, elements génétiques ajoutés". Comptes Rendus de l'Académie des Sciences de Paris 247 (1): 154–156. PMID 13561654.
- ↑ Hayes, W (1969). "What are episomes and plasmids?". En Gordon E. W. Wolstenholme; Maeve O'Connor. Bacterial Episomes and Plasmids (CIBA Foundation Symposium). pp. 4–8. ISBN 978-0700014057.
- ↑ Gordon E. W. Wolstenholme; Maeve O'Connor, eds. (1969). Bacterial Episomes and Plasmids (CIBA Foundation Symposium). pp. 244–245. ISBN 978-0700014057.
- ↑ T. A. Brown (2011). Introduction to Genetics: A Molecular Approach. Garland Science. p. 238. ISBN 978-0815365099.
- ↑ Kathleen Van Craenenbroeck, Peter Vanhoenacker and Guy Haegeman (2000). "Episomal vectors for gene expression in mammalian cells". Eur. J. Biochem. 267 (18): 5665–5678. PMID 10971576. doi:10.1046/j.1432-1327.2000.01645.x.
- ↑ Colosimo A1, Goncz KK, Holmes AR, Kunzelmann K, Novelli G, Malone RW, Bennett MJ, Gruenert DC. (2000). "Transfer and expression of foreign genes in mammalian cells" (PDF). Biotechniques 29 (2): 314–8, 320–2, 324 passim. PMID 10948433. Arquivado dende o orixinal (PDF) o 24 de xullo de 2011. Consultado o 12 de decembro de 2018.
- ↑ Gerdes K, Rasmussen PB, Molin S (1986). "Unique type of plasmid maintenance function: postsegregational killing of plasmid-free cells". Proc. Natl. Acad. Sci. U.S.A. 83 (10): 3116–20. Bibcode:1986PNAS...83.3116G. PMC 323463. PMID 3517851. doi:10.1073/pnas.83.10.3116.
- ↑ Kroll J, Klinter S, Schneider C, Voß I, Steinbüchel A (2010). "Plasmid addiction systems: perspectives and applications in biotechnology". Microb. Biotechnol. 3 (6): 634–657. PMC 3815339. PMID 21255361. doi:10.1111/j.1751-7915.2010.00170.x.
- ↑ Gunge, N; Murata, K; Sakaguchi, K (July 1982). "Transformation of Saccharomyces cerevisiae with linear DNA killer plasmids from Kluyveromyces lactis". Journal of Bacteriology 151 (1): 462–4. PMC 220260. PMID 7045080.
- ↑ "Vector NTI feedback video". The DNA Lab.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]- Cromosoma artificial bacteriano
- Bacteriófago
- Provirus
- Segrosoma
- Transposón
- Apareamento triparental
- Plasmidoma
- ADN recombinante
Bibliografía
[editar | editar a fonte]- Klein, Donald W.; Prescott, Lansing M.; Harley, John (1999). Microbiology. Boston: WCB/McGraw-Hill.
- Smith, Christopher U. M. (2002). Elements of Molecular Neurobiology. Wiley. pp. 101, 111.
- Albert G. Moat; John W. Foster; Michael P. Spector (2002). Microbial Physiology. Wiley-Liss. ISBN 0-471-39483-1.
- Brock Biology of Microorganisms - Madigan, Martinko Parker -2003
Episomas
[editar | editar a fonte]- Piechaczek C, Fetzer C, Baiker A, Bode J, Lipps HJ (1999). "A vector based on the SV40 origin of replication and chromosomal S/MARs replicates episomally in CHO cells". Nucleic Acids Res 27 (2): 426–428. PMC 148196. PMID 9862961. doi:10.1093/nar/27.2.426.
- Bode J; Fetzer CP; Nehlsen K; Scinteie M; Hinrichsen B-H; Baiker A; Piechazcek C; Benham C; Lipps HJ (2001). "The Hitchhiking principle: Optimizing episomal vectors for the use in gene therapy and biotechnology" (PDF). Gene Ther Mol Biol 6: 33–46. Arquivado dende o orixinal (PDF) o 30 de maio de 2009. Consultado o 12 de decembro de 2018.
- Nehlsen K, Broll S, Bode J (2006). "Replicating minicircles: Generation of nonviral episomes for the efficient modification of dividing cells" (PDF). Gene Ther Mol Biol 10: 233–244. Arquivado dende o orixinal (PDF) o 30 de maio de 2009. Consultado o 12 de decembro de 2018.
- Ehrhardt A, Haase R, Schepers A, Deutsch MJ, Lipps HJ, Baiker A (2008). "Episomal vectors for gene therapy". Curr Gene Ther 8 (3): 147–161. PMID 18537590. doi:10.2174/156652308784746440. Arquivado dende o orixinal o 26 de setembro de 2011. Consultado o 12 de decembro de 2018.
- Argyros O, Wong SP, Niceta M, Waddington SN, Howe SJ, Coutelle C, Miller AD, Harbottle RP (2008). "Persistent episomal transgene expression in liver following delivery of a scaffold/matrix attachment region containing non-viral vector". Gene Therapy 15 (24): 1593–1605. PMID 18633447. doi:10.1038/gt.2008.113.
- Wong SP, Argyros O, Coutelle C, Harbottle RP (2009). "Strategies for the episomal modification of cells". Current Opinion in Molecular Therapeutics 11 (4): 433–441. PMID 19649988. Arquivado dende o orixinal o 17 de setembro de 2011. Consultado o 12 de decembro de 2018.
- Haase R, Argyros O, Wong SP, Harbottle RP, Lipps HJ, Ogris M, Magnusson T, Vizoso Pinto MG, Haas J, Baiker A (2010). "pEPito: a significantly improved non-viral episomal expression vector for mammalian cells" (PDF). BMC Biotechnol 10: 433–441. doi:10.1186/1472-6750-10-20.
Ligazóns externas
[editar | editar a fonte]- International Society for Plasmid Biology and other Mobile Genetic Elements
- Historia dos plásmidos con liña de tempo
