ARN termómetro
Un ARN termómetro ou termómetro de ARN, tamén chamado ARN termosensor (ou termosensor de ARN) é unha molécula de ARN non codificante sensible á temperatura que regula a expresión xénica. Os termómetros de ARN adoitan regular xenes que actúan durante a resposta a un choque térmico (quente) ou un choque frío, pero foron tamén implicados noutras tarefas regulatorias como na patoxenicidade e a inanición.[1] Atópanse principalmente en bacterias, pero non exclusivamente.
En xeral os termómetros de ARN funcionan cambiando a súa estrutura secundaria en resposta ás flutuacións da temperatura. Esta transición estrutural pode entón expoñer ou agochar importantes rexións do ARN como un sitio de unión ao ribosoma, o que así afecta a taxa de tradución de proteínas codificadas nun xene próximo.
Os termómetros de ARN, xunto cos riboswitches, póñense como exemplos que apoian a hipótese do mundo de ARN. Esta teoría propón que o ARN foi a inicios da orixe da vida o único ácido nucleico presente nas primeiras células, e máis tarde foi substituído polo actual sistema ADN → ARN → proteína.[2]
Exemplos de termómetros de ARN son: FourU,[3] o elemento regulatorio en cis Hsp90,[4] o elemento ROSE,[5] o termómetro ARN lig,[6] e o termómetro Hsp17.[7]
Descubrimento[editar | editar a fonte]
O primeiro elemento de ARN sensible á temperatura foi descuberto en 1989.[8] Antes deste descubrimento, sabíase que as mutacións augas arriba do sitio de inicio da transcrición nun ARNm cIII de fago lambda (λ) afectaban o nivel de tradución da proteína cIII.[9] Esta proteína está implicada na selección dos cilos de vida lítico ou lisoxénico no fago λ, na que as altas concentracións de cIII promovían a lisoxenia.[9] Estudos posteriores desta rexión do ARN augas arriba identificaron que podía existir en dúas estrutura secundarias alternativas, que cambiaban entre si e eran dependentes da concentración de ión magnesio e da temperatura.[8][10] Agora pénsase que este termómetro de ARN favorece a entrada no ciclo lítico baixo unha situación de estrés de calor para que o bacteriófago se replique rapidamente e escape da célula hóspede.[1]
O termo "termómetro de ARN" (RNA thermometer) non se acuñou ata 1999,[11] cando foi aplicado ao elemento de ARN rpoH identificable en Escherichia coli.[12] Máis recentemente, empregáronse investigacións bioinformáticas para descubrir novos posibles termómetros de ARN.[13] As investigacións tradicionais baseadas na secuencia non son eficaces para isto, xa que a estrutura secundaria do elemento está moito máis conservada que a secuencia do ácido nucleico.[13]
Distribución[editar | editar a fonte]
A maioría dos termómetros de ARN coñecidos están localizados na rexión non traducida 5′ (UTR) dos ARNm que codifican proteínas de choque térmico, aínda que se suxeriu que isto se podía deber, en parte, a un nesgo de mostraxe e ás dificultades inherentes á detección de secuencias de ARN non conservadas e curtas nos datos xenómicos.[14][15]
Aínda que se atoparon predominantemente en procariotas, tamén se atopou un potencial termómetro de ARN en mamíferos, incluíndo os humanos e noutros animais.[16] O termosensor candidato ARN de choque de calor 1 (HSR1) activa o factor de transcrición de choque térmico 1 (HSF1) e induce proteínas protectoreas cando a temperatura da célula excede os 37 °C (a temperatura normal do corpo humano), o que impide o sobrequentamento das células.[16]
Estrutura[editar | editar a fonte]

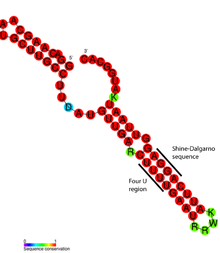
Os termómetros de ARN son estruturalmente simples e poden estar formados por secuencias de ARN curtas; as menores de só 44 nucleótidos e atópanse no ARNm da proteína de choque térmico hsp17, en Synechocystis sp. PCC 6803.[18][19] Xeralmente estes elementos de ARN varían en lonxitude desde 60 a 110 nucleótidos[20] e conteñen tipicamente unha forquita cun pequeno número de pares de bases desapareadas que reducen a estabilidade da estrutura, permitindo así un despregamento máis doado en resposta ao incremento de temperatura.[21]
Unha análise estrutural detallada do termómetro de ARN ROSE revelou que as bases desapareadas están en realidade ligadas entre si formando apareamentos non estándar que conservan a estrutura helicoidal do ARN (ver figura). Os apareamentos de bases non habituais consisten en pares G-G, U-U e UC-Us. Como estes pares de bases non canónicos son relativamente inestables, un incremento de temperatura causa a 'fusión' local da estrutura do ARN nesta rexión (rompen as pontes de H entre as bases), expoñendo a secuencia Shine-Dalgarno.[17]
Algúns termómetros do ADN son significativamente máis complexos que unha simple forquita, como no caso dunha rexión atopada no ARNm de CspA que se pensa contén un pseudonó xunto con múltiples forquitas.[22][23]
Deseñáronse termómetros de ARN sintéticos cunha soa estrutura de forquita simple.[24] Porén, a estrutura secundaria de tales termómetros de ARN curtos pode ser sensible á mutación, xa que un só cambio de base pode facer que a forquita quede inactiva in vivo.[25]
Mecanismo[editar | editar a fonte]

Os termómetros de ARN encóntranse na 5′-UTR do ARN mensaxeiro, augas arriba dun xene que codifica proteínas.[1] Alí poden agochar o sitio de unión ao ribosoma (RBS) e impiden a tradución do ARNm a proteínas.[14] A medida que a temperatura aumenta, a estrutura da forquita pode 'fundirse' e expón o RBS ou a secuencia Shine-Dalgarno para permitir a unión da subunidade ribosómica menor (30S), que despois ensambla outra maquinaria de tradución necesaria.[1] O codón de iniciación, que normalmente se encontra a 8 nucleótidos augas abaixo da secuencia Shine-Dalgarno,[14] sinaliza o comezo dun xene codificante de proteínas, que é despois traducido a un polipéptido polo ribosoma. Ademais diste mecanismo que actúa en cis, atopouse tamén un único termómetro de ARN que actúa en trans no ARNm RpoS, que se pensa que está implicado na resposta á inanición.[1]
Un exemplo específico dun motivo termómetro de ARN é o termómetro FourU de Salmonella enterica.[3] Cando está exposto a temperaturas por riba de 45 °C, o talo-bucle que establece emparellamentos de bases fronte á secuencia Shine-Dalgarno queda desemparellada e isto permite que o ARNm entre no ribosoma para que teña lugar a tradución.[25] A concentración de ión Mg2+ tamén afecta á estabilidade de FourU.[26] O termómetro de ARN mellor estudado encóntrase no xene rpoH de Escherichia coli.[27] Este termosensor regula á alza as proteínas de choque térmico a temperaturas maiores por medio de σ32, un factor sigma de choque térmico especializado.[11]
Aínda que os termómetros de ARN están tipicamente asociados coa expresión de proteínas inducida por calor, poden tamén regular as proteínas de choque frío.[22] Por exemplo, a expresión de dúas proteínas de 7 kDa é regulada por un termómetro de ARN na bacteria termófila Thermus thermophilus[28] e identificouse un mecanismo similar en Enterobacteriales.[23]
Os termómetros de ARN sensibles a temperaturas de 37 °C poden ser utilizadas por patóxenos para activar xenes específicos de infeccións.[14] Por exemplo, a regulación á alza de prfA, que codifica un regulador transcricional clave de xenes de virulencia en Listeria monocytogenes, foi demostrada ao fusionar o ADN 5′ de prfA co xene da proteína fluorescente verde; o xene de fusión foi despois transcrito desde o promotor T7 en E. coli, e observouse fluorescencia a 37 °C pero non a 30 °C.[29]
Implicacións para a hipótese do mundo de ARN[editar | editar a fonte]
- Artigo principal: Hipótese do mundo de ARN.
A hipótese do mundo de ARN afirma que o ARN foi nos inicios da aparición da vida na Terra tanto o portador da información hereditaria coma unha molécula activa encimaticamente, cuxas diferentes secuencias actuaban como biocatalizadores, reguladores e sensores.[30] A hipótese propoñía que despois evolucionou a vida moderna baseada no ADN, ARN e proteínas e a selección substituíu a maioría das funcións antes exercidas polo ARN polas exercidas por outras moléculas.[2]
Pénsase que os termómetros de ARN e os riboswitches son evolutivamente antigos debido á súa distribución ampla en organismos moi distanciados taxonomicamente.[31] Propúxose que no mundo de ARN, os termosensores de ARN serían os responsables da regulación dependente da temperatura doutras moléculas de ARN.[2][32] Os termómetros de ARN nos organismos modernos poden ser fósiles moleculares que poderían insinuar a súa importancia previa máis xeneralizada no mundo de ARN.[2]
Outros exemplos[editar | editar a fonte]
- O elemento regulador en cis Hsp90, que regula a hsp90 en Drosophila, incrementando a taxa de tradución das proteínas de choque térmico a altas temperaturas.[4]
- O operón ibpAB de E. coli predise que contén dous termómetros de ARN cooperativos: un elemento ROSE e o termómetro IbpB.[33]
- Os ROSE1 e ROSEAT2 encóntranse nos hifomicrobiais Bradyrhizobium japonicum e Agrobacterium tumefaciens, respectivamente. Existen no 5′ UTR do ARNm de HspA e reprimen a tradución de proteínas de choque térmico a temperaturas fisiolóxicas.[5][34]
- Os termómetros de ARN cianobacterianos.
- O termómetro de ARN interxénico.
- Os termómetros de ARN de Neisseria.
- O termómetro de ARN lig.
Notas[editar | editar a fonte]
- ↑ 1,0 1,1 1,2 1,3 1,4 1,5 Narberhaus F, Waldminghaus T, Chowdhury S (xaneiro de 2006). "RNA thermometers". FEMS Microbiol. Rev. 30 (1): 3–16. PMID 16438677. doi:10.1111/j.1574-6976.2005.004.x.
- ↑ 2,0 2,1 2,2 2,3 Atkins JF, Gesteland RF, Cech T (2006). The RNA world: the nature of modern RNA suggests a prebiotic RNA world. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-739-6.
- ↑ 3,0 3,1 Waldminghaus T, Heidrich N, Brantl S, Narberhaus F (xullo de 2007). "FourU: a novel type of RNA thermometer in Salmonella". Mol. Microbiol. 65 (2): 413–424. PMID 17630972. doi:10.1111/j.1365-2958.2007.05794.x.
- ↑ 4,0 4,1 Ahmed R, Duncan RF (2004). "Translational regulation of Hsp90 mRNA. AUG-proximal 5′-untranslated region elements essential for preferential heat shock translation". J Biol Chem 279 (48): 49919–49930. PMID 15347681. doi:10.1074/jbc.M404681200.
- ↑ 5,0 5,1 Nocker A, Hausherr T, Balsiger S, Krstulovic NP, Hennecke H, Narberhaus F (2001). "A mRNA-based thermosensor controls expression of rhizobial heat shock genes". Nucleic Acids Res 29 (23): 4800–4807. PMC 96696. PMID 11726689. doi:10.1093/nar/29.23.4800.
- ↑ Matsunaga J, Schlax PJ, Haake DA (2013-11-15). "Role for cis-Acting RNA Sequences in the Temperature-Dependent Expression of the Multiadhesive Lig Proteins in Leptospira interrogans". Journal of Bacteriology (en inglés) 195 (22): 5092–5101. ISSN 0021-9193. PMC 3811586. PMID 24013626. doi:10.1128/jb.00663-13.
- ↑ Kortmann J, Sczodrok S, Rinnenthal J, Schwalbe H, Narberhaus F (2011). "Translation on demand by a simple RNA-based thermosensor.". Nucleic Acids Res 39 (7): 2855–2868. PMC 3074152. PMID 21131278. doi:10.1093/nar/gkq1252.
- ↑ 8,0 8,1 Altuvia S, Kornitzer D, Teff D, Oppenheim AB (1989-11-20). "Alternative mRNA structures of the cIII gene of bacteriophage lambda determine the rate of its translation initiation". Journal of Molecular Biology 210 (2): 265–280. PMID 2532257. doi:10.1016/0022-2836(89)90329-X.
- ↑ 9,0 9,1 Altuvia S, Oppenheim AB (xullo de 1986). "Translational regulatory signals within the coding region of the bacteriophage lambda cIII gene". Journal of Bacteriology 167 (1): 415–419. PMC 212897. PMID 2941413. doi:10.1128/jb.167.1.415-419.1986.
- ↑ Altuvia S, Kornitzer D, Kobi S, Oppenheim AB (1991-04-20). "Functional and structural elements of the mRNA of the cIII gene of bacteriophage lambda". Journal of Molecular Biology 218 (4): 723–733. PMID 1827163. doi:10.1016/0022-2836(91)90261-4.
- ↑ 11,0 11,1 Storz G (1999-03-15). "An RNA thermometer". Genes & Development 13 (6): 633–636. PMID 10090718. doi:10.1101/gad.13.6.633.
- ↑ Morita MT, Tanaka Y, Kodama TS, Kyogoku Y, Yanagi H, Yura T (1999-03-15). "Translational induction of heat shock transcription factor sigma32: evidence for a built-in RNA thermosensor". Genes & Development 13 (6): 655–665. PMC 316556. PMID 10090722. doi:10.1101/gad.13.6.655.
- ↑ 13,0 13,1 Waldminghaus T, Gaubig LC, Narberhaus F (novembro de 2007). "Genome-wide bioinformatic prediction and experimental evaluation of potential RNA thermometers". Molecular Genetics and Genomics 278 (5): 555–564. PMID 17647020. doi:10.1007/s00438-007-0272-7.
- ↑ 14,0 14,1 14,2 14,3 Narberhaus F (2010). "Translational control of bacterial heat shock and virulence genes by temperature-sensing mRNAs". RNA Biol 7 (1): 84–89. PMID 20009504. doi:10.4161/rna.7.1.10501. Consultado o 2011-04-23.
- ↑ Johansson J (2009). "RNA thermosensors in bacterial pathogens". Bacterial Sensing and Signaling. Contrib Microbiol. Contributions to Microbiology 16 (Basel). pp. 150–160. ISBN 978-3-8055-9132-4. PMID 19494584. doi:10.1159/000219378.
- ↑ 16,0 16,1 Shamovsky I, Ivannikov M, Kandel ES, Gershon D, Nudler E (marzo de 2006). "RNA-mediated response to heat shock in mammalian cells". Nature 440 (7083): 556–560. Bibcode:2006Natur.440..556S. PMID 16554823. doi:10.1038/nature04518.
- ↑ 17,0 17,1 Chowdhury S, Maris C, Allain FH, Narberhaus F (2006-06-07). "Molecular basis for temperature sensing by an RNA thermometer". The EMBO Journal 25 (11): 2487–2497. PMC 1478195. PMID 16710302. doi:10.1038/sj.emboj.7601128.
- ↑ Kortmann J, Sczodrok S, Rinnenthal J, Schwalbe H, Narberhaus F (abril de 2011). "Translation on demand by a simple RNA-based thermosensor.". Nucleic Acids Research 39 (7): 2855–2868. PMC 3074152. PMID 21131278. doi:10.1093/nar/gkq1252.
- ↑ Kortmann J, Sczodrok S, Rinnenthal J, Schwalbe H, Narberhaus F (abril de 2011). "Translation on demand by a simple RNA-based thermosensor". Nucleic Acids Res. 39 (7): 2855–2868. PMC 3074152. PMID 21131278. doi:10.1093/nar/gkq1252.
- ↑ Waldminghaus T, Fippinger A, Alfsmann J, Narberhaus F (decembro de 2005). "RNA thermometers are common in alpha- and gamma-proteobacteria". Biol. Chem. 386 (12): 1279–1286. PMID 16336122. doi:10.1515/BC.2005.145.
- ↑ Narberhaus F (xaneiro-febreiro de 2010). "Translational control of bacterial heat shock and virulence genes by temperature-sensing mRNAs.". RNA Biology 7 (1): 84–89. PMID 20009504. doi:10.4161/rna.7.1.10501.
- ↑ 22,0 22,1 Breaker RR (xaneiro de 2010). "RNA switches out in the cold". Mol. Cell 37 (1): 1–2. PMC 5315359. PMID 20129048. doi:10.1016/j.molcel.2009.12.032.
- ↑ 23,0 23,1 Giuliodori AM, Di Pietro F, Marzi S, Masquida B, Wagner R, Romby P, Gualerzi CO, Pon CL (xaneiro de 2010). "The cspA mRNA is a thermosensor that modulates translation of the cold-shock protein CspA". Mol. Cell 37 (1): 21–33. PMID 20129052. doi:10.1016/j.molcel.2009.11.033.
- ↑ Neupert J, Karcher D, Bock R (novembro de 2008). "Design of simple synthetic RNA thermometers for temperature-controlled gene expression in Escherichia coli". Nucleic Acids Research 36 (19): e124. PMC 2577334. PMID 18753148. doi:10.1093/nar/gkn545.
- ↑ 25,0 25,1 Nikolova EN, Al-Hashimi HM (setembro de 2010). "Thermodynamics of RNA melting, one base pair at a time". RNA 16 (9): 1687–1691. PMC 2924531. PMID 20660079. doi:10.1261/rna.2235010.
- ↑ Rinnenthal J, Klinkert B, Narberhaus F, Schwalbe H (2011-07-04). "Modulation of the stability of the Salmonella fourU-type RNA thermometer". Nucleic Acids Research 39 (18): 8258–8270. PMC 3185406. PMID 21727085. doi:10.1093/nar/gkr314.
- ↑ Shah P, Gilchrist MA (2010). Spirin AS, ed. "Is thermosensing property of RNA thermometers unique?". PLOS ONE 5 (7): e11308. Bibcode:2010PLoSO...511308S. PMC 2896394. PMID 20625392. doi:10.1371/journal.pone.0011308.
- ↑ Mega R, Manzoku M, Shinkai A, Nakagawa N, Kuramitsu S, Masui R (agosto de 2010). "Very rapid induction of a cold shock protein by temperature downshift in Thermus thermophilus". Biochem. Biophys. Res. Commun. 399 (3): 336–340. PMID 20655297. doi:10.1016/j.bbrc.2010.07.065.
- ↑ Johansson J, Mandin P, Renzoni A, Chiaruttini C, Springer M, Cossart P (setembro de 2002). "An RNA thermosensor controls expression of virulence genes in Listeria monocytogenes". Cell 110 (5): 551–561. PMID 12230973. doi:10.1016/S0092-8674(02)00905-4.
- ↑ Gilbert W (febreiro de 1986). "The RNA World". Nature 319 (6055). p. 618. Bibcode:1986Natur.319..618G. doi:10.1038/319618a0.
- ↑ Serganov A, Patel DJ (outubro de 2007). "Ribozymes, riboswitches and beyond: regulation of gene expression without proteins". Nature Reviews Genetics 8 (10): 776–790. PMC 4689321. PMID 17846637. doi:10.1038/nrg2172.
- ↑ Bocobza SE, Aharoni A (outubro de 2008). "Switching the light on plant riboswitches". Trends in Plant Science 13 (10): 526–533. PMID 18778966. doi:10.1016/j.tplants.2008.07.004.
- ↑ Gaubig LC, Waldminghaus T, Narberhaus F (xaneiro de 2011). "Multiple layers of control govern expression of the Escherichia coli ibpAB heat-shock operon". Microbiology 157 (Pt 1): 66–76. PMID 20864473. doi:10.1099/mic.0.043802-0.
- ↑ Balsiger S, Ragaz C, Baron C, Narberhaus F (2004). "Replicon-specific regulation of small heat shock genes in Agrobacterium tumefaciens". J Bacteriol 186 (20): 6824–6829. PMC 522190. PMID 15466035. doi:10.1128/JB.186.20.6824-6829.2004.
