Piridoxal fosfato
| Piridoxal fosfato | |
|---|---|
 | |
 | |
(4-formil-5-hidroxi-6-metilpiridin-3-il)metil fosfato | |
Outros nomes Piridoxal 5-fosfato, PAL-P, PLP, Vitamina B6 fosfato | |
| Identificadores | |
| Número CAS | 54-47-7 |
| PubChem | 1051 |
| MeSH | Pyridoxal+Phosphate |
| ChEBI | CHEBI:18405 |
| ChEMBL | CHEMBL82202 |
| Ligando IUPHAR | 5249 |
| Imaxes 3D Jmol | Image 1 |
| |
| Propiedades | |
| Fórmula molecular | C8H10NO6P |
| Masa molecular | 247,142 g/mol |
| Densidade | 1,638±0,06 g/cm3[1] |
| Punto de fusión | 139–142 °C; 282–288 °F; 412–415 K |
| Acidez (pKa) | 1,56[1] |
| Perigosidade | |
| Punto de inflamabilidade | 296,0±32,9 °C[1] |
Se non se indica outra cousa, os datos están tomados en condicións estándar de 25 °C e 100 kPa. | |
O piridoxal fosfato, tamén chamado PLP, piridoxal 5'-fosfato, P5P ou fosfato de piridoxal, é un grupo prostético ligado a certos encimas. O PLP é tamén a forma activa da vitamina B6, a cal comprende tres compostos naturais, piridoxal, piridoxamina e piridoxina. Funciona como un cofactor que actúa como un sumidoiro de electróns para estabilizar os intermediarios carbaniónicos tanto nas reaccións de eliminación coma de substitución que implican compostos aminados. A Comisión de encimas catalogou máis de 140 actividades dependentes do PLP, que corresponden ao ~4% de todas as actividades clasificadas.[2]
Papel como coencima
[editar | editar a fonte]O PLP actúa como coencima en todas as reaccións de transaminación, e en certas reaccións de descarboxilación, desaminación e racemización de aminoácidos. O grupo aldehido do PLP forma unha estrutura tipo base de Schiff (unha aldimina interna) co grupo ε-amino dunha lisina situado no sitio activo da aminotransferase. O grupo α-amino do aminoácido substrato despraza o grupo ε-amino da lisina situada no sitio activo nun proceso coñecido como "transaldiminación". A aldimina externa resultante pode perder un protón, dióxido de carbono, ou a cadea lateral do aminoácido para formar un intermediario quinoide, o cal pode ademais actuar como nucleófilo en numerosas rutas de reacción.
Nas transaminacións, despois da desprotonación, o intermediario quinoide acepta un protón nunha posición diferente para converterse nunha cetimina. A cetimina resultante hidrolízase de forma que o grupo amino permanece no complexo.[3] Adicionalmente, o PLP é utilizado polas aminotransferases (tamén chamadas transaminases) que actúan sobre algúns azucres pouco comúns, como a perosamina e a desosamina.[4] Nestas reaccións, o PLP combínase con glutamato, o cal transfire o seu grupo alfa-amino ao PLP para formar piridoxamina fosfato (PMP). Logo este PMP transfire o seu nitróxeno ao azucre, formando un aminoazucre.
O PLP tamén se encontra envolvido en varias reaccións de eliminación tales como as que levan a cabo a serina deshidratase e a GDP-4-ceto-6-desoximanosa-3-deshidratase (Co1D).[4]
Tamén participa na reacción de condensación que ten lugar na síntese do hemo.
O PLP, tamén desempeña unha función na conversión da levodopa en dopamina, facilitando a transformación do neurotransmisor excitatorio glutamato no neurotransmisor inhibitorio GABA, e permite que a SAM poida descarboxilarse para converterse en propilamina, a cal é unha precursora das poliaminas.
Exemplos non clásicos de PLP
[editar | editar a fonte]O PLP tamén se pode encontrar na glicóxeno fosforilase no fígado, onde se utiliza para degradar o glicóxeno no proceso de glicoxenólise cando o glicagón ou a adrenalina sinalizan a necesidade de levar a cabo a reacción. Porén, este encima non aproveita a reactividade do grupo aldehido, senón que usa o grupo fosfato do PLP para realizar esta reacción.
Malia que a gran maioría dos encimas dependentes de PLP forman unha aldimina interna co PLP por medio dun residuo lisina no sitio activo, algúns encimas dependentes do PLP non posúen este residuo lisina, senón que no seu lugar teñen unha histidina no sitio activo. Nestes casos, a histidina non pode formar a aldimina interna, e, por tanto, o cofactor non queda covalentemente unido ao encima. Un exemplo deste tipo de encimas é a GDP-4-ceto-6-desoximanosa-3-deshidratase (ColD).[5]
Mecanismo catalítico
[editar | editar a fonte]Os encimas dependentes de piridoxal 5'-fosfato (encimas PLP) catalizan un milleiro de reaccións bioquímicas. Aínda que nunha primeira ollada as reaccións catalizadas por PLP parecen ser enormemente diversas, comparten unha característica bastante simple: no estado inactivo, o cofactor (PLP) está unido covalentemente ao grupo amino dunha lisina presente no sitio activo, formando unha aldimina interna. Cando o substrato amino interacciona co sitio activo, fórmase unha nova base de Schiff, que adoita denominarse aldimina externa. Despois deste paso, a vía mecanística para cada unha das reacciones catalizadas por PLP diverxe. Aplicáronse métodos de densidade funcional para investigar a reacción de transiminación e o resultado demostrou que a reacción comprende tres pasos secuenciais:
- Formación dun intermediario tetraédrico coa lisina do sitio activo e o substrato amino unidos ao cofactor PLP.
- Transferencia indirecta dun protón entre o substrato amino e o residuo de lisina.
- Formación dunha aldimina externa logo da disociación do residuo de lisina.
A reacción total é exotérmica (-12 Kcal/mol), e o paso limitante da velocidade é o segundo, cunha enerxía de activación de 12,6 kcal/mol.[6]

Especificidade
[editar | editar a fonte]A especificidade débese a que dos catro enlaces do carbono alfa no estado aldimina aminoácido, o que rompe é o enlace perpendicular ao anel de piridina (hipótese estereoelectrónica de Dunathan).[7] En consecuencia, a especificidade está causada pola maneira en como o encima se liga aos seus substratos. A facilidade coa que o nitróxeno do anel de piridina se protona xoga un papel adicional na especificidade.[7]
Encimas PLP
[editar | editar a fonte]O PLP está fixado ao sitio activo non só grazas á lisina, senón tamén grazas á interacción do grupo fosfato co peto de unión do fosfato, e de forma menos intensa, grazas ao amontoamento de bases (base stacking) que se produce entre o anel de piridina e un residuo aromático colgante, xeralmente unha tirosina (o cal tamén intervén no mecanismo de catálise ácido-base). A pesar dos escasos requirimentos necesarios para que se forme o peto de unión de PLP, os encimas PLP pertencen só a cinco familias. Estas familias non gardan unha boa correlación cun tipo particular de reacción. As cinco familias clasifícanse con números romanos de acordo co seu tipo de pregamento.[7]
- Pregamento Tipo I — familia da aspartato aminotransferase
- Pregamento Tipo II — familia da triptófano sintase
- Pregamento Tipo III — familia da alanina racemase (barril TIM)
- Pregamento Tipo IV — familia da D-aminoácido aminotransferase
- Pregamento Tipo V — familia da glicóxeno fosforilase
Biosíntese
[editar | editar a fonte]A partir de vitámeros
[editar | editar a fonte]Os animais son auxótrofos para este cofactor encimático, e necesitan del ou dun intermediario como suplemento, de aí a súa clasificación como vitamina (a B
6); a diferenza do que ocorre, por exemplo, co cofactor de molibdeno ou o coencima CoQ10. O PLP sintetízase a partir de piridoxal por acción dun encima chamado piridoxal quinase, que necesita unha molécula de ATP. O metabolismo do PLP ten lugar no fígado.
Prototrofia
[editar | editar a fonte]Coñécense actualmente dúas vías metabólicas naturais para a síntese de PLP: unha require desoxixilulosa 5-fosfato (DXP) e a outra non; por tanto, denomínanse vía dependente do DXP e vía independente do DXP. Estas vías metabólicas foron estudadas intensamente en Escherichia coli e Bacillus subtilis, respectivamente. As dúas vías, a pesar da súa diferenza en canto aos compostos iniciais e ao distinto número de pasos que requiren, teñen moitas semellanzas.[8]
Biosíntese dependente de DXP
[editar | editar a fonte]Para a síntese dependente de DXP cómpren varios pasos e a converxencia de dúas ramas metabólicas subsidiarias, unha que produce 3-hidroxi-1-aminoacetona fosfato a partir de eritrosa 4-fosfato, e outra que produce deoxixilulosa 5-fosfato (DXP) a partir de gliceraldehido 3-fosfato (GAP) e piruvato. A condensación do 3-hidroxi-1-aminoacetona fosfato e deoxixilulosa 5-fosfato produce piridoxina 5-fosfato. A condensación é catalizada pola PNP sintase, encima codificado polo xene pdxJ.[9] O paso final é catalizado por unha PNP oxidase (pdxH), a cal cataliza a oxidación do grupo OH 4', para formar un aldehido utilizando unha molécula de oxíxeno diatómico e producindo peróxido de hidróxeno como subproduto.
En E. coli a primeira rama da vía é catalizada polos encimas codificados polos xenes epd, pdxB, serC e pdxA. Esta vía comparte similitudes mecanísticas e homoloxía cos tres encimas da síntese da serina (serA, homóloga de pdxB; serC, serB; epd é un homólogo de gap), o cal apunta a unha orixe evolutiva compartida entre ambas as vías metabólicas.[10] En varias especies hai dous homólogos do xene serC de E. coli, xeralmente un no operón ser, e outro no operón pdx, e nese caso é denominado pdxF.
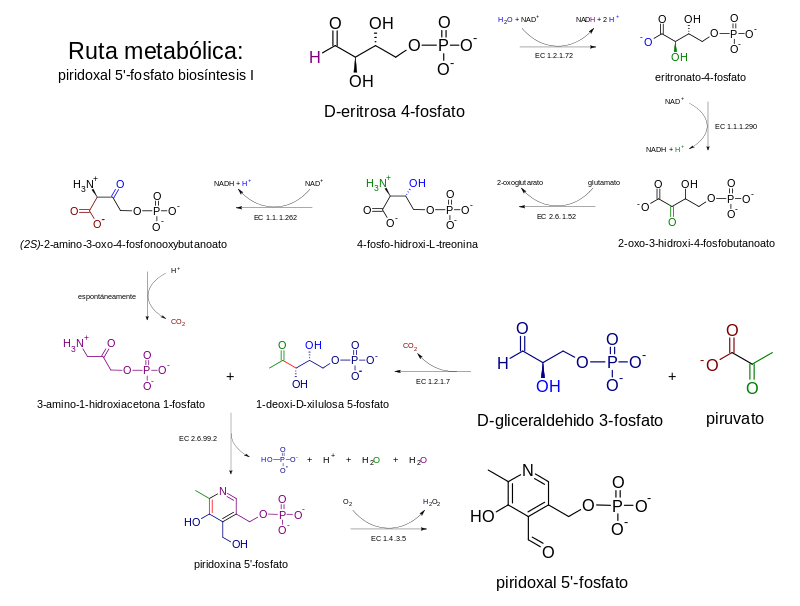
Encontrouse tamén unha vía serendípica en E. coli nunha biblioteca de sobreexpresión, que pode suprimir a auxotrofia causada pola deleción de pdxB (o xene que codifica o encima eritronato 4 fosfato deshidroxenase). Esta vía accidental é moi ineficiente, pero é posible grazas á promiscuidade encimática de varios encimas. Esta vía comeza con 3-fosfohidroxipiruvato (3PHP) (o produto do xene serA que participa na biosíntese da serina) e non require de eritronato 4-fosfato. O 3PHP desfosforílase, producindo un composto inestable que se descarboxila espontaneamente (de alí a presenza de fosfato na síntese de serina), para formar glicaldehido. O glicaldehido condénsase con glicina e o produto fosforilado é a 4-fosfohidroxitreonina (4PHT), o substrato canónico do encima 4-PHT deshidroxenase (pdxA).[8]
Biosíntese independente de DXP
[editar | editar a fonte]A ruta biosintética de PLP independente de DXP consta dun único paso catalizado pola PLP-sintase, un encima composto por dúas subunidades: PdxS e PdxT. A subunidade PdxS cataliza a condensación de ribulosa 5-fosfato, gliceraldehido 3-fosfato e amonio para producir PLP. A subunidade PdxT produce o amonio necesario para a condensación a partir de glutamina. A PdxS é un barril (β/α)8 (coñecido tamén como barril TIM), que forma un dodecámero.[11]
Síntese prebiótica
[editar | editar a fonte]A ampla utilización do PLP no metabolismo central, especialmente na biosíntese de aminoácidos e a súa actividade en ausencia de encimas, suxiren que o PLP podería ter sido un composto prebiótico.[12] De feito, quentando NH
3 e glicoaldehido, fórmanse espontaneamente varios tipos de piridinas, incluíndo o piridoxal.[12] Baixo certas condicións, o PLP pode formarse a partir de cianoacetileno, diacetileno, monóxido de carbono, hidróxeno, auga e ácido fosfórico.[13]
Inhibidores
[editar | editar a fonte]Coñécense varios inhibidores dos encimas PLP.
Un tipo de inhibidor é o que forma un electrófilo co PLP, causando que reaccione irreversiblemente coa lisina do sitio activo. Exemplos deste tipo de inhibidores son os compostos acetilénicos (por exemplo a proparxilglicina) e vinílicos (por exemplo vinilglicina). Outro tipo de inhibidores son substratos análogos α-metil e amino-oxi, por exemplo o α-metilglutamato). Finalmente, outro grupo de inhibidores son aqueles que sendo bos grupos saíntes exercen un ataque nucleofílico sobre o PLP, tales como a clororanilina, a cal inhibe un gran número de encimas.[7]
Exemplos de inhibidores:
- A AlaP (alanina fosfonato) inihibe as alanina racemases (ALR), mais carece de especificidade, o que levou ao deseño de novos inhibidores da ALR.[14]
- A gabaculina e vigabatrina inhiben a GABA aminotransferase.
- A canalina e 5-fluorometilornitina inhiben a ornitina aminotransferase .
- A amino-oxi SAM inhibe a 1-aminociclopropano-1-carboxilato sintase (ACC sintase).
Notas
[editar | editar a fonte]- ↑ 1,0 1,1 1,2 Calculado usando Advanced Chemistry Development (ACD/Labs) Software V11.02 (© 1994-2011 ACD/Labs)
- ↑ Percudani R1, Peracchi A. (2003). "Genomic overview of pyridoxal-phosphate-dependent enzymes". EMBO Rep. 4 (9): 850–4. PMC 1326353. PMID 12949584. doi:10.1038/sj.embor.embor914.
- ↑ Toney, M. D. "Reaction specificity in pyridoxal enzymes." Archives of biochemistry and biophysics (2005) 433: 279-287.
- ↑ 4,0 4,1 Samuel, G. and Reeves, P. "Biosynthesis of O-antigens: genes and pathways involved in nucleotide sugar precusor synthesis and O-antigen assembly." Carbohydrate research (2003) 338:2503-2519.
- ↑ Cook P. D., Thoden J.B. and Holden H. M. "The structure of GDP-4-keto-6-deoxymannose-3-dehydratase: a unique coenzyme B6-dependent enzyme." Protein Science (2006) 15:2093-2106.
- ↑ N. M. F. S. A. Cerqueira, P. A. Fernandes, M. J. Ramos (2011). "Computational Mechanistic Studies Addressed to the Transimination Reaction Present in All Pyridoxal 5′-Phosphate-Requiring Enzymes". Journal of Chemical Theory and Computation 7 (5): 1356–1368. doi:10.1021/ct1002219.
- ↑ 7,0 7,1 7,2 7,3 Eliot, A. C.; Kirsch, J. F. (2004). "Pyridoxalphosphate enzymes: Mechanistic, Structural, and Evolutionary Considerations". Annual Review of Biochemistry 73: 383–415. PMID 15189147. doi:10.1146/annurev.biochem.73.011303.074021.
- ↑ 8,0 8,1 Fitzpatrick, T. B.; Amrhein, N.; Kappes, B.; Macheroux, P.; Tews, I.; Raschle, T. (2007). "Two independent routes of de novo vitamin B6 biosynthesis: Not that different after all". Biochemical Journal 407 (1): 1–13. PMID 17822383. doi:10.1042/BJ20070765.
- ↑ Sakai, A.; Kita, M.; Tani, Y. (2004). "Recent progress of vitamin B6 biosynthesis". Journal of nutritional science and vitaminology 50 (2): 69–77. PMID 15242009. doi:10.3177/jnsv.50.69.
- ↑ Lam, H. M.; Winkler, M. E. (1990). "Metabolic relationships between pyridoxine (vitamin B6) and serine biosynthesis in Escherichia coli K-12". Journal of bacteriology 172 (11): 6518–6528. PMC 526841. PMID 2121717.
- ↑ Zhu, J.; Burgner, J. W.; Harms, E.; Belitsky, B. R.; Smith, J. L. (2005). "A New Arrangement of (α/β)8 Barrels in the Synthase Subunit of PLP Synthase". Journal of Biological Chemistry 280 (30): 27914–27923. PMID 15911615. doi:10.1074/jbc.M503642200.
- ↑ 12,0 12,1 Austin, S. M.; Waddell, T. G. (1999). "Prebiotic synthesis of vitamin B6-type compounds". Origins of life and evolution of the biosphere : the journal of the International Society for the Study of the Origin of Life 29 (3): 287–296. PMID 10389266.
- ↑ Aylward, N.; Bofinger, N. (2006). "A plausible prebiotic synthesis of pyridoxal phosphate: Vitamin B6 – A computational study". Biophysical Chemistry 123 (2–3): 113–121. PMID 16730878. doi:10.1016/j.bpc.2006.04.014.
- ↑ Anthony, K. G.; Strych, U.; Yeung, K. R.; Shoen, C. S.; Perez, O.; Krause, K. L.; Cynamon, M. H.; Aristoff, P. A.; Koski, R. A. (2011). Ahmed, Niyaz, ed. "New Classes of Alanine Racemase Inhibitors Identified by High-Throughput Screening Show Antimicrobial Activity against Mycobacterium tuberculosis". PLoS ONE 6 (5): e20374. PMC 3102704. PMID 21637807. doi:10.1371/journal.pone.0020374.
