ARN nucleolar pequeno
O ARN nucleolar pequeno[1] (ARNnop[1] ou snoRNA nas súas siglas en inglés) é un tipo de pequenas moléculas de ARN que teñen como principal función dirixir a modificación química doutros ARN, fundamentalmente os ARN ribosómicos, ARN de transferencia e ARN nucleares pequenos. Hai dúas clases principais de snoRNA, os snoRNAs da caixa C/D, que están asociados á metilación, e os snoRNAs da caixa H/ACA, que están asociados á pseudouridilación. Os snoRNA denomínanse normalmente "ARN guías" pero non deben confundirse cos ARN guías que dirixen a modificación (editing) do ARN nos tripanosomas. Os snoRNA están asociados formando complexos con proteínas, que se denominan ribonucleoproteínas nucleolares pequenas ou snoRNP.
Modificacións dirixidas polos snoRNA
[editar | editar a fonte]Despois da transcrición, as moléculas de ARNr que se están a formar (denominadas pre-ARNr) deben de sufrir un procesamento para converterse en ARNr maduro. Antes da súa escisión pola acción de exo- e endonucleases o pre-ARNr sofre unha complexa serie de modificacións nos seus nucleósidos, que inclúen metilacións e pseudouridilacións, as cales son dirixidas polos snoRNAs.
- Metilación é a unión ou substitución de grupos metilo en diversos substratos. O ARNr humano presenta aproximadamente 115 modificacións de grupos metilo. A maioría destas son metilacións 2'O-ribosa (nas que o grupo metilo se une á ribosa).[2]
- Pseudouridilación é a conversión (isomerización) do nucleósido uridina a unha forma isomérica distinta, a pseudouridina (Ψ). Os ARNr maduros humanos presentan aproximadamente 95 modificacións Ψ.[2]
Cada molécula de snoRNA actúa como guía para facer só unha (ou dúas) modificacións individuais nun ARN diana. Para levar a cabo estas modificacións, cada snoRNA asóciase con polos menos catro moléculas proteicas formando un complexo ARN/proteína denominado ribonucleoproteína nucleolar pequena (snoRNP). As proteínas que se asocian con cada ARN dependen do tipo de molécula de snoRNA de que se trate (ver as familias de snoRNA guía máis abaixo). A molécula de snoRNA contén un elemento antisentido (un tramo de 10-20 nucleótidos) que ten complementariedade de bases coa secuencia que rodea a base (nucleótido) obxectivo da modificación na molécula do pre-ARN. Isto permite que a snoRNP recoñeza e se una ao seu ARN obxectivo. Unha vez que a snoRNP se uniu ao seu sitio obxectivo, as proteínas asociadas están no lugar fisicamente correcto para catalizar a modificación química da base obxectivo.
Familias de snoRNA guía
[editar | editar a fonte]Os dous tipos de modificacións do ARNr (metilación e pseudouridilación) son dirixidas por dous tipos diferentes de familias de snoRNA. Estas familias de snoRNA denomínanse snoRNA de caixa C/D antisentido e snoRNA de caixa H/ACA, distinción que está baseada na presenza de motivos de secuencia conservados nos snoRNA. Aínda que hai excepcións, como regra xeral os membros da caixa C/D (C/D box) dirixen a metilación e os membros H/ACA dirixen a pseudouridilación. Os membros de cada familia poden variar en bioxénese, estrutura e función, pero cada familia clasifícase polas seguintes características xerais. Para máis detalles ver a revisión.[3] Os snoRNA clasifícanse dentro dos ARN nucleares pequenos no MeSH.
snoRNA de caixa C/D
[editar | editar a fonte]
Os snoRNA de caixa C/D conteñen dous curtos motivos de secuencia conservados, C (UGAUGA) e D (CUGA) localizados preto dos extremos 5' e 3' do snoRNA respectivamente. Rexións curtas (~ 5 nucleótidos) situadas corrente arriba da caixa C e corrente abaixo da caixa D son normalmente complementarias en bases e forman unha estrutura caixa-talo que pon os motivos das caixas C e D en estreita proximidade. Esta estrutura caixa-talo é esencial para a correcta síntese dos snoRNA e para a súa localización nucleolar.[4] Moitos snoRNA de caixa C/D conteñen tamén unha copia adicional menos conservada dos motivos C e D (denominada C' e D') situada na porción central da molécula de snoRNA. Unha rexión conservada de 10-21 nucleótidos situada corrente arriba da caixa D é complementaria do sitio de metilación do ARN diana e permite que o snoRNA forme un dúplex de ARN co ARN.[5] O nucleótido que debe ser modificado no ARN diana está localizado normalmente na quinta posición corrente arriba a partir da caixa D (ou da caixa D').[6][7] Os snoRNA de caixa C/D asócianse con catro proteínas esenciais conservadas na evolución, que son en eucariotas: fibrilarina (Nop1p), Nop56p, Nop58p, e Snu13 (o seu homólogo en arqueas é L7Ae)) as cales forman o núcleo das snoRNP de caixa C/D.[3]
Existe tamén un snoRNA de caixa C/D eucariótico (snoRNA U3) que non se viu que dirixa metilacións 2'-O. En vez diso, funciona dirixindo a escisión do pre-ARNr.
snoRNA de caixa H/ACA
[editar | editar a fonte]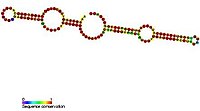
Os snoRNA de caixa H/ACA teñen unha estrutura secundaria común que consiste en dúas forquitas e dúas rexións dunha soa cadea chamadas estruturas forquita-bisagra-forquita-cola.[3] Os snoRNA de caixa H/ACA tamén conteñen motivos de secuencia conservados coñecidos como caixa H (consenso ANANNA) e a caixa ACA (ACA). Ambos os motivos están normalmente situados nas rexións dunha soa cadea da estrutura secundaria. O motivo H está localizado na bisagra e o motivo ACA na rexión da cola, a 3 nucleótidos do extremo 3' da secuencia.[8] As rexións forquita conteñen protuberancias internas coñecidas como bucles de recoñecemento, nos cales están localizadas as secuencias guía antisentido (complementarias en bases coa secuencia obxectivo). Esta secuencia de recoñecemento é bipartita (construída a partir de cada un dos dous brazos da rexión bucle) e forma pseudonós complexos co ARN obxectivo. O snoRNA de caixa H/ACA asócianse con catro proteínas esenciais conservadas evolutivamente, que son: disquerina (Cbf5p), Gar1p, Nhp2p e Nop10p, que forman o núcleo das snoRNP de caixa H/ACA.[3] Porén, en eucariotas inferiores como os tripanosomas, existen ARN similares na forma de estrutura en forquita simple e unha caixa AGA en lugar da caixa ACA no extremo 3' do ARN.[9]
O compoñente ARN da telomerase humana (hTERC) contén un dominio H/ACA para a formación de pre-RNP e a localización nucleolar da propia RNP telomerase.[10] Un dato importante é que a snoRNP H/ACA foi implicada na rara enfermidade xenética disqueratose conxénita debido á súa asociación coa telomerase humana. As mutacións no compoñente proteico da snoRNP H/ACA orixinan unha redución dos niveis fisiolóxicos de telomerase (TERC). Isto foi claramente correlaciondo coa patoloxía asociada á disqueratose conxénita, a cal parece ser unha doenza producida principalmente por un deficiente mantemento do telómero.
snoRNA compostos de caixa H/ACA e C/D
[editar | editar a fonte]Identificouse un snoRNA U85 guía pouco común que funciona tanto na metilación 2'-O-ribosa coma na pseudouridilación do ARN nuclear pequeno (snRNA) U5.[11] Este snoRNA composto contén tanto dominios de caixa C/D coma de H/ACA e asóciase coas proteínas específicas de ambas as clases de snoRNA (fibrilarina e Gar1p respectivamente). Foron caracterizados máis snoRNA compostos.[12]
Estes snoRNA compostos viuse que se acumulan nun orgánulo subcelular chamado corpo de Cajal e denomínanse ARN específicos dos corpos de Cajal. Isto contraponse ao que se observa na maioría dos snoRNA de caixa C/D ou de caixa H/ACA, os cales se localizan no nucléolo. Estes ARN específicos dos corpos de Cajal suponse que poden estar implicados na modificación dos ARN espliceosomais U1, U2, U4, U5 e U12 transcritos pola ARN polimerase II.[12] Non todos os snoRNA que foron localizados nos corpos de Cajal son snoRNA compostos de caixa C/D e H/ACA.
snoRNA "orfos"
[editar | editar a fonte]As dianas de novos snoRNA identificados recentemente poden predicirse baseándose na complementariedade da secuencia entre os supostos ARN diana e os elementos antisentido ou bucles de recoñecemento na secuencia dos snoRNA. Porén, hai un crecente número de snoRNA guías "orfos" para os que non se coñece ningún ARN diana, o cal suxire que podería haber máis proteínas ou transcritos implicados no procesamento do ARNr cós que previamente se pensaba e/ou que algúns snoRNA teñen outras funcións que non teñen que ver co ARNr.[13][14] Hai probas de que algúns destes snoRNA orfos regulan transcritos de splicing alternativo.[15] Por exemplo, parece que o snoRNA de caixa C/D SNORD115, regula o splicing alternativo do ARNm do receptor 2C da serotonina por medio dunha rexión conservada complementaria.[16][17] Para outro snoRNA de caixa C/D, o SNORD116, que se encontra no mesmo clúster ca SNORD115, predicíronse 23 posibles dianas nos xenes codificadores de proteínas usando un método bioinformático. Destes, unha gran proporción viuse que sufrían splicing alternativo, o que suxire un papel do SNORD116 na regulación do splicing alternativo.[18]
Modificacións da diana
[editar | editar a fonte]O efecto preciso das modificacións de metilación e pseudouridilación na función dos ARN maduros non se coñece aínda. As modificacións non parecen ser esenciais pero sábese que incrementan sutilmente o pregamento do ARN e a interacción con proteínas ribosómicas. Apoia a súa importancia o feito de que as modificacións dos sitios diana están exclusivamente localizados nos dominios conservados e funcionalmente importantes do ARN maduro e están normalmente conservados en especies eucariotas moi afastadas na árbore evolutiva.[3]
- A ribosa con metilación 2'-O causa un incremento na conformación 3'-endo.
- A pseudouridina (psi/Ψ) engade outra opción para a unión a H.
- O ARN moi metilado está protexido da hidrólise. O ARNr actúa como un ribozima catalizando a súa propia hidrólise e splicing.
Organización xenómica
[editar | editar a fonte]A maioría dos xenes de snoRNA de vertebrados están codificados nos intróns de proteínas implicadas na síntese do ribosoma ou tradución, e son sintetizados pola ARN polimerase II, pero os snoRNA tamén poden ser transcritos a partir dos seus propios promotores polas ARN polimerases II ou III.
Loci impresos
[editar | editar a fonte]No xenoma humano hai polo menos dous exemplos de snoRNA de caixa C/D que foron atopados en repeticións en tándem dentro de loci impresos (imprinted loci). Estes dous loci (14q32 no cromosoma 14 e 15q11q13 no cromosoma 15) foron extensivamente caracterizados e en ambas as rexións foron atopados múltiples snoRNA situados dentro de intróns en agrupacións de copias estreitamente relacionadas.
En 15q11q13, foron identificados cinco snoRNA: SNORD64, SNORD107, SNORD108, SNORD109 (dúas copias), SNORD116 (29 copias) e SNORD115 (48 copias). A perda das 29 copias de SNORD116 desta rexión é a causa da síndrome de Prader-Willi[19][20][21][22] e a ganancia de copias adicionais de SNORD115 foi asociada co autismo.[23][24] [25]
A rexión 14q32 contén repeticións de dous snoRNAs: SNORD113 (9 copias) e SNORD114 (31 copias) dentro de intróns dun transcrito de ARN non codificante específico de tecido (MEG8). O dominio 14q32 comparte características xenéticas co loci impreso 15q11-q13 e foi suxerida unha posible función para as repeticións en tándem de snoRNA de caixa C/D na evolución ou no mecanismo de loci impresos.[26]
Outras funcións dos snoRNA
[editar | editar a fonte]Os snoRNA poden funcionar como microARN. Demostrouse que o ARN ACA45 humano é un snoRNA que pode ser procesdo nun microARN maduro de 21 nucleótidos de longo por unha endonuclease Dicer da familia da RNase III.[27] Este produto snoRNA fora previamente identificado como mmu-miR-1839 Arquivado 28 de xullo de 2011 en Wayback Machine. e demostrouse que era procesado independente da endorribonuclease Drosha que xera os outros microARN.[28] Análises bioinformáticos revelaron que fragmentos similares a microARN supostamente derivados de snoRNA están presentes en diversos organismos.[29]
Recentemente, descubriuse que os snoRNA poden ter funcións non relacionadas co ARNr. Unha desas funcións é a regulación do splicing alternativo do transcrito transxene, realizado polo snoRNA HBII-52, tamén coñecido por SNORD115.[30]
Outro recente descubrimento tamén mostra a existencia de características de snoRNA, microARN e piRNA nun novo ARN non codificante, o ARN non codificante x (x-ncRNA) e a súa implicación biolóxica no Homo sapiens.[31]
Notas
[editar | editar a fonte]- ↑ 1,0 1,1 Coordinadores: Jaime Gómez Márquez, Ana Mª Viñas Díaz e Manuel González González. Redactores: David Villar Docampo e Luís Vale Ferreira. Revisores lingüísticos: Víctor Fresco e Mª Liliana Martínez Calvo. (2010). Dicionario de bioloxía galego-castelán-inglés. (PDF). Xunta de Galicia. p. 25. ISBN 978-84-453-4973-1.
- ↑ 2,0 2,1 Maden BE, Hughes JM (1997). "Eukaryotic ribosomal RNA: the recent excitement in the nucleotide modification problem". Chromosoma 105 (7-8): 391–400. PMID 9211966. doi:10.1007/BF02510475.
- ↑ 3,0 3,1 3,2 3,3 3,4 Bachellerie, JP; Cavaille J, Huttenhofer A (2002). "The expanding snoRNA world". Biochimie 84 (8): 775–790. PMID 12457565. doi:10.1016/S0300-9084(02)01402-5.
- ↑ Samarsky, DA; Fournier MJ, Singer RH, Bertrand E (1998). "The snoRNA box C/D motif directs nucleolar targeting and also couples snoRNA synthesis and localization". EMBO 17 (13): 3747–57. PMC 1170710. PMID 9649444. doi:10.1093/emboj/17.13.3747.
- ↑ Kiss-László Z, Henry Y, Kiss T (1998). "Sequence and structural elements of methylation guide snoRNAs essential for site-specific ribose methylation of pre-rRNA". EMBO J. 17 (3): 797–807. PMC 1170428. PMID 9451004. doi:10.1093/emboj/17.3.797.
- ↑ Cavaillé J, Nicoloso M, Bachellerie JP (1996). "Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides". Nature 383 (6602): 732–5. PMID 8878486. doi:10.1038/383732a0.
- ↑ Kiss-László Z, Henry Y, Bachellerie JP, Caizergues-Ferrer M, Kiss T (1996). "Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs". Cell 85 (7): 1077–88. PMID 8674114. doi:10.1016/S0092-8674(00)81308-2.
- ↑ Ganot P, Caizergues-Ferrer M, Kiss T (1997). "The family of box ACA small nucleolar RNAs is defined by an evolutionarily conserved secondary structure and ubiquitous sequence elements essential for RNA accumulation". Genes Dev. 11 (7): 941–56. PMID 9106664. doi:10.1101/gad.11.7.941.
- ↑ Liang XH, Liu L, Michaeli S (2001). "Identification of the first trypanosome H/ACA RNA that guides pseudouridine formation on rRNA.". JBC. 276 (43): 40313–8. PMID 11483606. doi:10.1074/jbc.M104488200.
- ↑ Trahan C, Dragon F (2009). "Dyskeratosis congenita mutations in the H/ACA domain of human telomerase RNA affect its assembly into a pre-RNP.". RNA 15 (2): 235–43. PMC 2648702. PMID 19095616. doi:10.1261/rna.1354009.
- ↑ Jády BE, Kiss T (2001). "A small nucleolar guide RNA functions both in 2'-O-ribose methylation and pseudouridylation of the U5 spliceosomal RNA". EMBO J. 20 (3): 541–51. PMC 133463. PMID 11157760. doi:10.1093/emboj/20.3.541.
- ↑ 12,0 12,1 Darzacq X, Jády BE, Verheggen C, Kiss AM, Bertrand E, Kiss T (2002). "Cajal body-specific small nuclear RNAs: a novel class of 2'-O-methylation and pseudouridylation guide RNAs". EMBO J. 21 (11): 2746–56. PMC 126017. PMID 12032087. doi:10.1093/emboj/21.11.2746.
- ↑ Jády, B. K. (Mar 2000). "Characterisation of the U83 and U84 small nucleolar RNAs: two novel 2′-O-ribose methylation guide RNAs that lack complementarities to ribosomal RNAs" (Free full text). Nucleic Acids Research 28 (6): 1348–1354. doi:10.1093/nar/28.6.1348. ISSN 0305-1048. PMC 111033. PMID 10684929. http://nar.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=10684929.
- ↑ Li, G. .; Zhou, H. .; Luo, P. .; Zhang, P. .; Qu, H. . (Apr 2005). "Identification and functional analysis of 20 Box H/ACA small nucleolar RNAs (snoRNAs) from Schizosaccharomyces pombe" (Free full text). The Journal of Biological Chemistry 280 (16): 16446–16455. doi:10.1074/jbc.M500326200. ISSN 0021-9258. PMID 15716270. http://www.jbc.org/cgi/pmidlookup?view=long&pmid=15716270 Arquivado 25 de setembro de 2019 en Wayback Machine..
- ↑ Kishore, S. .; Stamm, S. . (2006). "Regulation of alternative splicing by snoRNAs". Cold Spring Harbor symposia on quantitative biology 71: 329–334. doi:10.1101/sqb.2006.71.024. ISSN 0091-7451. PMID 17381313.
- ↑ Kishore S, Stamm S (2006). "The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C". Science 311 (5758): 230–2. Bibcode:2006Sci...311..230K. PMID 16357227. doi:10.1126/science.1118265.
- ↑ Doe, M. .; Relkovic, D. .; Garfield, S. .; Dalley, W. .; Theobald, E. .; Humby, T. .; Wilkinson, S. .; Isles, R. . (Jun 2009). "Loss of the imprinted snoRNA mbii-52 leads to increased 5htr2c pre-RNA editing and altered 5HT2CR-mediated behaviour". Human Molecular Genetics 18 (12): 2140–2148. doi:10.1093/hmg/ddp137. ISSN 0964-6906. PMC 2685753. PMID 19304781. //www.ncbi.nlm.nih.gov/pmc/articles/PMC2685753/.
- ↑ Bazeley PS, Shepelev V, Talebizadeh Z, Butler MG, Fedorova L, Filatov V, Fedorov A (2008). "snoTARGET shows that human orphan snoRNA targets locate close to alternative splice junctions.". Gene 408 (1-2): 172–9. PMID 18160232. doi:10.1016/j.gene.2007.10.037.
- ↑ Skryabin BV, Gubar LV, Seeger B; et al. (2007). "Deletion of the MBII-85 snoRNA gene cluster in mice results in postnatal growth retardation". PLoS Genet. 3 (12): e235. PMC 2323313. PMID 18166085. doi:10.1371/journal.pgen.0030235. Arquivado dende o orixinal o 05 de marzo de 2014. Consultado o 29 de agosto de 2011.
- ↑ Sahoo T, del Gaudio D, German JR, Shinawi M, Peters SU, Person RE, Garnica A, Cheung SW, Beaudet AL (2008). "Prader-Willi phenotype caused by paternal deficiency for the HBII-85 C/D box small nucleolar RNA cluster.". Nat Genet 40 (6): 719–21. PMC 2705197. PMID 18500341. doi:10.1038/ng.158.
- ↑ Ding F, Li HH, Zhang S, Solomon NM, Camper SA, Cohen P, Francke U (2008). "SnoRNA Snord116 (Pwcr1/MBII-85) deletion causes growth deficiency and hyperphagia in mice.". PLoS ONE 3 (3): e1709. PMC 2248623. PMID 18320030. doi:10.1371/journal.pone.0001709.
- ↑ Ding F, Prints Y, Dhar MS, Johnson DK, Garnacho-Montero C, Nicholls RD, Francke U (2005). "Lack of Pwcr1/MBII-85 snoRNA is critical for neonatal lethality in Prader-Willi syndrome mouse models.". Mamm Genome 16 (6): 424–31. PMID 16075369. doi:10.1007/s00335-005-2460-2.
- ↑ Nakatani J, Tamada K, Hatanaka F; et al. (2009). "Abnormal behavior in a chromosome-engineered mouse model for human 15q11-13 duplication seen in autism". Cell 137 (7): 1235–46. PMID 19563756. doi:10.1016/j.cell.2009.04.024.
- ↑ Bolton PF, Veltman MW, Weisblatt E; et al. (2004). "Chromosome 15q11-13 abnormalities and other medical conditions in individuals with autism spectrum disorders". Psychiatr. Genet. 14 (3): 131–7. PMID 15318025. doi:10.1097/00041444-200409000-00002.
- ↑ Cook EH, Scherer SW (2008). "Copy-number variations associated with neuropsychiatric conditions". Nature 455 (7215): 919–23. PMID 18923514. doi:10.1038/nature07458.
- ↑ Cavaillé J, Seitz H, Paulsen M, Ferguson-Smith AC, Bachellerie JP (2002). "Identification of tandemly-repeated C/D snoRNA genes at the imprinted human 14q32 domain reminiscent of those at the Prader-Willi/Angelman syndrome region". Hum. Mol. Genet. 11 (13): 1527–38. PMID 12045206. doi:10.1093/hmg/11.13.1527.
- ↑ Ender, C.; Krek, A.; Friedländer, M.; Beitzinger, M.; Weinmann, L.; Chen, W.; Pfeffer, S.; Rajewsky, N. et al. (2008). "A human snoRNA with microRNA-like functions". Molecular Cell 32 (4): 519–528. doi:10.1016/j.molcel.2008.10.017. PMID 19026782.
- ↑ Babiarz, J.; Ruby, J.; Wang, Y.; Bartel, D.; Blelloch, R. (2008). "Mouse ES cells express endogenous shRNAs, siRNAs, and other Microprocessor-independent, Dicer-dependent small RNAs". Genes & Development 22 (20): 2773–2785. doi:10.1101/gad.1705308. PMC 2569885. PMID 18923076. //www.ncbi.nlm.nih.gov/pmc/articles/PMC2569885/.
- ↑ Taft, R.; Glazov, E.; Lassmann, T.; Hayashizaki, Y.; Carninci, P.; Mattick, J. (2009). "Small RNAs derived from snoRNAs". RNA 15 (7): 1233–1240. doi:10.1261/rna.1528909. PMC 2704076. PMID 19474147. //www.ncbi.nlm.nih.gov/pmc/articles/PMC2704076/.
- ↑ Kishore S, Stamm S (2006). "The snoRNA HBII-52 regulates alternative splicing of the serotonin receptor 2C". Science 311 (5758): 230–1. Bibcode:2006Sci...311..230K. PMID 16357227. doi:10.1126/science.1118265.
- ↑ Kandhavelu M, Lammi C, Buccioni M, Dal Ben D, Volpini R, Marucci G (2009). "Existence of snoRNA, microRNA, piRNA characteristics in a novel non-coding RNA: x-ncRNA and its biological implication in Homo sapiens". Journal of Bioinformatics and Sequence Analysis 1 (2): 031–040.
Véxase tamén
[editar | editar a fonte]Ligazóns externas
[editar | editar a fonte]- plant snoRNA databaseArquivado 07 de abril de 2014 en Wayback Machine.
- snoRNAbase: human H/ACA and C/D box snoRNA database
- snoRNP Database
- The yeast snoRNA database
- Rfam page for C/D box snoRNAs[Ligazón morta]
- Rfam page for H/ACA box snoRNAs[Ligazón morta]
- Rfam page for scaRNA snoRNAs[Ligazón morta]
