Dicer

| |
| Estrutura dos dominios de unión ao ARN bicatenario e de RNaseIIIb do Dicer de rato | |
Dicer 1, ribonuclease tipo III
| |
| Identificadores | |
| Símbolo | DICER1 |
| Símbolos alt. | DCR1; Dicer; Dicer1e; HERNA; K12H4.8-LIKE; MNG1; RMSE2 |
| Entrez | 23405 |
| OMIM | |
| RefSeq | NP_001182502 |
| UniProt | Q9UPY3 |
| Outros datos | |
| Número EC | 3.1.26.3 |
| Locus | Cr. 14 :(95.09 – 95.16 Mb) |
Dicer (/díθer/), tamén chamada endorribonuclease Dicer ou helicase con motivo de RNase, é unha proteína encimática que nos humanos está codificada no xene DICER1, situado no cromosoma 14. Dicer forma parte da familia da RNase III e cliva (corta) ARNs bicatenarios e pre-microARN (pre-miARN) en curtos fragmentos de ARN bicatenarios chamados ARNs interferentes pequenos (cando proceden de ARNs bicatenarios) e microARNs (cando proceden de pre-miARNs). Estes fragmentos son de aproximadamente 20-25 pares de bases de lonxitude e teñen un extremo 3' que sobresae de 2 bases. Dicer facilita a activación do complexo silenciador inducido por ARN (RISC), que é esencial para que se poida realizar a interferencia de ARN. O RISC ten un compoñente catalítico chamado Argonauta, que é unha endonuclease con capacidade de degradar ARNs mensaxeiros (ARNm).
Descubrimento
[editar | editar a fonte]A Dicer deulle o seu nome en 2001 Emily Bernstein, unha estudante graduada no laboratorio de Greg Hannon no Cold Spring Harbor Laboratory, que trataba de descubrir cal era o encima responsable de xerar os pequenos fragmentos de ARN a partir dos ARN bicatenarios. A capacidade de Dicer de xerar fragmentos de ARN de ~22 nucleótidos descubriuse ao separalo do complexo encimático RISC despois de iniciar a vía de interferencia de ARN con transfección de ARN bicatenario. Este experimento mostrou que o RISC non era o responsable de xerar os fragmentos nucleotídicos pequenos observados. Posteriores experimentos comprobaron as capacidades dos encimas da familia da RNase III para crear fragmentos de ARN, o que reduciu a busca ao encima CG4792 de Drosophila, agora chamado Dicer.[1]
Existen ortólogos de Dicer en moitos outros organismos.[2] No musgo Physcomitrella patens DCL1b, unha das catro proteínas DICER non está implicada na bioxénese de miARN senón que o que fai é cortar en cachos os transcritos diana de miARN. Deste xeito, descubriuse un novo mecanismo de regulación da expresión xénica, o silenciamento epixenético de xenes por miARNs.[3]
En canto á súa estrutura cristalina, o primeiro Dicer que foi examinado foi o do protozoo Giardia intestinalis. Descubriuse por cristalografía de raios X un dominio PAZ e dous dominios RNase III. O tamaño da proteína neste protozoo é 82 kDa, pero son maiores noutros organismos; por exemplo, a humana é de 219 kDa. A diferenza en tamaño entre o Dicer humano e o de G. intestinalis débese a que no humano hai polo menos cinco dominios adicionais. Estes dominios son importantes na regulación da actividade de Dicer, no procesamento de ARNs bicatenarios, e no funcionamento de factores proteicos que inician a interferencia de ARN.[4]
Dominios funcionais
[editar | editar a fonte]
A clasificación do Dicer humano (tamén denominado hsDicer ou DICER1) como unha ribonuclease III débese a que contén tanto dominios helicase coma PAZ (Piwi/Argonauta/Zwille).[6][7] Ademais destes dominios, o hsDicer contén outros catro dominios funcionais: dous dominios de RNaseIII e dous dominios de unión a ARN bicatenario (DUF283 e dsRBD).[4][8]
Investigacións recentes indican que o dominio PAZ ten a capacidade de unirse ao extremo 3' que sobresae de 2 nucleótidos do ARN bicatenario, mentres que os dominios catalíticos de RNaseIII forman un pseudo-dímero arredor do ARN bicatenario para iniciar a clivaxe das febras. Isto ten como resultado un acurtamento funcional da febra de ARN bicatenario. A distancia entre os dominios PAZ e RNaseIII está determinada polo ángulo da hélice conectora e inflúe na lonxitude do produto microARN.[5] O dominio dsRBD únese ao ARN bicatenario, aínda que o sitio de unión específico do dominio non foi definido. É posible que este dominio funcione como parte dun complexo formado con outras proteínas reguladoras (TRBP en humanos, R2D2, Loqs en Drosophila) para colocar de forma efectiva os dominios de RNaseIII e así controlar a especificidade dos produtos de pequenos ARNs.[9] O dominio helicase foi implicado no procesamento de substratos longos.[9]
Papel na interferencia de ARN
[editar | editar a fonte]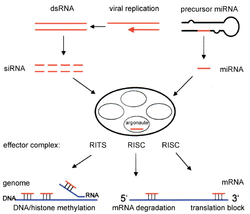
MicroARN
[editar | editar a fonte]A interferencia de ARN é un proceso no que mediante a degradación de moléculas de ARN orixinando miARN se inhibe a expresión xénica de secuencias de ARNm do hóspede específicas. O miARN prodúcese na célula empezando polo transcrito primario miARN (pri-miARN) no núcleo celular. Estas longas secuencias son clivadas orixinando un miARN precursor máis pequeno (pre-miARN), que ten xeralmente uns 70 nucleótidos e unha estrutura en forquita. Os pri-miARNs son identificados polo encima DGCR8 (Pasha en Drosophila) e clivados polo encima Drosha para formar o pre-miARN. Estes pre-miARN son despois clivados por Dicer para formar o miARN maduro.[11]
ARN interferente pequeno
[editar | editar a fonte]Os ARNs interferentes pequenos (siRNA) prodúcense e funcionan de xeito similar ao miARN ao clivar ARN bicatenario con Dicer en fragmentos máis pequenos de 21 a 23 nucleótidos de lonxitude.[9] Tanto os microARNs coma os ARNs interferentes pequenos activan o complexo silenciador inducido por ARN (RISC), que encontra a diana de secuencia complementaria de ARNm e cliva o ARN usando a súa RNase.[12] Esta á súa vez silencia un determinado xene por medio da interferencia de ARN.[13] Os ARNs interferentes pequenos e os microARNs difiren en que os siRNAs son totalmente específicos para unha secuencia de ARNm, mentres que os miARNs non son completamente complementarios desa secuencia de ARNm. Os microARNs poden interaccionar con dianas que teñen secuencias similares, o cal inhibe a tradución de diferentes xenes.[14] En xeral, a interferencia de ARN é unha parte esencial dos procesos normais de organismos como os humanos, e é unha área que está a ser investigada como unha ferramenta de diagnóstico e terapéutica para dianas no cancro.[11]

Doenzas
[editar | editar a fonte]Dexeneración macular
[editar | editar a fonte]A dexeneración macular relacionada coa idade é unha importante causa de cegueira nos países desenvolvidos. O papel de Dicer nesta doenza fíxose evidente cando se descubriu que os pacientes aectados mostraban uns niveis diminuídos de Dicer nas súas células do epitelio pigmentado da retina. Os ratos con knock-out para Dicer, que carecían de Dicer só no seu epitelio pigmentado da retina, mostraban síntomas similares. Porén, outros ratos que carecían de importantes proteínas da vía de interferencia de ARN como Drosha e Pasha, non presentaban síntomas de dexeneración macular, a diferenza dos ratos con knock-out para Dicer. Esta observación suxire que Dicer ten un papel específico na saúde da retina que é independente da vía de interferencia de ARN e non é unha función debida á xeración de si/miRNA. Atopouse tamén que unha forma de ARN chamada ARN Alu (que son os transcritos de ARN dos elementos Alu xenéticos) estaba elevada en pacientes con niveis insuficientes de Dicer. Estas febras non codificantes de ARN poden dobrarse en bucle formando estruturas de ARN bicatenario que serían degradadas por Dicer nunha retina con boa saúde. Porén, con niveis insuficientes de Dicer, a acumulación de ARN Alu leva á dexeneración do pigmento pigmentario da retina como resultado de inflamación.[15][16]
Cancro
[editar | editar a fonte]Os perfís de expresión alterada de miARN en cancros malignos indica un papel importante dos miARNs e de Dicer no desenvolvemento e prognose do cancro. Os miARNs poden fncionar como supresores de tumores e, por tanto, a súa expresión alterada pode ter como resultado a xeración dun tumor.[17] Na análise de cancros de pulmón e de ovario, hai unha correlación entre o mal prognóstico e a diminución do tempo de supervivencia do paciente e a diminución da expresión de Dicer e Drosha. A diminución dos ARNm de Dicer correlaciónase cun estado avanzado do tumor. Non obstante, tamén se atopou que unha alta expresión de Dicer noutros cancros, como o de próstata e o de esófago, se correlaciona cun peor prognóstico para o paciente. Esta discrepancia entre tipos de cancro suxire que hai procesos regulatorios de interferencia de ARN únicos que implican a Dicer en diferentes tipos de tumores.[11]
Dicer está tamén implicado na reparación do ADN. Os danos no ADN increméntanse en células de mamíferos que teñen unha expresión diminuída de Dicer como resultado da diminución da eficacia na reparación dos danos no ADN e outros mecanismos. Por exemplo, o siRNA procedente de roturas de dobre cadea no ADN (producido por Dicer) pode actuar como guía para complexos proteicos que interveñen nos mecanismos de reparación das roturas de dobre cadea e pode tamén dirixir modificacións na cromatina. Adicionalmente, os patróns de expresión dos miARNs cambian como resultado dos danos no ADN causados por radiacións ionizantes ou ultravioletas. Os mecanismos de interferencia de ARN son responsables do silenciamento de transposóns e, na súa ausencia, cando Dicer é sometido a knock-out ou knock-down, isto pode xerar transposóns activados que causen danos no ADN. A acumulación de danos no ADN pode orixinar células con mutacións oncoxénicas que poden desenvolver un tumor.[11]
Patoxénese viral
[editar | editar a fonte]A infección por virus de ARN pode desencadear a fervenza de reaccións da interferencia de ARN. Probablemente Dicer está envolto na inmunidade viral, xa que os virus que infectan tanto a células animais coma de plantas conteñen proteínas deseñadas para inhibir a resposta de interferencia de ARN. Nos humanos, os virus VIH-1, influenza e vaccinia codifican ditas proteínas supresoras da interferencia de ARN. A inhibición de Dicer é beneficiosa para o virus, xa que Dicer pode clivar o ARN bicatenario viral e cargar o produto no complexo RISC, o que ten como resultado a degradación de ARNs diana, que son os ARNm virais, o que serve para loitar contra a infección. Outro mecanismo potencial da patoxénese viral é o bloqueo de Dicer como un modo de inhibir as vías de miARN celulares.[18]
En insectos
[editar | editar a fonte]Os insectos poden utilizar Dicer como un potente antiviral. Isto é especialmente importante dado que os mosquitos son os vectores responsables da transmisión de moitas doenzas virais como as producidas polos potencialmente mortais arbovirus: virus do Nilo Occidental, virus do dengue e virus da febre amarela.[19] A vía de interferencia de ARN nos insectos é moi similar á doutros animais: Dicer-2 cliva o ARN viral e cárgao no complexo RISC, no cal unha febra do ARN serve como molde para a produción de produtos da interferencia de ARN e a outra é degradada. Os insectos con mutacións que orixinan compoñentes non funcionais das súas vías de interferencia de ARN mostran un incremento na súa carga viral para os virus dos que son portadores ou un incremento da susceptibilidade para os virus dos cales son hóspedes. Igual que nos virus humanos, nos virus de insectos evolucionaron mecanismos para evitar as vías de interferencia de ARN. Por exemplo, o virus C de Drosophila codifica a proteína 1A, que se une a ARNs bicatenarios e dese modo protéxese da clivaxe polo encima Dicer do insecto e da súa carga no complexo RISC. O ascovirus 3a de Heliothis virescens (un lepidóptero) codifica un encima RNase III similar aos dominios de RNase III de Dicer, que pode competir polo substrato de ARN bicatenario e degrada os dúplex de siRNA para impedir a súa carga en RISC.[20]
Diagnóstico e aplicacións terapéuticas
[editar | editar a fonte]Dicer pode utilizarse para detectar a presenza de tumores no corpo baseándose no nivel de expresión do encima. Un estudo mostrou que en moitos pacientes que tiñan cancro decrecera o nivel de expresión de Dicer. O mesmo estudo mostrou que unha expresión máis baixa de Dicer correlacionábase cun menor tempo de supervivencia do paciente.[11] Pero, como se dixo máis arriba, isto non se cumpre en todos os tipos de cancro. Ademais de ser unha ferramenta de diagnóstico, Dicer pode utilizarse para tratar pacientes por medio de inxeccións intravenosas de siRNA alleo que causen silenciamento xénico.[21]
O siRNA foi administrado de dúas maneiras en especies de mamíferos como o rato. Unha forma é a inxección directa no sistema, que non requiriría a función de Dicer. Outra forma é introducilo indirectamente por medio de plásmidos que codifican curtos ARN en forquita, que son clivados por Dicer orixinando siRNA.[22]
Unha das vantaxes de usar Dicer para producir siRNA terapeuticamente é a especificidade e diversidade de dianas ás que pode afectar en comparación aos antibióticos ou pequenas moléculas inhibidoras que se usan actualmente. En xeral, as pequenas moléculas inhibidoras son difíciles de utilizar debido á súa especificidade e aos seus efectos secundarios pouco tolerables. Os anticorpos son tan específicos coma os siRNA, pero teñen a limitación de que só se poden usar contra ligandos ou receptores da superficie celular. Por outra parte, o principal obstáculo para a inxección de siRNAs é a baixa eficacia na súa captación intracelular.[11] O siRNA inxectado ten unha baixa estabilidade no sangue e causa estimulacións de inmunidade inespecífica.[23] Ademais, producir terapeuticamente miARN carecería de especificidade porque só se necesita un apareamentode bases de 6 a 8 nucleótidos para que o miARN se una ao ARNm.[24]
Proteínas similares a Dicer
[editar | editar a fonte]Os xenomas das plantas codifican proteínas similares a Dicer con funcións similares e dominios proteicos como os dos Dicer de insectos e outros animais. Por exemplo, no organismo modelo Arabidopsis thaliana, atopáronse catro proteínas similares a Dicer, que se denominan de DCL1 a DCL4. A DCL1 está implicada na xeración de miARN e produción de siRNA a partir de repeticións invertidas. A DCL2 crea siRNA a partir de transcritos antisentido que actúan en cis, que colaboran na inmunidade antiviral e defensa. A DCL3 xera siRNA, que intervén na modificación da cromatina e a DCL4 está implicada no metabolismo do siRNA que actúa en trans e no silenciamento de transcritos a nivel postranscricional. Ademais, as DCL 1 e 3 son importantes para a floración de Arabidopsis. En Arabidopsis, o knock-out para a DCL non causa graves problemas de desenvolvemento.
A planta do arroz e a vide tamén producen DCLs, xa que o mecanismo de Dicer é unha estratexia de defensa común de moitos organismos. No arroz evolucionaron outras funcións para os cinco DCLs que produce e estes desempeñan un papel máis importante no funcionamento e desenvolvemento que en Arabidopsis. Ademais, os patróns de expresión difiren entre distintos tipos celulares da planta do arroz, mentres que a expresión en Arabidopsis é máis homoxénea. A expresión de DCL no arroz pode ser afectada por condicións de estrés biolóxico, como a seca, a salinidade e o frío, polo que estes estresantes poden diminuír a resistencia viral das plantas. A diferenza de Arabidopsis, a perda de función das proteínas DCL causa defectos no desenvolvemento do arroz.[25]
Notas
[editar | editar a fonte]- ↑ Bernstein E, Caudy AA, Hammond SM, Hannon GJ (2001). "Role for a bidentate ribonuclease in the initiation step of RNA interference". Nature 409 (6818): 363–6. PMID 11201747. doi:10.1038/35053110.
- ↑ Jaskiewicz L, Filipowicz W (2008). "Role of Dicer in posttranscriptional RNA silencing". Curr. Top. Microbiol. Immunol. 320: 77–97. PMID 18268840. doi:10.1007/978-3-540-75157-1_4.
- ↑ Khraiwesh B, Arif MA, Seumel GI, Ossowski S, Weigel D, Reski R, Frank W (January 2010). "Transcriptional control of gene expression by microRNAs". Cell 140 (1): 111–22. PMID 20085706. doi:10.1016/j.cell.2009.12.023.
- ↑ 4,0 4,1 Lau PW, Potter CS, Carragher B, MacRae IJ (2009). "Structure of the human Dicer-TRBP complex by electron microscopy". Structure 17 (10): 1326–32. PMC 2880462. PMID 19836333. doi:10.1016/j.str.2009.08.013.
- ↑ 5,0 5,1 Macrae IJ, Zhou K, Li F, Repic A, Brooks AN, Cande WZ, Adams PD, Doudna JA (2006). "Structural basis for double-stranded RNA processing by Dicer". Science 311 (5758): 195–8. PMID 16410517. doi:10.1126/science.1121638.
- ↑ "Entrez Gene: DICER1 Dicer1, Dcr-1 homolog (Drosophila)".
- ↑ Matsuda S, Ichigotani Y, Okuda T, Irimura T, Nakatsugawa S, Hamaguchi M (January 2000). "Molecular cloning and characterization of a novel human gene (HERNA) which encodes a putative RNA-helicase". Biochim. Biophys. Acta 1490 (1–2): 163–9. PMID 10786632. doi:10.1016/S0167-4781(99)00221-3.
- ↑ Hammond SM (2005). "Dicing and slicing: the core machinery of the RNA interference pathway". FEBS Lett. 579 (26): 5822–9. PMID 16214139. doi:10.1016/j.febslet.2005.08.079.
- ↑ 9,0 9,1 9,2 Cenik ES, Fukunaga R, Lu G, Dutcher R, Wang Y, Tanaka Hall TM, Zamore PD (2011). "Phosphate and R2D2 restrict the substrate specificity of Dicer-2, an ATP-driven ribonuclease". Mol. Cell 42 (2): 172–84. PMC 3115569. PMID 21419681. doi:10.1016/j.molcel.2011.03.002.
- ↑ Hammond SM, Bernstein E, Beach D, Hannon GJ (2000). "An RNA-directed nuclease mediates post-transcriptional gene silencing in Drosophila cells". Nature 404 (6775): 293–6. PMID 10749213. doi:10.1038/35005107.
- ↑ 11,0 11,1 11,2 11,3 11,4 11,5 Merritt WM, Bar-Eli M, Sood AK (2010). "The dicey role of Dicer: implications for RNAi therapy". Cancer Res. 70 (7): 2571–4. PMC 3170915. PMID 20179193. doi:10.1158/0008-5472.CAN-09-2536.
- ↑ Vermeulen A, Behlen L, Reynolds A, Wolfson A, Marshall WS, Karpilow J, Khvorova A (2005). "The contributions of dsRNA structure to Dicer specificity and efficiency". RNA 11 (5): 674–82. PMC 1370754. PMID 15811921. doi:10.1261/rna.7272305.
- ↑ Watson JD (2008). Molecular Biology of the Gene. San Francisco, CA: Cold Spring Harbor Laboratory Press. pp. 641–648. ISBN 978-0-8053-9592-1.
- ↑ Zeng Y, Yi R, Cullen BR (2003). "MicroRNAs and small interfering RNAs can inhibit mRNA expression by similar mechanisms". Proc. Natl. Acad. Sci. U.S.A. 100 (17): 9779–84. PMC 187842. PMID 12902540. doi:10.1073/pnas.1630797100.
- ↑ Meister G (2011). "Vision: Dicer leaps into view". Nature 471 (7338): 308–9. PMID 21412326. doi:10.1038/471308a.
- ↑ Tarallo V, Hirano Y, Gelfand BD, Dridi S, Kerur N, Kim Y, Cho WG, Kaneko H, Fowler BJ, Bogdanovich S, Albuquerque RJ, Hauswirth WW, Chiodo VA, Kugel JF, Goodrich JA, Ponicsan SL, Chaudhuri G, Murphy MP, Dunaief JL, Ambati BK, Ogura Y, Yoo JW, Lee DK, Provost P, Hinton DR, Núñez G, Baffi JZ, Kleinman ME, Ambati J (2012). "DICER1 loss and Alu RNA induce age-related macular degeneration via the NLRP3 inflammasome and MyD88". Cell 149 (4): 847–59. PMC 3351582. PMID 22541070. doi:10.1016/j.cell.2012.03.036.
- ↑ Tang KF, Ren H (2012). "The role of dicer in DNA damage repair". Int J Mol Sci 13 (12): 16769–78. PMC 3546719. PMID 23222681. doi:10.3390/ijms131216769.
- ↑ Berkhout B, Haasnoot J (2006). "The interplay between virus infection and the cellular RNA interference machinery". FEBS Lett. 580 (12): 2896–902. PMID 16563388. doi:10.1016/j.febslet.2006.02.070.
- ↑ "Mosquito-borne Diseases". National Center for Infections Disease, Center for Disease Control and Prevention. Arquivado dende o orixinal o 31 de xaneiro de 2014. Consultado o 22 April 2014.
- ↑ Bronkhorst AW, van Rij RP (2014). "The long and short of antiviral defense: small RNA-based immunity in insects". Curr Opin Virol 7C: 19–28. PMID 24732439. doi:10.1016/j.coviro.2014.03.010.
- ↑ Kamlah F, Eul BG, Li S, Lang N, Marsh LM, Seeger W, Grimminger F, Rose F, Hänze J (2009). "Intravenous injection of siRNA directed against hypoxia-inducible factors prolongs survival in a Lewis lung carcinoma cancer model". Cancer Gene Ther. 16 (3): 195–205. PMID 18818708. doi:10.1038/cgt.2008.71.
- ↑ "Gene silencing by RNA interference is being used routinely to study gene function in cultured mammalian cells". Life Technologies. Arquivado dende o orixinal o 20 de outubro de 2014. Consultado o 23 April 2014.
- ↑ Schiffelers RM, Ansari A, Xu J, Zhou Q, Tang Q, Storm G, Molema G, Lu PY, Scaria PV, Woodle MC (2004). "Cancer siRNA therapy by tumor selective delivery with ligand-targeted sterically stabilized nanoparticle". Nucleic Acids Res. 32 (19): e149. PMC 528817. PMID 15520458. doi:10.1093/nar/gnh140.
- ↑ Chi SW, Zang JB, Mele A, Darnell RB (2009). "Argonaute HITS-CLIP decodes microRNA–mRNA interaction maps". Nature 460 (7254): 479–86. PMC 2733940. PMID 19536157. doi:10.1038/nature08170.
- ↑ Liu, Q; Feng, Y; Zhu, Z (Aug 2009). "Dicer-like (DCL) proteins in plants.". Functional & integrative genomics 9 (3): 277–86. PMID 19221817. doi:10.1007/s10142-009-0111-5.
