Xenómica comparada

A xenética comparada é un campo da investigación biolóxica na cal se comparan as características xenómicas de diferentes organismos.[2][3] As características xeómicas poden incluír a secuencia de ADN, xenes, orde dos xenes, secuencias regulatorias e outros puntos de referencia estruturais xenómicos.[3] Nesta rama da xenómica, compáranse grandes partes do xenoma ou o xenoma completo resultantes dos proxectos xenoma para estudar semellanzas e diferenzas biolóxicas básicas así como as relacións evolutivas entre organismos.[2][4][5] O principio básico da xenómica comparada é que as características comúns de dous organismos serán a miúdo codificadas por ADN que está conservada evolutivamente entre eles.[6] Por tanto, as estratexias da xenómica comparada empezan facendo algunha forma de aliñamento de secuencias xenómicas e buscando secuencias ortólogas (secuencias que comparten unha ascendencia común) nos xenomas aliñados e comprobando en que medida estas secuencias están conservadas. Baseándose nisto, infirense a evolución do xenoma e a molecular e isto, á súa vez, pode poñerse en contexto, por exemplo, na evolución fenotípica ou na xenética de poboacións.[7]
Empezou virtualmente axiña que se puido dispoñer, en 1995, dos xenomas completos de dous organismos (que foron os xenomas das bacterias Haemophilus influenzae e Mycoplasma genitalium), e agora a xenómica comparada é un compoñente estándar da análise de cada nova secuencia de xenómica.[2][8] Coa exposición no número de proxectos xenoma debido aos avances nas tecnoloxías de secuenciación do ADN, especialmente os métodos de secuenciación de seguinte xeración a finais da década do 2000, este campo fíxose mais sofisticado, facendo posible tratar con moitos xenomas nun só estudo.[9] A xenómica comparada revelou a existencia de grandes niveis de similitude entre organismos estreitamente relacionados, como os humanos e os chimpancés e, máis sorprendentemente, a semellanza entre organismos que teñen unha relación moi distante, como humanos e o lévedo Saccharomyces cerevisiae.[4] Tamén mostrou a extrema diversidade da composición xénica en diferentes liñaxes evolutivas.[8]
Historia
[editar | editar a fonte]A xenómica comparativa ten as súas raíces na comparación dos xenomas de virus que se empezou a facer a inicios da década de 1980.[8] Por exemplo, comparáronse os pequenos virus de ARN que infectan animais (picornavirus) e plantas (virus do mosaico de Vigna unguiculata) e viuse que compartían unha similitude de secuencia significativa e, en parte, a orde dos xenes.[10] En 1986, publicouse o primeiro estudo xenómico comparativo a grande escala que comparaba os xenomas do virus da varicela-zóster e do virus de Epstein-Barr, que contñan máis de 100 xenes cada un.[11]
A primeira secuencia xenómica completa dun organismo celular foi o da bacteria Haemophilus influenzae Rd, que se publicou en 1995.[12] O segundo artigo de secuenciación dun xenoma bacteriano que apareceu foi o da pequena bacteria parasita Mycoplasma genitalium publicado ese mesmo ano.[13] Empezando por ese artigo, os informes de novos xenomas levaron inevitablemente a facer estudos de xenómica comparada.[8]
Xenomas microbianos. O primeiro sistema de comparación de xenomas completos de alta resolución de xenomas microbianos de 10-15kbp desenvolvérono en 1998 Art Delcher, Simon Kasif e Steven Salzberg cos seus colaboradores do Instituto para a Investigación Xenómica (TIGR) e aplicárono á comparación de xenomas enteiros de organismos microbianos moi relacionados. O sistema denomínase MUMMER e describiuse nunha publicación na revista Nucleic Acids Research en 1999. O sistema axuda a identificar grandes redistribucións xenómicas, mutacións dunha soa base, inversións, expansións de repeticións en tándem e outros polimorfismos. En bacterias, MUMMER permite a identificación de polimorfismos responsables da virulencia, patoxenicidade e resistencia a antibióticos. O sistema foi tamén aplicado no Proxecto do Organismo Mínimo no TIGR e seguidamente a moitos outros proxectos xenómicos comparativos.
Xenomas eucariotas. O lévedo de panadaría Saccharomyces cerevisiae foi o primeiro eucariota do que se publicou a secuencia xenómica completa, en 1996.[14] Despois da publicación do xenoma do verme nematodo Caenorhabditis elegans en 1998[15] e xunto co da mosca do vinagre Drosophila melanogaster en 2000,[16] Gerald M. Rubin e o seu equipo publicaron un artigo titulado "Comparative Genomics of the Eukaryotes" ("Xenómica comparativa dos eucariotas"), no cal comparaban os xenomas dos eucariotas D. melanogaster, C. elegans e S. cerevisiae, así como o do procariota H. influenzae.[17] Ao mesmo tempo, Bonnie Berger, Eric Lander, e os seus equipos publicaron un artigo sobre a comparación dos xenomas completos dos ratos e humanos.[18]
Coa publicación de grandes xenomas de vertebrados na década de 2000, incluíndo o humano, o do peixe Takifugu rubripes e o do rato doméstico, liberáronse os resultados precomputados de comparacións de grandes xenomas que se podían descargar e visualizar nun buscador de xenomas. En lugar de realizar as súas propias análises, a maioría dos biólogos podían acceder a estas grandes comparacións entre especies e evitar os inconvenientes prácticos causados polo tamaño dos xenomas.[19]
Os métodos de secuenciación de seguinte xeración, que se empezaron a usar en 2007, produciuse unha enorme cantidade de datos xenómicos e isto permitiu xerar á vez múltiples secuencias xenómicas borrador (procariotas). Estes métodos poden tamén revelar rapidamente polimorfismos dun só nucleótido, insercións e delecións mapando lecturas non ensambladas contra un xenoma de referencia ben anotado e así proporcionar unha lista de posibles diferenzas xénicas que poden ser a base de calquera variación funcional entre cepas.[9]
Principios evolutivos
[editar | editar a fonte]- Artigo principal: Evolución.
Unha característica básica da bioloxía é a evolución. A teoría evolutiva é tamén o fundamento teórico da xenómica comparativa e ao mesmo tempo os resultados da xenómica comparativa enriqueceron dunha forma sen precedentes e desenvolveron a teoría da evolución. Cando se comparan dúas ou máis secuencias xenómicas, poden deducirse as relacións evolutivas das secuencias nunha árbore filoxenética. Baseándose nunha variedade de datos xenómicos biolóxicos e no estudo de procesos de evolución vertical e horizontal, pódense comprender as partes vitais da estrutura xénica e da súa función regulatoria.
A similitude dos xenomas relacionados é a base da xenómica comparativa. Se dúas criaturas teñen un antepasado común recente, as diferenzas entre os xenomas das dúas especies evolucionaron do xenoma do antepasado. Canto máis próxima é a relación entre os dous organismos, maiores son as semellanzas entre os seus xenomas. Se hai unha relación próxima entre eles, entón os seus xenomas presentarán un comportamento linear (sintenia), concretamente algunhas das secuencias xenéticas estararán conservadas. Así, as xecuencias xenómicas poden utilizarse para identificar a función xénica ao analizar a súa homoloxia (similitude de secuencia) con xenes de función coñecida.

As secuencias ortólogas son secuencias relacionadas de diferentes especies: un xene existe na especie orixinal, a especie divídese en dúas especies (especiación), así que os xenes das novas especies son ortólogos da secuencia que había na especie orixinal. As secuencias parálogas están separadas por clonación xénica (duplicación xénica): se un xene particular do xenoma se copia, entón a copia das súas secuencias é paráloga do xene orixinal. Un par de secuencias ortólogas denomínase par ortólogo (ortólogos); un par de secuencias parálogas denomínase par colateral (parálogos). Os pares ortólogos xeralmente teñen unha función idéntica ou similar, o cal non é necesariamente o caso dos pares colaterais, porque nos pares colaterais as secuencias tenden a evolucionar adquirindo diferentes funcións.
A xenómica comparada aproveita tanto as semellanzas coma as diferenzas nas proteínas, ARN e rexións regulatorias de diferentes organismos para inferir como actuou a selección sobre estes elementos. Os elementos que son responsables das semellanzas entre diferentes especies deberían estar conservados ao longo do tempo (selección estabilizante), mentres que os elementos responsables das diferenzas entre especies deberían ser diverxentes (selección positiva). Finalmente, os elementos que non son importantes para o éxito evolutivo do organismo non serán conservados (a selección é neutra).
Un dos obxectivos importantes neste eido é a identificación dos mecanismos da evolución do xenoma eucariota. Porén, adoita ser complicado pola multiplicade de eventos que tiveron lugar ao longo da historia de liñaxes individuais, deixando só trazas distorsionadas e superpostas no xenoma de cada organismo vivo. Por esta razón os estudos de xenómica comparada de pequenos organismos modelo (por exemplo o modelo Caenorhabditis elegans e o estreitamente relacionado Caenorhabditis briggsae) son de grande importancia para aumentar a nosa comprensión dos mecanismos xerais da evolución.[20][21]
Métodos
[editar | editar a fonte]Os métodos computacionais son necesarios para as comparacións xenómicas dada a gran cantidade de datos codificados nos xenomas. Moitas ferramentas están agora dispoñibles publicamente, e van desde comparacións de xenoma completo a análises de expresión xénica.[22] Isto inclúe métodos para os sistemas e o control, teoría da información, análise de cadeas (strings) e minaría de datos.[23] As estratexias computacionais seguirán sendo fundamentais para a investigación e ensinanza, especialmente cando a ciencia da información e a bioloxía xenómica se ensinan conxuntamente.[24]

A xenómica comparativa empeza con comparacións básicas do tamaño do xenoma e densidade de xenes. Por exemplo, o tamaño do xenoma é importante para a capacidade de codificación e posiblemente por razóns regulatorias. Unha alta densidade de xenes facilita a anotación do xenoma, a análise de selección ambiental. En contraste, unha densidade baixa de xenes dificulta o mapado de enfermidades xenéticas, como ocorre no xenoma humano.
Aliñamento de secuencias
[editar | editar a fonte]Os aliñamentos utilízanse para captar información sobre secuencias similares como os antepasados, a descendencia evolutiva común ou unha común estrutura e función. Os aliñamentos poden facerse tanto para secuencias xenéticas coma proteicas.[26][27] Os aliñamentos consisten en aliñamentos locais ou globais por pares e aliñamentos de secuenias múltiples. Un xeito de encontrar aliñamentos globais é usar un algoritmo de programación dinámico chamado algoritmo de Needleman–Wunsch. Este algoritmo pode modificarse e usarse para atopar aliñamentos locais.

Reconstrución filoxenética
[editar | editar a fonte]Outro método computacional de xenómica comparada é a reconstrución filoxenética. Utilízase para describir as relacións evolutivas en termos de devanceiros comúns. As relacións represéntanse xeralmente nunha árbore chamada árbore filoxenética. De xeito similar, a teoría coalescente é un modleo retrospectivo para rastrexar alelos dun xene nunha poboación ata unha soa copia ancestral compartida polos membros da poboación. Isto tamén se coñece como o antepasado común máis recente. A análise baseada na teoría da coalescencia trata de predicir a cantidade de tempo entre a introdución dunha mutación e un particular alelo ou distribución xénica nunha poboación. Este período de tempo é igual ao tempo que hai que existiu o antepasado común máis recente. As relacións de herdanza visualízanse en forma similar a unha árbore filoxenética. A coalescencia (ou xenealoxía dos xenes) pode visualizarse usando dendrogramas.[28]
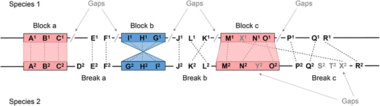
Mapas xenómicos
[editar | editar a fonte]Un método adicional en xenómica comparada é o mapado xenético. No mapado xenético a visualización da sintenia é un modo de ver a conservación da orde dos xenes nos cromosomas. Utilízase xeralmente para cromosomas de especies relacionadas, ambas as dúas orixinadas a partir dun antepasado común.[30] Este e outros métodos poden botar luz sobre a historia evolutiva. Un estudo recente usou a xenómica comparada para reconstruír 16 cariotipos ancestrais na filoxenia dos mamíferos. A reconstrución computacional mostra como os cromosomas se rearranxaron durante a evolución dos mamíferos. Proporciona información sobre a conservación de rexións seleccionadas a míúdo asociadas con procesos de control do desenvolvemento. Ademais, axudou a comprender a evolución dos cromosomas e enfermidades xenéticas asociadas con rearranxos do ADN.[Cómpre referencia]

Ferramentas
[editar | editar a fonte]Están desenvolvéndose rapidamente ferramentas computacionais para analizar secuencias e xenomas completos debido á dispoñibilidade de grandes cantidades de datos xenómicos. A mesmo tempo, as ferramentas de análise comparativa están a ser melloradas. Nos retos sobre as análises, é moi importante para visualizar os resultados comparativos.[32]
A visualización da conservación de secuencias é un duro traballo de análise de secuencias comparativa. É moi ineficaz examinar o aliñamento de longas rexións xenómicas longas manualmente. Os buscadores de xenoma de Internet proporcionan moitas ferramentas útiles para investigar as secuencias xenómicas debido a integrar toda a información biolóxica baseada en secuencias sobre rexións xenómicas. Cando extraemos grandes cantidades de datos relevantes bioloxicamente, poden ser moi doados de usar e levan menos tempo.[32]
- UCSC Browser: Este sitio contén a secuencia de referencia e ensamblaxes de borrador de traballo para unha gran colección de xenomas.[33]
- Ensembl: O proxecto Ensembl produce bases de datos xenómicos para vertebrados e outras especies eucariotas e fai que esta información estea dispoñible libremente on line.[34]
- MapView: O Map Viewer proporciona unha ampla variedade de mapado xenómico e datos de secuenciación.[35]
- VISTA é unha suite de progamas completa e bases de datos para a análise comparativa de secuencias xenómicas. Construíuse para visualizar os resultados de análises comparativas baseadas en aliñamentos de ADN. A presentación de datos comparativos xerados por VISTA pode facilmente adaptar datos tanto a pequena coma a grande escala.[36]
- BlueJay Genome Browser: unha ferramenta de visualización autónoma para a visión multiescala de xenomas anotados e outros elementos xenómicos.[37]
Unha vantaxe de utilizar ferramentas on line é que estes sitios web están a ser desenvolvidos e actualizados decote. Hai moitas novas configuracións e contidos que poden usarse on line para mellorar a eficiencia.[32]
Aplicacións seleccionadas
[editar | editar a fonte]Agricultura
[editar | editar a fonte]A agricultura é un campo que recolle os beneficios da xenómica comparada. Identificar os loci de xenes vantaxosos é un paso clave na mellora vexetal, que son optimizados para que teñan un maior rendemento, rendibilidade económica, calidade e resistencia a enfermidades. Por exemplo, un estudo de asociación de xenoma completo realizado en 517 variedades naturais de arroz revelou 80 loci asociados con varios tipos de rendemento agronómico, como o peso dos grans, o contido en amilosa e a tolerancia á seca. Moitos dos loci non foran caracterizados previamente.[38] Esta metodoloxía non só é poderosa, senón tamén rápida. Os métodos anteriores para identificar loci asociados co rendemento agronómico requirían varias xeracións de cultivo coidadosamente monitorizado das cepas parentais, un esforzo que leva moito tempo que é innecesario para estudos de xenómica comparada.[39]
Medicina
[editar | editar a fonte]Desenvolvemento de vacinas
[editar | editar a fonte]O campo da medicina tamén se beneficia do estudo da xenómica comparada. Nunha estratexia coñecida como vacinoloxía inversa, os investigadores poden descubrir antíxenos candidatos para o desenvolvemento de vacinas analizando o xenoma dun patóxeno ou familia de patóxenos.[40] Aplicar unha estratexia de xenómica comparada analizando os xenomas de varios patóxenos relacionados pode levar ao desenvolvemento de vacinas que son multiprotectoras. Un equipo de investigadores empregou esa estratexia para crear unha vacina universal para estreptococos do grupo B, un grupo de bacterias responsables de graves infeccións neonatais.[41] A xenómica comparada pode tamén usarse para xerar especificidade nas vacinas contra patóxenos que están estreitamente relacionados con microorganismos comensais. Por exemplo, utilizouse unha análise xenómica comparativa de cepas do comensal e patóxenas de E. coli para identificar xenes específicos de patóxeno como base para atopar antíxenos que orixinan unha resposta inmune contra cepas patóxenas pero non contra as comensais.[42] En maio de 2019, usando o Global Genome Set, un equipo do Reino Unido e Australia secuenciou miles de illamentos recollidos globalmente de esteptococos do grupo A, proporcionando posibles etiquetas para desenvolver unha vacina contra o patóxeno, tamén coñecido como S. pyogenes.[43]

Modelos de ratos en inmunoloxía
[editar | editar a fonte]As células T (tamén chamadas linfocitos T ou timocitos) son células inmunitarias que crecen a partir de células nais da medula ósea. Axudan a defender o corpo de infeccións e poden loitar contra o cancro. Debido ás súas características morfolóxicas, fisiolóxicas e xenéticas as semellanzas entre humanos, ratos e ratas, estas foron durante moito tempo as especies preferidas para a investigación biomédica en modelos animais. A investigación en medicina comparada está dándonos a capacidade de usar información dunha especie para comprender o mesmo proceso noutras. Podemos obter novos coñecementos en vías moleculares comparando as células T humanas e de ratos e os seus efectos no sistema inmune utilizando xenómica comparada. Para comprender os seus receptores de células T (TCRs) e os seus xenes, Glusman realizou investigacións sobre a secuenciación dos loci TCR de humanos e ratos. Os xenes TCR son ben coñecidos e serven como unha fonte significativa para estudar a xenómica funcional e comprender como contribúen os xenes e rexións interxénicas do xenoma aos procesos biolóxicos.[44]
Os receptores inmunes de células T son importantes para estudar o mundo dos patóxenos no sistema inmunitario celular. Unha das razóns para secuenciar os loci TCR humanos e de ratos era emparellar as secuencias da familia de xenes ortólogos e descubrir as áreas conservadas usando xenómica comparada. Pénsase que estes reflectirían dous tipos de información biolóxica: (1) exóns e (2) secuencias regulatorias. De feito, con este método poderían identificarse a maioría dos exóns V, D, J, e C. As rexións variables están codificadas por múltiples elementos do ADN únicos que son recolocados e conectados durante a diferenciación das células T: elementos variable (V), de diversidade (D) e de unión (J) [Figura 1]. Porén, varios bloques conservados non codificantes curtos están en gran medida agrupados no xenoma de humanos e ratos nunha rexión de 200 bp [Figura 2]; identificáronse os chamados amplificadores 3′ (enhancers) no TCR, e unha rexión conservada de 100 bp no intrón J do rato viuse seguidamente que tiña unha función regulatoria.

As comparacións de secuencias xenómicas dentro de cada sitio físico ou localización dun xene específico nun cromosoma (locus) e a través de especies permite a investigación doutros mecanismos e outros sinais regulatorios. Suxeriuse unha nova hipótese sobre a evolución dos TCRs, que terá que ser comprobada (e mellorada) por comparación co complemento do xene TCR doutras especies de vertebrados. Unha investigación xenómica comparativa de humanos e ratos permitirá o descubrimento e anotación de moitos outros xenes, así como a identificación noutras especies de secuencias regulatorias.[44]
Investigación
[editar | editar a fonte]A xenómica comparada tamén abre novos camiños noutras áreas de investigación. Como a tecnoloxía de secuenciación do ADN se fixo mais accesible, o número de xenomas secuenciados foi crecendo. Co crecente depósito de datos xenómicos dispoñibles, a potencia da inferencia da xenómica comparativa medrou tamén.
Un caso notable deste incremento da potencia pódese ver nunha investigación recente sobre primates. Os métodos xenómicos comparativos permitiron reunir información sobre variación xenética, expresión diferencial de xenes e dinámica evolutiva en primates que era indiscernible usando datos e métodos previos.[45]
Proxecto Xenoma de Grandes Simios
[editar | editar a fonte]O Proxecto Xenoma de Grandes Simios utilizou métodos de xenómica comparativa para investigar a variación xenética en seis especies de grandes simios, atopando niveis saudables de variación na súa poza xénica malia que os seus tamaños de poboaión están diminuíndo.[46] Outro estudo mostrou que os padróns de metilación do ADN, que son un mecanismo de regulación da expresión xénica, difiren no córtex prefrontal de humanos fronte a chimpancés e esta diferenza estaba implicada na diverxencia evolutiva das dúas especies.[47]
Notas
[editar | editar a fonte]- ↑ Darling AE, Miklós I, Ragan MA (xullo de 2008). "Dynamics of genome rearrangement in bacterial populations". PLOS Genetics 4 (7): e1000128. PMC 2483231. PMID 18650965. doi:10.1371/journal.pgen.1000128.
- ↑ 2,0 2,1 2,2 Touchman J (2010). "Comparative Genomics". Nature Education Knowledge 3 (10): 13.
- ↑ 3,0 3,1 Xia X (2013). Comparative Genomics. SpringerBriefs in Genetics. Heidelberg: Springer. ISBN 978-3-642-37145-5. doi:10.1007/978-3-642-37146-2.
- ↑ 4,0 4,1 Russel PJ, Hertz PE, McMillan B (2011). Biology: The Dynamic Science (2ª ed.). Belmont, CA: Brooks/Cole. pp. 409–410.
- ↑ Primrose SB, Twyman RM (2003). Principles of Genome Analysis and Genomics (3rd ed.). Malden, MA: Blackwell Publishing. ISBN 9781405101202.
- ↑ Hardison RC (novembro de 2003). "Comparative genomics". PLOS Biology 1 (2): E58. PMC 261895. PMID 14624258. doi:10.1371/journal.pbio.0000058.
- ↑ Ellegren H (novembro de 2008). "Comparative genomics and the study of evolution by natural selection". Molecular Ecology 17 (21): 4586–4596. PMID 19140982. doi:10.1111/j.1365-294X.2008.03954.x.
- ↑ 8,0 8,1 8,2 8,3 Koonin EV, Galperin MY (2003). Sequence - Evolution - Function: Computational approaches in comparative genomics. Dordrecht: Springer Science+Business Media.
- ↑ 9,0 9,1 Hu B, Xie G, Lo CC, Starkenburg SR, Chain PS (novembro de 2011). "Pathogen comparative genomics in the next-generation sequencing era: genome alignments, pangenomics and metagenomics". Briefings in Functional Genomics 10 (6): 322–333. PMID 22199376. doi:10.1093/bfgp/elr042.
- ↑ Argos P, Kamer G, Nicklin MJ, Wimmer E (setembro de 1984). "Similarity in gene organization and homology between proteins of animal picornaviruses and a plant comovirus suggest common ancestry of these virus families". Nucleic Acids Research 12 (18): 7251–7267. PMC 320155. PMID 6384934. doi:10.1093/nar/12.18.7251.
- ↑ McGeoch DJ, Davison AJ (maio de 1986). "DNA sequence of the herpes simplex virus type 1 gene encoding glycoprotein gH, and identification of homologues in the genomes of varicella-zoster virus and Epstein-Barr virus". Nucleic Acids Research 14 (10): 4281–4292. PMC 339861. PMID 3012465. doi:10.1093/nar/14.10.4281.
- ↑ Fleischmann RD, Adams MD, White O, Clayton RA, Kirkness EF, Kerlavage AR, et al. (xullo de 1995). "Whole-genome random sequencing and assembly of Haemophilus influenzae Rd". Science 269 (5223): 496–512. Bibcode:1995Sci...269..496F. PMID 7542800. doi:10.1126/science.7542800.
- ↑ Fraser CM, Gocayne JD, White O, Adams MD, Clayton RA, Fleischmann RD, et al. (outubro de 1995). "The minimal gene complement of Mycoplasma genitalium". Science 270 (5235): 397–403. Bibcode:1995Sci...270..397F. PMID 7569993. doi:10.1126/science.270.5235.397.
- ↑ Goffeau A, Barrell BG, Bussey H, Davis RW, Dujon B, Feldmann H, et al. (outubro de 1996). "Life with 6000 genes". Science 274 (5287): 546, 563–546, 567. Bibcode:1996Sci...274..546G. PMID 8849441. doi:10.1126/science.274.5287.546.
- ↑ The C. elegans Sequencing Consortium (decembro de 1998). "Genome sequence of the nematode C. elegans: a platform for investigating biology". Science 282 (5396): 2012–2018. Bibcode:1998Sci...282.2012.. PMID 9851916. doi:10.1126/science.282.5396.2012. Erro no estilo Vancouver: non-Latin character (Axuda)
- ↑ Adams MD, Celniker SE, Holt RA, Evans CA, Gocayne JD, Amanatides PG, et al. (marzo de 2000). "The genome sequence of Drosophila melanogaster". Science 287 (5461): 2185–2195. Bibcode:2000Sci...287.2185.. PMID 10731132. doi:10.1126/science.287.5461.2185.
- ↑ Rubin GM, Yandell MD, Wortman JR, Gabor Miklos GL, Nelson CR, Hariharan IK, et al. (marzo de 2000). "Comparative genomics of the eukaryotes". Science 287 (5461): 2204–2215. Bibcode:2000Sci...287.2204.. PMC 2754258. PMID 10731134. doi:10.1126/science.287.5461.2204.
- ↑ Batzoglou S, Pachter L, Mesirov JP, Berger B, Lander ES (xullo de 2000). "Human and mouse gene structure: comparative analysis and application to exon prediction". Genome Research 10 (7): 950–958. PMC 310911. PMID 10899144. doi:10.1101/gr.10.7.950.
- ↑ Ureta-Vidal A, Ettwiller L, Birney E (abril de 2003). "Comparative genomics: genome-wide analysis in metazoan eukaryotes". Nature Reviews. Genetics 4 (4): 251–262. PMID 12671656. doi:10.1038/nrg1043.
- ↑ Stein LD, Bao Z, Blasiar D, Blumenthal T, Brent MR, Chen N, et al. (novembro de 2003). "The genome sequence of Caenorhabditis briggsae: a platform for comparative genomics". PLOS Biology 1 (2): E45. PMC 261899. PMID 14624247. doi:10.1371/journal.pbio.0000045.
- ↑ "Newly Sequenced Worm a Boon for Worm Biologists". PLOS Biology 1 (2): e4. 2003. doi:10.1371/journal.pbio.0000044.

- ↑ Cristianini N, Hahn M (2006). Introduction to Computational Genomics. Cambridge University Press. ISBN 978-0-521-67191-0.
- ↑ Pratas D, Silva RM, Pinho AJ, Ferreira PJ (maio de 2015). "An alignment-free method to find and visualise rearrangements between pairs of DNA sequences". Scientific Reports 5: 10203. Bibcode:2015NatSR...510203P. PMC 4434998. PMID 25984837. doi:10.1038/srep10203.
- ↑ Via A, De Las Rivas J, Attwood TK, Landsman D, Brazas MD, Leunissen JA, et al. (outubro de 2011). "Ten simple rules for developing a short bioinformatics training course". PLOS Computational Biology 7 (10): e1002245. Bibcode:2011PLSCB...7E2245V. PMC 3203054. PMID 22046119. doi:10.1371/journal.pcbi.1002245.

- ↑ Damas J, Corbo M, Kim J, Turner-Maier J, Farré M, Larkin DM, et al. (outubro de 2022). "Evolution of the ancestral mammalian karyotype and syntenic regions". Proceedings of the National Academy of Sciences of the United States of America 119 (40): e2209139119. PMC 9550189. PMID 36161960. doi:10.1073/pnas.2209139119.
- ↑ Altschul SF, Pop M (2017). "Sequence Alignment". En Rosen KH, Shier DR, Goddard W. Handbook of Discrete and Combinatorial Mathematics (2nd ed.). Boca Raton (FL): CRC Press/Taylor & Francis. ISBN 978-1-58488-780-5. PMID 29206392. Consultado o 2022-12-18.
- ↑ Prjibelski AD, Korobeynikov AI, Lapidus AL (2019-01-01). "Sequence Analysis". En Ranganathan S, Gribskov M, Nakai K, Schönbach C. Encyclopedia of Bioinformatics and Computational Biology (en inglés). Oxford: Academic Press. pp. 292–322. ISBN 978-0-12-811432-2. doi:10.1016/b978-0-12-809633-8.20106-4.
- ↑ Haubold B, Wiehe T (September 2004). "Comparative genomics: methods and applications". Die Naturwissenschaften 91 (9): 405–421. PMID 15278216. doi:10.1007/s00114-004-0542-8.
- ↑ Liu D, Hunt M, Tsai IJ (January 2018). "Inferring synteny between genome assemblies: a systematic evaluation". BMC Bioinformatics 19 (1): 26. PMC 5791376. PMID 29382321. doi:10.1186/s12859-018-2026-4.
- ↑ Duran C, Edwards D, Batley J (2009). "Genetic maps and the use of synteny". Plant Genomics. Methods in Molecular Biology 513. pp. 41–55. ISBN 978-1-58829-997-0. PMID 19347649. doi:10.1007/978-1-59745-427-8_3.
- ↑ Damas J, Corbo M, Kim J, Turner-Maier J, Farré M, Larkin DM, et al. (outubro de 2022). "Evolution of the ancestral mammalian karyotype and syntenic regions". Proceedings of the National Academy of Sciences of the United States of America 119 (40): e2209139119. PMC 9550189. PMID 36161960. doi:10.1073/pnas.2209139119.
- ↑ 32,0 32,1 32,2 Bergman NH (2007). Bergman NH, ed. Comparative Genomics: Volumes 1 and 2. Totowa, New Jersey: Humana Press. ISBN 978-193411-537-4. PMID 21250292.
- ↑ "UCSC Browser".
- ↑ "Ensembl Genome Browser". Arquivado dende o orixinal o 2013-10-21.
- ↑ "Map Viewer".
- ↑ "VISTA tools".
- ↑ Soh J, Gordon PM, Sensen CW (marzo de 2012). "The Bluejay genome browser". Current Protocols in Bioinformatics (John Wiley & Sons, Inc.) 37: Unit10.9. ISBN 9780471250951. PMID 22389011. doi:10.1002/0471250953.bi1009s37.
- ↑ Huang X, Wei X, Sang T, Zhao Q, Feng Q, Zhao Y, et al. (novembro de 2010). "Genome-wide association studies of 14 agronomic traits in rice landraces". Nature Genetics 42 (11): 961–967. PMID 20972439. doi:10.1038/ng.695.

- ↑ Morrell PL, Buckler ES, Ross-Ibarra J (decembro de 2011). "Crop genomics: advances and applications". Nature Reviews. Genetics 13 (2): 85–96. PMID 22207165. doi:10.1038/nrg3097.

- ↑ Seib KL, Zhao X, Rappuoli R (outubro de 2012). "Developing vaccines in the era of genomics: a decade of reverse vaccinology". Clinical Microbiology and Infection. 18 Suppl 5 (SI): 109–116. PMID 22882709. doi:10.1111/j.1469-0691.2012.03939.x.

- ↑ Maione D, Margarit I, Rinaudo CD, Masignani V, Mora M, Scarselli M, et al. (xullo de 2005). "Identification of a universal Group B streptococcus vaccine by multiple genome screen". Science 309 (5731): 148–150. Bibcode:2005Sci...309..148M. PMC 1351092. PMID 15994562. doi:10.1126/science.1109869.

- ↑ Rasko DA, Rosovitz MJ, Myers GS, Mongodin EF, Fricke WF, Gajer P, et al. (outubro de 2008). "The pangenome structure of Escherichia coli: comparative genomic analysis of E. coli commensal and pathogenic isolates". Journal of Bacteriology 190 (20): 6881–6893. PMC 2566221. PMID 18676672. doi:10.1128/JB.00619-08.

- ↑ "Group a Streptococcus Vaccine Target Candidates Identified from Global Genome Set". 28 de maio de 2019.
- ↑ 44,0 44,1 44,2 44,3 Glusman G, Rowen L, Lee I, Boysen C, Roach JC, Smit AF, et al. (setembro de 2001). "Comparative genomics of the human and mouse T cell receptor loci". Immunity 15 (3): 337–349. PMID 11567625. doi:10.1016/s1074-7613(01)00200-x.
- ↑ Rogers J, Gibbs RA (maio de 2014). "Comparative primate genomics: emerging patterns of genome content and dynamics". Nature Reviews. Genetics 15 (5): 347–359. PMC 4113315. PMID 24709753. doi:10.1038/nrg3707.

- ↑ Prado-Martinez J, Sudmant PH, Kidd JM, Li H, Kelley JL, Lorente-Galdos B, et al. (xullo de 2013). "Great ape genetic diversity and population history". Nature 499 (7459): 471–475. Bibcode:2013Natur.499..471P. PMC 3822165. PMID 23823723. doi:10.1038/nature12228.

- ↑ Zeng J, Konopka G, Hunt BG, Preuss TM, Geschwind D, Yi SV (setembro de 2012). "Divergent whole-genome methylation maps of human and chimpanzee brains reveal epigenetic basis of human regulatory evolution". American Journal of Human Genetics 91 (3): 455–465. PMC 3511995. PMID 22922032. doi:10.1016/j.ajhg.2012.07.024.

Véxase tamén
[editar | editar a fonte]utros artigos
[editar | editar a fonte]- Minaría de datos
- Evolución molecular
- Anatomía comparativa
- Homoloxía
- Minaría de secuencias
- Análise de secuencias libre de aliñamento
Bibliografía
[editar | editar a fonte]- Kellis M, Patterson N, Endrizzi M, Birren B, Lander ES (maio de 2003). "Sequencing and comparison of yeast species to identify genes and regulatory elements". Nature 423 (6937): 241–254. Bibcode:2003Natur.423..241K. PMID 12748633. doi:10.1038/nature01644.
- Cliften P, Sudarsanam P, Desikan A, Fulton L, Fulton B, Majors J, et al. (xullo de 2003). "Finding functional features in Saccharomyces genomes by phylogenetic footprinting". Science 301 (5629): 71–76. Bibcode:2003Sci...301...71C. PMID 12775844. doi:10.1126/science.1084337.
- Boffelli D, McAuliffe J, Ovcharenko D, Lewis KD, Ovcharenko I, Pachter L, Rubin EM (febreiro de 2003). "Phylogenetic shadowing of primate sequences to find functional regions of the human genome". Science 299 (5611): 1391–1394. PMID 12610304. doi:10.1126/science.1081331.
- Dujon B, Sherman D, Fischer G, Durrens P, Casaregola S, Lafontaine I, et al. (xullo de 2004). "Genome evolution in yeasts". Nature 430 (6995): 35–44. Bibcode:2004Natur.430...35D. PMID 15229592. doi:10.1038/nature02579.
- Filipski A, Kumar S (2005). "Comparative genomics in eukaryotes". En Gregory TR. The Evolution of the Genome. San Diego: Elsevier. pp. 521–583.
- Gregory TR, DeSalle R (2005). "Comparative genomics in prokaryotes". En Gregory TR. The Evolution of the Genome. San Diego: Elsevier. pp. 585–675.
- Xie X, Lu J, Kulbokas EJ, Golub TR, Mootha V, Lindblad-Toh K, et al. (marzo de 2005). "Systematic discovery of regulatory motifs in human promoters and 3' UTRs by comparison of several mammals". Nature 434 (7031): 338–345. Bibcode:2005Natur.434..338X. PMC 2923337. PMID 15735639. doi:10.1038/nature03441.
- Champ PC, Binnewies TT, Nielsen N, Zinman G, Kiil K, Wu H, et al. (marzo de 2006). "Genome update: purine strand bias in 280 bacterial genomes". Microbiology 152 (Pt 3): 579–583. PMID 16514138. doi:10.1099/mic.0.28637-0.
- Kumar L, Breakspear A, Kistler C, Ma LJ, Xie X (marzo de 2010). "Systematic discovery of regulatory motifs in Fusarium graminearum by comparing four Fusarium genomes". BMC Genomics 11: 208. PMC 2853525. PMID 20346147. doi:10.1186/1471-2164-11-208.
- Batzoglou S, Pachter L, Mesirov JP, Berger B, Lander ES (xullo de 2000). "Human and mouse gene structure: comparative analysis and application to exon prediction". Genome Research 10 (7): 950–958. PMC 310911. PMID 10899144. doi:10.1101/gr.10.7.950.
Ligazóns externas
[editar | editar a fonte]- Genomes OnLine Database (GOLD)
- Genome News Network
- JCVI Comprehensive Microbial Resource
- Pathema: A Clade Specific Bioinformatics Resource Center
- CBS Genome Atlas Database Arquivado 16 de maio de 2016 en Arquivo.pt
- The UCSC Genome Browser
- The U.S. National Human Genome Research Institute
- Ensembl The Ensembl Genome Browser
- Genolevures, comparative genomics of the Hemiascomycetous yeasts
- Phylogenetically Inferred Groups (PhIGs), a recently developed method incorporates phylogenetic signals in building gene clusters for use in comparative genomics.
- Metazome Arquivado 2006-08-10 en Wayback Machine., a resource for the phylogenomic exploration and analysis of Metazoan gene families.
- IMG The Integrated Microbial Genomes system, for comparative genome analysis by the DOE-JGI.
- Dcode.org Dcode.org Comparative Genomics Center.
- SUPERFAMILY Arquivado 17 de outubro de 2008 en Wayback Machine. Protein annotations for all completely sequenced organisms
- Comparative Genomics
- Blastology and Open Source: Needs and Deeds
- Alignment-free comparative Genomics tool
