Dinucleótido de nicotinamida e adenina
| Dinucleótido de nicotinamida e adenina | |
|---|---|
 | |
 | |
Outros nomes Difosfopiridina nucleótido (DPN+), Coencima I | |
| Identificadores | |
| Número CAS | 53-84-9, 58-68-4 (NADH) |
| PubChem | 925 |
| ChemSpider | 5681 |
| UNII | 0U46U6E8UK |
| DrugBank | DB00157 |
| KEGG | C00003 |
| ChEBI | CHEBI:13389 |
| ChEMBL | CHEMBL1628272 |
| Ligando IUPHAR | 2451 |
| Número RTECS | UU3450000 |
| Imaxes 3D Jmol | Image 1 |
| |
| |
| Propiedades | |
| Fórmula molecular | C21H27N7O14P2 |
| Masa molecular | 663,43 g/mol |
| Aspecto | po branco |
| Punto de fusión | 160 °C |
| Perigosidade | |
| Principais perigos | Non perigosa |
| NFPA 704 | |
Se non se indica outra cousa, os datos están tomados en condicións estándar de 25 °C e 100 kPa. | |
O dinucleótido de nicotinamida e adenina[1], tamén chamado nicotinamida adenina dinucleótido ou nicotín adenín dinucleótido, abreviado como NAD+ (forma oxidada) ou NADH (forma reducida), é un dos coencimas máis importantes da célula, que intervén en reaccións redox (con encimas oxidorredutases) e pode ceder electróns á cadea de transporte electrónico, polo que é importante na produción de enerxía na célula. É un dinucleótido, xa que está formado por dous nucleótidos unidos polos seus grupos fosfato, un deles leva a base nitroxenada adenina e o outro a nicotinamida, que deriva da vitamina B3.
No metabolismo o NAD+/NADH está implicado en reaccións de oxidación-redución, nas que capta/cede electróns e hidróxenos, e pode servir para levar electróns dunha reacción química a outra. Pode atoparse en dúas formas: en forma oxidada ou NAD+, que funciona nas reaccións como oxidante doutras moléculas á vez que se reduce a NADH, ou en forma reducida ou NADH, que funciona como redutor doutras moléculas á vez que se oxida a NAD+. As reaccións de transferencia de electróns son a principal función do NAD+, que tamén se emprega noutros procesos celulares, o máis importante dos cales é ser substrato de encimas que adicionan ou eliminan grupos químicos das proteínas nas modificacións postraducionais. Debido á importancia destas funcións, os encimas implicados no metabolismo do NAD+ son obxectivos para o descubrimento de fármacos.
Nos organismos o NAD+ pode sintetizarse a partir de biomoléculas sinxelas como os aminoácidos triptófano ou ácido aspártico ou poden obterse os seus compoñentes ou precursores dos alimentos, como a vitamina niacina ou B3. Tamén se poden obter compoñentes nas reaccións de descomposición do NAD+. Estes compoñentes preformados pasan entón por unha vía de reciclaxe que os converte de novo na forma activa. Parte do NAD+ convértese tamén en nicotinamida adenina dinucleótido fosfato (NADP+), que é outro importante coencima, que actúa quimicamente igual, pero intervén noutras vías metabólicas.
Propiedades físicas e químicas
[editar | editar a fonte]O dinucleótido de nicotinamida e adenina, igual que todos os dinucleótidos, está formado por dous nucleótidos unidos por un par de grupos fosfato que actúan como ponte. Ditos nucleótidos constan de dúas ribosas, unha unida á base nitroxenada adenina por enlace N-glicosídico, e a outra unida á nicotinamida de xeito similar. A porción de nicotinamida pode enlazarse con dúas orientacións distintas ao carbono anomérico do azucre. Debido a estas dúas posibles estruturas, o composto pode existir como dous diastereoisómeros, dos cales o diastereoisómero β-nicotinamida de NAD+ é a forma que se atopa na célula.[2]
No metabolismo o composto acepta ou cede electróns en reaccións redox.[3] Estas reaccións (resumidas na fórmula de abaixo) implican a extracción de dous átomos de hidróxeno do reactivo (RH2), en forma dun ión hidruro (:H-), e un protón (H+). O protón libérase no medio de reacción, mentres que o reactivo se oxida (e queda na forma R) e o NAD+ redúcese a NADH debido á transferencia do hidruro ao anel de nicotinamida. Pasaron á nicotinamida un átomo de hidróxeno (que pasa ao carbono 4) e un electrón, que neutraliza a carga + que tiña o nitróxeno do anel da molécula (é dicir, pasou un ión hidruro), e, como consecuencia, hai unha redistribución dos dobres enlaces no anel.
- RH2 + NAD+ → NADH + H+ + R
O potencial de punto medio do par redox NAD+/NADH é -0,32 voltios, o cal fai do NADH un forte axente redutor.[4] A reacción é facilmente reversible cando o NADH reduce outra molécula e queda oxidado a NAD+. Isto significa que este coencima está cambiando ciclicamente entre as súas formas NAD+ e NADH sen ser consumida.[2]

Todas as formas deste coencima en estado sólido teñen aspecto de pos brancos amorfos que son higroscópicos e moi solubles en auga.[5] A forma sólida é estable se se conserva seca e na escuridade. As solucións de NAD+ son incoloras e estables durante aproximadamente unha semana a 4 °C e un pH neutro (igual a 7), pero descompóñense rapidamente en ácidos ou álcalis. Tras a súa descomposición forman produtos que son inhibidores encimáticos.[6]
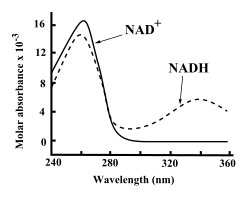
Tanto o NAD+ como o NADH absorben fortemente as radiacións electromagnéticas na banda ultravioleta debido á presenza da base nitroxenada adenina. Por exemplo, o pico de absorción do NAD+ sitúase nunha lonxitude de onda de 259 nanómetros (nm), cun coeficiente molar de absorción de 16.900 M−1cm−1. Mentres que o NADH presenta ademais outro pico de absorción ultravioleta nos 339 nm cun coeficiente molar de absorción de 6.220 M−1cm.[7] Esta diferenza na absorción de espectros ultravioleta entre a forma oxidada e a reducida do coencima nas lonxitudes de onda máis altas fai que sexa sinxelo medir a conversión dunha forma á outra nos ensaios encimáticos, medindo a cantidade de absorción de raios UV de 340 nm nun espectrómetro.[7]
O NAD+ e o NADH tamén difiren na súa fluorescencia, xa que o NADH ten en solución un pico de emisión a 460 nm e un tempo de vida de fluorescencia de 0,4 nanosegundos (ns), entanto que a forma oxidada (NAD+) non ten fluorescencia.[8] As propiedades do sinal de fluorescencia cambian cando o NADH se une ás proteínas, polo que estes cambios poden ser utilizados para medir as constantes de disociación, as cales son útiles no estudo da cinética encimática.[8][9] Estes cambios son utilizados tamén para medir os cambios no estado redox de células vivas, a través do microscopio de fluorescencia.[10]
Concentración e estado nas células
[editar | editar a fonte]Nun fígado de rata a cantidade total de NAD+ e NADH é aproximadamente de 1 μmol por gramo de peso fresco, unhas 10 veces a concentración de NADP+ e NADPH nas mesmas células.[11] A concentración real de NAD+ no citosol da célula é máis difícil de medir, con estimacións recentes en células animais que están entre 0,3 mM,[12][13] e aproximadamente de 1,0 a 2,0 mM en lévedos.[14] Porén, máis do 80% está unido a proteínas, así que a concentración en solución é moito máis baixa.[15]

Os datos sobre outros compartimentos celulares son limitados, aínda que na mitocondria a concentración de NAD+ é similar á do citosol.[13] Este NAD+ transpórtase ao interior da mitocondria por unha proteína específica dentro da membrana, xa que o coencima non pode pasar a través das membranas por difusión.[16]
O balance entre a forma oxidada e reducida do dinucleótido de nicotinamida e adenina chámase proporción NAD+/NADH. Esta proporción é un compoñente do que se chama estado redox dunha célula, unha medición que reflicte tanto as actividades metabólicas como a saúde das células.[17] Os efectos da proporción NAD+/NADH son complexos, pois controlan a actividade de varios encimas chaves, incluíndo a gliceraldehido-3-fosfato deshidroxenase e a piruvato deshidroxenase. Nos tecidos sans de mamíferos a estimación da proporción entre a NAD+ libre e a NADH no citoplasma encóntrase tipicamente arredor de 700; polo cal a proporción é favorable para reaccións de oxidación.[18][19] A proporción de NAD+/NADH total é moito máis baixa, con estimacións entre 0,05 e 4.[20] Polo contrario, a proporción de NADP+/NADPH está normalmente arredor de 0,005, así que o NADPH é a forma dominante deste coencima.[21] Estas distintas proporcións son a chave para entendermos os diferentes papeis metabólicos que teñen o NADH e o NADPH.
Biosíntese
[editar | editar a fonte]O NAD+ sintetízase a través de dúas vías metabólicas: unha ruta de novo a partir de aminoácidos, ou en vías de recuperación por medio da reciclaxe de compoñentes preformados como a nicotinamida utilizada de novo para sintetizar NAD+.
Produción de novo
[editar | editar a fonte]
A maioría dos organismos sintetizan NAD+ a partir de compoñentes simples.[3] O sitio específico da reacción é diferente entre os organismos, pero unha característica común é a xeración de ácido quinolínico (QA) a partir dun aminoácido; xa sexa o triptófano (Trp) en animais e algunhas bacterias, ou o ácido aspártico nalgunhas bacterias e plantas.[22][23] O ácido quinolínico convértese en mononucleótido de ácido nicotínico (NaMN) por medio da transferencia do grupo fosforribosa. Un grupo adenina transfírese entón para formar dinucleótido de ácido nicotínico e adenina (NaAD). Finalmente, o grupo de ácido nicotínico do NaAD sofre amidación e convértese nun grupo nicotinamida (Nam), formando así o dinucleótido de nicotinamida e adenina (NAD+).[3]
Unha transformación posterior pode ser a conversión dalgúns NAD+ en NADP+ por acción do encima NAD+ quinase, que cataliza a fosforilación do NAD+.[24] Na maioría dos organismos, este encima utiliza ATP como fonte do grupo fosfato, aínda que nalgunhas bacterias, como a Mycobacterium tuberculosis e algunhas archaea hipertermófilas como a Pyrococcus horikoshii do xénero Pyrococcus, usan polifosfatos inorgánicos como doantes alternativos de fosforilo (grupos fosfato).[25][26]

Vías de recuperación
[editar | editar a fonte]Ademais de ensamblar o NAD+ de novo a partir de aminoácidos precursores simples, as células tamén recuperan compostos preformados que conteñen nicotinamida. Aínda que se coñecen outros precursores, os tres compostos naturais que conteñen o anel de nicotinamida e son usados nestas vías metabólicas de recuperación ou "rescate" son o ácido nicotínico (Na), a nicotinamida (Nam) e a nicotinamida ribósido (NR).[27] Os precursores introdúcense na vía biosintética do NAD(P)+ (mostrada abaixo) a través de reaccións de adenilación e fosforribosilación.[3] Estes compostos poden tomarse na dieta, na cal a mestura de ácido nicotínico e nicotinamida se coñece como vitamina B3 ou niacina. Porén, estes compostos tamén son producidos no interior das células, cando o grupo nicotinamida se libera do NAD+ nas reaccións de transferencia de ADP-ribosa. De feito, os encimas implicados en ditas vías de recuperación parecen estar concentradas no núcleo celular, o que pode compensar o alto nivel de reaccións que consomen NAD+ neste orgánulo.[28] As células poden tamén tomar NAD+ extracelular do seu medio.[29]
A pesar da presenza da vía de novo, as reaccións de recuperación son esenciais nos seres humanos; unha carencia de niacina na dieta provoca a enfermidade de déficit vitamínico coñecida como pelagra.[30] Esta elevada esixencia de NAD+ débese ao constante consumo do coencima en reaccións como modificacións postraducionais, xa que a transformación cíclica do NAD+ entre as forma oxidada e reducida nas reaccións redox non cambia en nada os niveis xerais do coencima.[3]
As vías de recuperación utilizadas en microorganismos difiren das usadas en mamíferos.[31] Por exemplo, algúns axentes patóxenos, como o lévedo Candida glabrata e a bacteria Haemophilus influenzae son auxótrofos de NAD+, é dicir, non poden sintetizar NAD+, pero posúen vías de recuperación e, por tanto, son dependentes de fontes externas de NAD+ ou dos seus precursores.[32][33]
Aínda máis sorprendente é o patóxeno intracelular Chlamydia trachomatis, que no seu xenoma carece de candidatos reconocibles para calquera dos xenes implicados na biosíntese ou a recuperación tanto do NAD+ coma do NADP+, polo cal debe adquirir estes coencimas a partir do seu hospedante.[34]
Funcións
[editar | editar a fonte]
O NAD+/NADH ten varias funcións esenciais no metabolismo. Actúa como coencima nas reaccións redox, como doante de grupos ADP-ribosa nas reaccións de ADP-ribosilación, como precursor do segundo mensaxeiro da molécula cíclica ADP-ribosa, e tamén actúa como substrato para as ADN ligases bacterianas e un grupo de encimas chamados sirtuínas, que utilizan NAD+ para eliminar os grupos acetilo das proteínas acetiladas.
Oxidorredutases
[editar | editar a fonte]O papel principal do NAD+ no metabolismo é a transferencia de electróns dunha molécula a outra. As reaccións deste tipo son catalizadas por un gran grupo de encimas que se denominan oxidorredutases. Os nomes correctos para estes encimas conteñen os nomes dos seus substratos: por exemplo, a NADH-ubiquinona oxidorredutase (ou NADH deshidroxenase) cataliza a oxidación do NADH polo coencima Q.[36] Porén, estes encimas son tamén coñecidos como deshidroxenases ou redutases, co que a NADH-ubiquinona oxidorredutase chámase comunmente NADH deshidroxenase ou coencima Q redutase.[37]
Cando o NAD+ e o NADH están unidos a unha proteína mantéñense habitualmente dentro dun motivo estrutural da proteína coñecido como pregamento de Rossmann.[38] Este motivo recibe o seu nome do científico Michael Rossmann, que foi o primeiro en observar o común que é esta estrutura nas proteínas que se unen a nucleótidos.[39] Este pregamento contén tres ou máis láminas beta paralelas unidas por dúas hélices alfa na orde beta-alfa-beta-alfa-beta. Isto forma unha lámina beta flanqueada por unha capa de hélices alfa a cada lado. Debido a que cada pregamento de Rossmann se une a un nucleótido, os dominios de unión para o dinucleótido NAD+ están formados por dous pregamentos de Rossmann, nos que cada un dos pregamentos se une a un dos nucleótidos do cofactor.[39] Porén, este pregamento non é universal entre os encimas dependentes de NAD, xa que se descubriu recentemente que unha clase de encimas bacterianos implicados no metabolismo dos aminoácidos se une ao coencima, pero carece deste motivo.[40]

Cando se une ao sitio activo dunha oxidorredutase, o anel de nicotinamida do coencima colócase de modo que poida aceptar un hidruro do outro substrato. Debido a que o carbono C4 que acepta o hidróxeno é proquiral, isto pode ser aproveitado na cinética encimática para dar información sobre o mecanismo do encima. Isto faise por medio da mestura dun encima cun substrato que ten átomos de deuterio que substitúen os seus hidróxenos, polo que o encima reducirá o NAD+ transferindo deuterio en lugar de hidróxeno. Neste caso, un encima pode producir un dos dous estereoisómeros de NADH. Nalgúns encimas, o hidróxeno transfírese desde a parte superior do plano superior do anel de nicotinamida; estes denomínanse oxidorredutases de clase A, mentres que as de clase B transfiren o átomo desde abaixo.[41]
Malia esta similitude na forma en que as proteínas se unen aos dous coencimas, os encimas case sempre mostran un alto nivel de especificidade, xa sexa polo NAD+ ou o NADP+.[42] Esta especificidade reflicte as distintas funcións metabólicas dos respectivos coencimas, e é o resultado da presenza de diferentes series de residuos de aminoácidos nos dous tipos de sitio de unión para o coencima. Por exemplo, no sitio activo dos encimas dependentes de ADP, fórmase un enlace iónico entre a cadea lateral dun aminoácido básico e o grupo fosfato ácido do NADP+. Polo contrario, en encimas dependentes de NAD+, a carga neste oco invértese, evitando que se una o NADP+. Porén, hai unhas poucas excepcións a esta regra xeral, e encimas como a aldosa redutase, Glicosa-6-fosfato deshidroxenase (G6FD), e a metilentetrahidrofolato redutase poden utilizar ambos os coencimas nalgunhas especies.[43]
Funcións no metabolismo redox
[editar | editar a fonte]
As reaccións redox catalizadas por oxidorredutases son vitais en todo o metabolismo, pero dentro do proceso destas reaccións é especialmente importante a liberación de enerxía a partir dos nutrientes, onde os compostos reducidos, como a glicosa, se oxidan, liberando deste modo enerxía, que se transfire ao NAD+, reducíndoo a NADH, como parte da glicólise e o ciclo de Krebs. En células eucariotas, os electróns transportados polo NADH que se produce no citosol por glicólise, son transferidos ao interior da mitocondria (para a súa reoxidación a NAD+) pola lanzadeira do malato-aspartato ou a lanzadeira do glicerol-fosfato.[44] O NADH mitocondrial é entón oxidado á súa vez pola cadea de transporte de electróns, que bombea protóns ao longo da membrana e xera ATP por medio da fosforilación oxidativa.[45] Estes sistemas de lanzadeira tamén teñen a mesma función de transporte nos cloroplastos.[46]
Dado que as formas oxidada e reducida do dinucleótido de nicotinamida e adenina se usan nestes conxuntos enlazados de reaccións, a célula mantén concentracións significativas tanto de NAD+ coma de NADH, cunha alta proporción NAD+/NADH que permite a este coencima actuar como axente oxidante e axente redutor. Polo contrario, a función principal do NADP+ é a de ser axente redutor no anabolismo en vías como a biosíntese de ácidos graxos e a fotosíntese. Como o NADPH é necesario para impulsar as reaccións redox como un forte axente redutor, a proporción NADP+/NADPH mantense moi baixa.[47]
Aínda que é importante no catabolismo, o NADH utilízase tamén en reaccións anabólicas como a gliconeoxénese.[48] Esta necesidade de NADH no anabolismo presenta un problema para os procariotas que crecen utilizando nutrientes que liberan só pequenas cantidades de enerxía. Por exemplo, as bacterias nitrificantes como Nitrobacter oxidan o nitrito a nitrato, o que libera enerxía suficiente para bombear os protóns e xerar ATP, pero non para producir NADH directamente.[49] Como o NADH segue sendo necesario para as reaccións anabólicas, estas bacterias usan unha nitrito oxidorredutase para producir suficiente forza protón-motriz e dirixir, en parte, a circulación de electróns na cadea de transporte de electróns en sentido inverso, xerando NADH.[50]
Funcións non redox
[editar | editar a fonte]O NAD+ consómese tamén nas reaccións de transferencia de ADP-ribosa. Por exemplo, os encimas chamados ADP-ribosiltransferases engaden o grupo ADP-ribosa desta molécula ás proteínas, nunha modificación postraducional chamada ADP-ribosilación.[51] O NAD+ pode ser tamén engadido ao ARN como unha modificación de base.[52] A ADP-ribosilación implica quer a adición dun só grupo ADP-ribosa (mono-ADP-ribosilación) quer a transferencia de ADP-ribosa ás proteínas en cadeas longas ramificadas (poli(ADP-ribosil)ación)[53]. A mono-ADP-ribosilación identificouse por primeira vez como o mecanismo utilizado por un grupo de toxinas bacterianas, particularmente a toxina colérica, pero tamén está implicada na comunicación celular normal.[54][55] A poli(ADP-ribosil)ación lévana a cabo as poli(ADP-ribosa) polimerases.[53][56] A estrutura da poli-(ADP-ribosa) está implicada na regulación de varios procesos celulares, e é a máis importante no núcleo celular, actuando en procesos como a reparación do ADN e o mantemento dos telómeros.[56] Ademais destas funcións dentro da célula, descubriuse recentemente un grupo de ADP-ribosiltransferases extracelulares, pero as súas funcións non están claras.[57]

Este coencima actúa tamén na comunicación celular como precursor da ADP-ribosa cíclica; que se produce a partir de NAD+ pola acción de ADP-ribosil ciclases, funcionando o coencima como un segundo mensaxeiro.[58] Esta molécula actúa na sinalización de calcio liberando calcio das reservas intracelulares.[59] Isto realízao uníndose e abrindo unha clase de canles de calcio chamadas receptores de rianodina, que se encontran localizadas nas membranas de orgánulos como o retículo endoplasmático.[60]
O NAD+ tamén é consumido polas sirtuínas, que son deacetilases dependentes de NAD+, como a Sir2.[61] Estes encimas actúan transferindo un grupo acetilo das súas proteínas substrato ao grupo ADP-ribosa do NAD+; isto rompe o coencima e libera nicotinamida e O-acetil-ADP-ribosa. As sirtuínas parecen estar implicadas principalmente na regulación da transcrición xenética por medio da desacetilación de histonas e a alteración da estrutura do nucleosoma.[62] Porén, as sirtuínas poden desacetilar tamén proteínas non histonas. Estas actividades das sirtuínas son particularmente interesantes debido á súa importancia na regulación do [[envellecemento.[63]
Outros encimas dependentes do NAD+ inclúen as ADN ligases bacterianas, que unen dous extremos do ADN usando NAD+ como un substrato para doar un grupo de adenosín monofosfato (AMP) ao fosfato 5' dun extremo do ADN. Este intermediario é entón atacado polo grupo hidroxilo 3' do outro extremo de ADN, formando un novo enlace fosfodiéster.[64] Isto contrasta coas ADN ligases eucariotas, que utilizan ATP para formar o intermediario ADN-AMP.[65]
Usos farmacolóxicos
[editar | editar a fonte]Os encimas que xeran e utilizan NAD+ e NADH son importantes tanto para a farmacoloxía actual coma na investigación de tratamentos de diversas doenzas.[66] O deseño e desenvolvemento de fármacos utiliza o NAD+ de tres formas: (1) como branco directo de fármacos, (2) por medio do deseño de inhibidores encimáticos ou outros activadores baseados na súa estrutura que cambia a actividade de encimas dependentes do NAD, e (3) tamén tratando de inhibir a biosíntese de NAD+.[67]
O NAD+ non se usa actualmente como tratamento de ningunha enfermidade. Non obstante, é potencialmente útil como axente terapéutico nas enfermidades neurodexenerativas como a enfermidade de Alzheimer e a enfermidade de Parkinson.[3] As probas que existen sobre os beneficios do uso de NAD + para evitar a dexeneración neuronal son ambivalentes: en ratos son prometedoras,[68] mentres que un ensaio clínico sobre a enfermidade de Parkinson no que se incluían grupos de control aos que se lles administraba placebo non puido demostrar efectos apreciables.[69] O NAD+ tamén é un branco directo do fármaco isoniazida, o cal se usa no tratamento da tuberculose, unha infección producida por Mycobacterium tuberculosis. A isoniazida é un profármaco e unha vez que entra na bacteria, é activada por unha peroxidase, a cal oxida o composto á súa forma de radical libre.[70] Este radical despois reacciona coa NADH, para producir adutos que son inhibidores moi potentes dos encimas enoíl-ACP redutases,[71] e dihidrofolato redutase.[72]
Como un gran número de oxidorredutases utilizan NAD+ e NADH como substratos, e se unen a eles utilizando un motivo estrutural altamente conservado, a idea de que inhibidores baseados en NAD+ poderían ser específicos dun encima é sorprendente.[73] Non obstante, isto podería ser posible: por exemplo, os inhibidores baseados nos compostos ácido micofenólico e tiazofurina inhiben a IMP deshidroxenase no sitio de unión do NAD+. Debido á importancia deste encima no metabolismo das purinas, estes compostos poderían ser útiles como fármacos anticanceríxenos, antivirais ou inmunosupresores.[73][74] Outros fármacos non son encimas inhibidores, senón que activan encimas implicados no metabolismo do NAD+. As sirtuínas son un branco particularmente interesante para estes fármacos, xa que a activación destas desacetilases dependentes de NAD aumenta a lonxevidade.[75] Os compostos como o resveratrol aumentan a actividade destes encimas, que poden ser importantes dada a súa capacidade de atrasar o envellecemento tanto en organismos modelo de vertebrados coma de invertebrados.[76][77][78]
Debido ás diferenzas nas vías metabólicas da biosíntese de NAD+ entre organismos tan distintos como bacterias e humanos, esta área do metabolismo é prometedora para o desenvolvemento de novos antibióticos.[79][80] Por exemplo, o encima nicotinamidase, que converte a nicotinamida en ácido nicotínico, é un branco para o deseño de fármacos, xa que este encima está ausente en humanos, pero presente en fungos unicelulares e bacterias.[31]
Historia
[editar | editar a fonte]
O coencima NAD+ foi descuberto polos bioquímicos británicos Arthur Harden e William Youndin en 1906.[81] Notificaron que ao engadiren extracto de lévedo fervido e filtrado a extractos de lévedo sen ferver, acelerábase moito a fermentación alcohólica. Chamaron cofermento ao factor non identificado responsable deste efecto. A través dunha longa e dificultosa purificación a partir de extractos de lévedo, o sueco Hans von Euler-Chelpin identificou este factor termoestable como un nucleótido de azucre-fosfato.[82] En 1936, o científico alemán Otto Heinrich Warburg demostrou a función deste coencima nucleotídico na transferencia de hidruro (:H-) e identificou a porción de nicotinamida como o sitio das reaccións redox.[83]
En 1938, Conrad Elvehjem identificou unha fonte de nicotinamida ao purificar niacina procedente do fígado e demostrou que esta vitamina contiña ácido nicotínico e nicotinamida,[84] e logo, en 1939, proporcionou a primeira evidencia sólida de que a niacina se usaba para sintetizar NAD+.[85] A principios da década de 1940, Arthur Kornberg realizou outra importante contribución para a comprensión do metabolismo do NAD+, ao ser o primeiro en detectar un encima na súa vía biosintética.[86] Posteriormente, en 1949, os bioquímicos estadounidenses Morris Friedkin e Albert L. Lehninger descubriron que o NADH estaba vencellado a vías metabólicas como o ciclo do ácido cítrico e a síntese de ATP na fosforilación oxidativa.[87]
Finalmente, en 1959, Jack Preiss e Philip Handler descubriron os intermediarios e encimas que participan na biosíntese do NAD+;[88][89] debido ao que a síntese de novo é frecuentemente chamada «vía de Preiss-Handler» na súa honra.
As funcións non redox do NAD+ e o NADP+ son un descubrimento recente.[2] A primeira destas funcións que foi identificada foi o uso do NAD+ como doante de ADP-ribosa nas reaccións de ADP-ribosilación observadas a comezos da década de 1960.[90] Estudos posteriores nos anos 80 e 90, revelaron as actividades dos metabolitos de NAD+ e NADP+ na comunicación celular; tales como a acción de ADP-ribosa cíclica, que foi descuberta en 1987.[91] O metabolismo do NAD+ segue a ser hoxe en día unha área de intensa investigación, cun maior interese despois de que no ano 2000 Shinichiro Imai e uns colaboradores, descubrisen no Instituto de Tecnoloxía de Massachusetts (MIT) as proteínas chamadas sirtuínas; que son encimas deacetilases dependentes do NAD+.[92]
Notas
[editar | editar a fonte]- ↑ Son múltiples as variantes do nome que se teñen usado para nomear este composto en libros científicos e textos científicos de Internet: dinucleótido de nicotinamida e adenina, nicotinamida-adenina dinucleótido, nicotinamida adenín dinucleótido, nicotín adenín dinucleótido, nicotín-adenín dinucleótido (e outras variantes con -) etc. Por exemplo, en castelán o libro de Bioquímica de Lehninger de 1980 utiliza "nicotinamida-adenín-dinucleótido" (todo xunto), pero o Principios de Bioquímica de Lehninger de 1988 xa usa "dinucleótido de nicotinamida y de adenina". Pero, con diferenza, o que máis se usa son as abreviaturas.
- ↑ 2,0 2,1 2,2 Pollak, N; Dölle C, Ziegler M (2007). "The power to reduce: pyridine nucleotides—small molecules with a multitude of functions". Biochem. J. 402 (2): 205–18. PMC 1798440. PMID 17295611. doi:10.1042/BJ20061638.
- ↑ 3,0 3,1 3,2 3,3 3,4 3,5 Belenky P; Bogan KL, Brenner C (2007). "NAD+ metabolism in health and disease" (PDF). Trends Biochem. Sci. 32 (1): 12–9. PMID 17161604. doi:10.1016/j.tibs.2006.11.006. Arquivado dende o orixinal (PDF) o 04 de xullo de 2009. Consultado o 23 de decembro de 2007.
- ↑ Unden G; Bongaerts J (1997). "Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors". Biochim. Biophys. Acta 1320 (3): 217–34. PMID 9230919. doi:10.1016/S0005-2728(97)00034-0.
- ↑ Windholz, Martha (1983). The Merck Index: an encyclopedia of chemicals, drugs, and biologicals (10th ed.). Rahway NJ, US: Merck. p. 909. ISBN 0911910271.
- ↑ Biellmann JF, Lapinte C, Haid E, Weimann G (1979). "Structure of lactate dehydrogenase inhibitor generated from coenzyme". Biochemistry 18 (7): 1212–7. PMID 218616. doi:10.1021/bi00574a015.
- ↑ 7,0 7,1 Dawson, R. Ben (1985). Data for biochemical research (3rd ed.). Oxford: Clarendon Press. p. 122. ISBN 0-19-855358-7.
- ↑ 8,0 8,1 Lakowicz JR, Szmacinski H, Nowaczyk K, Johnson ML (1992). "Fluorescence lifetime imaging of free and protein-bound NADH". Proc. Natl. Acad. Sci. U.S.A. 89 (4): 1271–5. PMC 48431. PMID 1741380. doi:10.1073/pnas.89.4.1271.
- ↑ Jameson DM, Thomas V, Zhou DM (1989). "Time-resolved fluorescence studies on NADH bound to mitochondrial malate dehydrogenase". Biochim. Biophys. Acta 994 (2): 187–90. PMID 2910350.
- ↑ Kasimova MR, Grigiene J, Krab K; et al. (2006). "The free NADH concentration is kept constant in plant mitochondria under different metabolic conditions". Plant Cell 18 (3): 688–98. PMC 1383643. PMID 16461578. doi:10.1105/tpc.105.039354.
- ↑ Reiss PD, Zuurendonk PF, Veech RL (1984). "Measurement of tissue purine, pyrimidine, and other nucleotides by radial compression high-performance liquid chromatography". Anal. Biochem. 140 (1): 162–71. PMID 6486402. doi:10.1016/0003-2697(84)90148-9.
- ↑ Yamada K, Hara N, Shibata T, Osago H, Tsuchiya M (2006). "The simultaneous measurement of nicotinamide adenine dinucleotide and related compounds by liquid chromatography/electrospray ionization tandem mass spectrometry". Anal. Biochem. 352 (2): 282–5. PMID 16574057. doi:10.1016/j.ab.2006.02.017.
- ↑ 13,0 13,1 Yang H, Yang T, Baur JA, Perez E, Matsui T, Carmona JJ, Lamming DW, Souza-Pinto NC, Bohr VA, Rosenzweig A, de Cabo R, Sauve AA, Sinclair DA. (2007). "Nutrient-Sensitive Mitochondrial NAD+ Levels Dictate Cell Survival". Cell 130 (6): 1095–107. PMID 17889652. doi:10.1016/j.cell.2007.07.035.
- ↑ Belenky P, Racette FG, Bogan KL, McClure JM, Smith JS, Brenner C (2007). "Nicotinamide riboside promotes Sir2 silencing and extends lifespan via Nrk and Urh1/Pnp1/Meu1 pathways to NAD+". Cell 129 (3): 473–84. PMID 17482543. doi:10.1016/j.cell.2007.03.024.
- ↑ Blinova K, Carroll S, Bose S; et al. (2005). "Distribution of mitochondrial NADH fluorescence lifetimes: steady-state kinetics of matrix NADH interactions". Biochemistry 44 (7): 2585–94. PMID 15709771. doi:10.1021/bi0485124.
- ↑ Todisco S, Agrimi G, Castegna A, Palmieri F (2006). "Identification of the mitochondrial NAD+ transporter in Saccharomyces cerevisiae". J. Biol. Chem. 281 (3): 1524–31. PMID 16291748. doi:10.1074/jbc.M510425200. Arquivado dende o orixinal o 13 de abril de 2020. Consultado o 15 de agosto de 2011.
- ↑ Schafer F, Buettner G (2001). "Redox environment of the cell as viewed through the redox state of the glutathione disulfide/glutathione couple". Free Radic Biol Med 30 (11): 1191–212. PMID 11368918. doi:10.1016/S0891-5849(01)00480-4.
- ↑ Williamson DH, Lund P, Krebs HA (1967). "The redox state of free nicotinamide-adenine dinucleotide in the cytoplasm and mitochondria of rat liver". Biochem. J. 103 (2): 514–27. PMC 1270436. PMID 4291787.
- ↑ Zhang Q, Piston DW, Goodman RH (2002). "Regulation of corepressor function by nuclear NADH". Science 295 (5561): 1895–7. PMID 11847309. doi:10.1126/science.1069300.
- ↑ Lin SJ, Guarente L (2003). "Nicotinamide adenine dinucleotide, a metabolic regulator of transcription, longevity and disease". Curr. Opin. Cell Biol. 15 (2): 241–6. PMID 12648681. doi:10.1016/S0955-0674(03)00006-1.
- ↑ Veech RL, Eggleston LV, Krebs HA (1969). "The redox state of free nicotinamide-adenine dinucleotide phosphate in the cytoplasm of rat liver". Biochem. J. 115 (4): 609–19. PMC 1185185. PMID 4391039.
- ↑ Katoh A, Uenohara K, Akita M, Hashimoto T (2006). "Early steps in the biosynthesis of NAD in Arabidopsis start with aspartate and occur in the plastid". Plant Physiol. 141 (3): 851–7. PMC 1489895. PMID 16698895. doi:10.1104/pp.106.081091.
- ↑ Foster JW, Moat AG (1 de marzo de 1980). "Nicotinamide adenine dinucleotide biosynthesis and pyridine nucleotide cycle metabolism in microbial systems". Microbiol. Rev. 44 (1): 83–105. PMC 373235. PMID 6997723.
- ↑ Magni G, Orsomando G, Raffaelli N (2006). "Structural and functional properties of NAD kinase, a key enzyme in NADP biosynthesis". Mini reviews in medicinal chemistry 6 (7): 739–46. PMID 16842123. doi:10.2174/138955706777698688.
- ↑ Sakuraba H, Kawakami R, Ohshima T (2005). "First archaeal inorganic polyphosphate/ATP-dependent NAD kinase, from hyperthermophilic archaeon Pyrococcus horikoshii: cloning, expression, and characterization". Appl. Environ. Microbiol. 71 (8): 4352–8. PMC 1183369. PMID 16085824. doi:10.1128/AEM.71.8.4352-4358.2005.
- ↑ Raffaelli N, Finaurini L, Mazzola F; et al. (2004). "Characterization of Mycobacterium tuberculosis NAD kinase: functional analysis of the full-length enzyme by site-directed mutagenesis". Biochemistry 43 (23): 7610–7. PMID 15182203. doi:10.1021/bi049650w.
- ↑ Tempel W, Rabeh WM, Bogan KL; et al. (2007). "Nicotinamide riboside kinase structures reveal new pathways to NAD+". PLoS Biol. 5 (10): e263. PMC 1994991. PMID 17914902. doi:10.1371/journal.pbio.0050263.
- ↑ Anderson RM, Bitterman KJ, Wood JG; et al. (2002). "Manipulation of a nuclear NAD+ salvage pathway delays aging without altering steady-state NAD+ levels". J. Biol. Chem. 277 (21): 18881–90. PMID 11884393. doi:10.1074/jbc.M111773200. Arquivado dende o orixinal o 13 de abril de 2020. Consultado o 15 de agosto de 2011.
- ↑ Billington RA, Travelli C, Ercolano E; et al. (2008). "Characterization of NAD Uptake in Mammalian Cells". J. Biol. Chem. 283 (10): 6367–74. PMID 18180302. doi:10.1074/jbc.M706204200. Arquivado dende o orixinal o 28 de maio de 2020. Consultado o 15 de agosto de 2011.
- ↑ Henderson LM (1983). "Niacin". Annu. Rev. Nutr. 3: 289–307. PMID 6357238. doi:10.1146/annurev.nu.03.070183.001445.
- ↑ 31,0 31,1 Rongvaux A, Andris F, Van Gool F, Leo O (2003). "Reconstructing eukaryotic NAD metabolism". Bioessays 25 (7): 683–90. PMID 12815723. doi:10.1002/bies.10297.
- ↑ Ma B, Pan SJ, Zupancic ML, Cormack BP (2007). "Assimilation of NAD(+) precursors in Candida glabrata". Mol. Microbiol. 66 (1): 14–25. PMID 17725566. doi:10.1111/j.1365-2958.2007.05886.x.
- ↑ Reidl J, Schlör S, Kraiss A, Schmidt-Brauns J, Kemmer G, Soleva E (2000). "NADP and NAD utilization in Haemophilus influenzae". Mol. Microbiol. 35 (6): 1573–81. PMID 10760156. doi:10.1046/j.1365-2958.2000.01829.x.
- ↑ Gerdes SY, Scholle MD, D'Souza M; et al. (2002). "From genetic footprinting to antimicrobial drug targets: examples in cofactor biosynthetic pathways". J. Bacteriol. 184 (16): 4555–72. PMC 135229. PMID 12142426. doi:10.1128/JB.184.16.4555-4572.2002.
- ↑ Senkovich O, Speed H, Grigorian A; et al. (2005). "Crystallization of three key glycolytic enzymes of the opportunistic pathogen Cryptosporidium parvum". Biochim. Biophys. Acta 1750 (2): 166–72. PMID 15953771. doi:10.1016/j.bbapap.2005.04.009.
- ↑ "Enzyme Nomenclature, Recommendations for enzyme names from the Nomenclature Committee of the International Union of Biochemistry and Molecular Biology". Arquivado dende o orixinal o 05 de decembro de 2007. Consultado o 6 de decembro de 2007.
- ↑ "NiceZyme View of ENZYME: EC 1.6.5.3". Expasy. Arquivado dende o orixinal o 19 de decembro de 2007. Consultado o 16 de decembro de 2007.
- ↑ Lesk AM (1995). "NAD-binding domains of dehydrogenases". Curr. Opin. Struct. Biol. 5 (6): 775–83. PMID 8749365. doi:10.1016/0959-440X(95)80010-7.
- ↑ 39,0 39,1 Rao S, Rossmann M (1973). "Comparison of super-secondary structures in proteins". J Mol Biol 76 (2): 241–56. PMID 4737475. doi:10.1016/0022-2836(73)90388-4.
- ↑ Goto M, Muramatsu H, Mihara H; et al. (2005). "Crystal structures of Delta1-piperideine-2-carboxylate/Delta1-pyrroline-2-carboxylate reductase belonging to a new family of NAD(P)H-dependent oxidoreductases: conformational change, substrate recognition, and stereochemistry of the reaction". J. Biol. Chem. 280 (49): 40875–84. PMID 16192274. doi:10.1074/jbc.M507399200. Arquivado dende o orixinal o 11 de xuño de 2008. Consultado o 15 de agosto de 2011.
- ↑ Bellamacina CR (1 de setembro de 1996). "The nicotinamide dinucleotide binding motif: a comparison of nucleotide binding proteins". FASEB J. 10 (11): 1257–69. PMID 8836039.
- ↑ Carugo O, Argos P (1997). "NADP-dependent enzymes. I: Conserved stereochemistry of cofactor binding". Proteins 28 (1): 10–28. PMID 9144787. doi:10.1002/(SICI)1097-0134(199705)28:1<10::AID-PROT2>3.0.CO;2-N.
- ↑ Vickers TJ, Orsomando G, de la Garza RD; et al. (2006). "Biochemical and genetic analysis of methylenetetrahydrofolate reductase in Leishmania metabolism and virulence". J. Biol. Chem. 281 (50): 38150–8. PMID 17032644. doi:10.1074/jbc.M608387200. Arquivado dende o orixinal o 13 de outubro de 2008. Consultado o 15 de agosto de 2011.
- ↑ Bakker BM, Overkamp KM, van Maris AJ; et al. (2001). "Stoichiometry and compartmentation of NADH metabolism in Saccharomyces cerevisiae". FEMS Microbiol. Rev. 25 (1): 15–37. PMID 11152939. doi:10.1111/j.1574-6976.2001.tb00570.x.
- ↑ Rich PR (2003). "The molecular machinery of Keilin's respiratory chain". Biochem. Soc. Trans. 31 (Pt 6): 1095–105. PMID 14641005. doi:10.1042/BST0311095.
- ↑ Heineke D, Riens B, Grosse H; et al. (1991). "Redox Transfer across the Inner Chloroplast Envelope Membrane". Plant Physiol 95 (4): 1131–1137. PMC 1077662. PMID 16668101. doi:10.1104/pp.95.4.1131.
- ↑ Nicholls DG; Ferguson SJ (2002). Bioenergetics 3 (1st ed.). Academic Press. ISBN 0-125-18121-3.
- ↑ Sistare FD, Haynes RC (15 de outubro de 1985). "The interaction between the cytosolic pyridine nucleotide redox potential and gluconeogenesis from lactate/pyruvate in isolated rat hepatocytes. Implications for investigations of hormone action". J. Biol. Chem. 260 (23): 12748–53. PMID 4044607. Arquivado dende o orixinal o 12 de abril de 2008. Consultado o 15 de agosto de 2011.
- ↑ Freitag A, Bock E (1990). "Energy conservation in Nitrobacter". FEMS Microbiology Letters 66 (1–3): 157–62. doi:10.1111/j.1574-6968.1990.tb03989.x.
- ↑ Starkenburg SR, Chain PS, Sayavedra-Soto LA; et al. (2006). "Genome sequence of the chemolithoautotrophic nitrite-oxidizing bacterium Nitrobacter winogradskyi Nb-255". Appl. Environ. Microbiol. 72 (3): 2050–63. PMC 1393235. PMID 16517654. doi:10.1128/AEM.72.3.2050-2063.2006.
- ↑ Ziegler M (2000). "New functions of a long-known molecule. Emerging roles of NAD in cellular signaling". Eur. J. Biochem. 267 (6): 1550–64. PMID 10712584. doi:10.1046/j.1432-1327.2000.01187.x.
- ↑ Chen, Y Grace; Walter E Kowtoniuk, Isha Agarwal, Yinghua Shen, David R Liu (decembro de 2009). "LC/MS analysis of cellular RNA reveals NAD-linked RNA". Nat Chem Biol 5 (12): 879–881. PMC 2842606. PMID 19820715. doi:10.1038/nchembio.235.
- ↑ 53,0 53,1 Diefenbach J, Bürkle A (2005). "Introduction to poly(ADP-ribose) metabolism". Cell. Mol. Life Sci. 62 (7–8): 721–30. PMID 15868397. doi:10.1007/s00018-004-4503-3.
- ↑ Berger F, Ramírez-Hernández MH, Ziegler M (2004). "The new life of a centenarian: signaling functions of NAD(P)". Trends Biochem. Sci. 29 (3): 111–8. PMID 15003268. doi:10.1016/j.tibs.2004.01.007.
- ↑ Corda D, Di Girolamo M (2003). "Functional aspects of protein mono-ADP-ribosylation". EMBO J. 22 (9): 1953–8. PMC 156081. PMID 12727863. doi:10.1093/emboj/cdg209.
- ↑ 56,0 56,1 Burkle A (2005). "Poly(ADP-ribose). The most elaborate metabolite of NAD+". FEBS J. 272 (18): 4576–89. PMID 16156780. doi:10.1111/j.1742-4658.2005.04864.x.
- ↑ Seman M, Adriouch S, Haag F, Koch-Nolte F (2004). "Ecto-ADP-ribosyltransferases (ARTs): emerging actors in cell communication and signaling". Curr. Med. Chem. 11 (7): 857–72. PMID 15078170. doi:10.2174/0929867043455611.
- ↑ Guse AH (2004). "Biochemistry, biology, and pharmacology of cyclic adenosine diphosphoribose (cADPR)". Curr. Med. Chem. 11 (7): 847–55. PMID 15078169. doi:10.2174/0929867043455602.
- ↑ Guse AH (2004). "Regulation of calcium signaling by the second messenger cyclic adenosine diphosphoribose (cADPR)". Curr. Mol. Med. 4 (3): 239–48. PMID 15101682. doi:10.2174/1566524043360771.
- ↑ Guse AH (2005). "Second messenger function and the structure-activity relationship of cyclic adenosine diphosphoribose (cADPR)". FEBS J. 272 (18): 4590–7. PMID 16156781. doi:10.1111/j.1742-4658.2005.04863.x.
- ↑ North B, Verdin E (2004). "Sirtuins: Sir2-related NAD-dependent protein deacetylases". Genome Biol 5 (5): 224. PMC 416462. PMID 15128440. doi:10.1186/gb-2004-5-5-224.
- ↑ Blander G, Guarente L (2004). "The Sir2 family of protein deacetylases". Annu. Rev. Biochem. 73: 417–35. PMID 15189148. doi:10.1146/annurev.biochem.73.011303.073651.
- ↑ Trapp J, Jung M (2006). "The role of NAD+ dependent histone deacetylases (sirtuins) in ageing". Curr Drug Targets 7 (11): 1553–60. PMID 17100594.
- ↑ Wilkinson A, Day J, Bowater R (2001). "Bacterial DNA ligases". Mol. Microbiol. 40 (6): 1241–8. PMID 11442824. doi:10.1046/j.1365-2958.2001.02479.x.
- ↑ Schär P, Herrmann G, Daly G, Lindahl T (1997). "A newly identified DNA ligase of Saccharomyces cerevisiae involved in RAD52-independent repair of DNA double-strand breaks". Genes and Development 11 (15): 1912–24. PMC 316416. PMID 9271115. doi:10.1101/gad.11.15.1912.
- ↑ Sauve AA (2008). "NAD+ and vitamin B3: from metabolism to therapies". The Journal of pharmacology and experimental therapeutics 324 (3): 883–93. PMID 18165311. doi:10.1124/jpet.107.120758.
- ↑ Khan JA, Forouhar F, Tao X, Tong L (2007). "Nicotinamide adenine dinucleotide metabolism as an attractive target for drug discovery". Expert Opin. Ther. Targets 11 (5): 695–705. PMID 17465726. doi:10.1517/14728222.11.5.695.
- ↑ Kaneko S, Wang J, Kaneko M; et al. (2006). "Protecting axonal degeneration by increasing nicotinamide adenine dinucleotide levels in experimental autoimmune encephalomyelitis models". J. Neurosci. 26 (38): 9794–804. PMID 16988050. doi:10.1523/JNEUROSCI.2116-06.2006.
- ↑ Swerdlow RH (1998). "Is NADH effective in the treatment of Parkinson's disease?". Drugs Aging 13 (4): 263–8. PMID 9805207. doi:10.2165/00002512-199813040-00002.
- ↑ Timmins GS, Deretic V (2006). "Mechanisms of action of isoniazid". Mol. Microbiol. 62 (5): 1220–7. PMID 17074073. doi:10.1111/j.1365-2958.2006.05467.x. Arquivado dende o orixinal o 22 de decembro de 2007. Consultado o 15 de agosto de 2011.
- ↑ Rawat R, Whitty A, Tonge PJ (2003). "The isoniazid-NAD adduct is a slow, tight-binding inhibitor of InhA, the Mycobacterium tuberculosis enoyl reductase: adduct affinity and drug resistance". Proc. Natl. Acad. Sci. U.S.A. 100 (24): 13881–6. PMC 283515. PMID 14623976. doi:10.1073/pnas.2235848100.
- ↑ Argyrou A, Vetting MW, Aladegbami B, Blanchard JS (2006). "Mycobacterium tuberculosis dihydrofolate reductase is a target for isoniazid". Nat. Struct. Mol. Biol. 13 (5): 408–13. PMID 16648861. doi:10.1038/nsmb1089.
- ↑ 73,0 73,1 Pankiewicz KW, Patterson SE, Black PL; et al. (2004). "Cofactor mimics as selective inhibitors of NAD-dependent inosine monophosphate dehydrogenase (IMPDH)—the major therapeutic target". Curr. Med. Chem. 11 (7): 887–900. PMID 15083807. doi:10.2174/0929867043455648.
- ↑ Franchetti P, Grifantini M (1999). "Nucleoside and non-nucleoside IMP dehydrogenase inhibitors as antitumor and antiviral agents". Curr. Med. Chem. 6 (7): 599–614. PMID 10390603.
- ↑ Kim EJ, Um SJ (2008). "SIRT1: roles in aging and cancer". BMB Rep 41 (11): 751–6. PMID 19017485.
- ↑ Valenzano DR, Terzibasi E, Genade T, Cattaneo A, Domenici L, Cellerino A (2006). "Resveratrol prolongs lifespan and retards the onset of age-related markers in a short-lived vertebrate". Curr. Biol. 16 (3): 296–300. PMID 16461283. doi:10.1016/j.cub.2005.12.038.
- ↑ Howitz KT, Bitterman KJ, Cohen HY; et al. (2003). "Small molecule activators of sirtuins extend Saccharomyces cerevisiae lifespan". Nature 425 (6954): 191–6. PMID 12939617. doi:10.1038/nature01960.
- ↑ Wood JG, Rogina B, Lavu S; et al. (2004). "Sirtuin activators mimic caloric restriction and delay ageing in metazoans". Nature 430 (7000): 686–9. PMID 15254550. doi:10.1038/nature02789.
- ↑ Rizzi M, Schindelin H (2002). "Structural biology of enzymes involved in NAD and molybdenum cofactor biosynthesis". Curr. Opin. Struct. Biol. 12 (6): 709–20. PMID 12504674. doi:10.1016/S0959-440X(02)00385-8.
- ↑ Begley TP, Kinsland C, Mehl RA, Osterman A, Dorrestein P (2001). "The biosynthesis of nicotinamide adenine dinucleotides in bacteria". Vitam. Horm. 61: 103–19. PMID 11153263. doi:10.1016/S0083-6729(01)61003-3.
- ↑ Harden, A; Young, WJ (1906). "The Alcoholic Ferment of Yeast-Juice". Proceedings of the Royal Society of London (Series B, Containing Papers of a Biological Character ed.) 78 (526): 369–375.
- ↑ "Fermentation of sugars and fermentative enzymes" (PDF). Nobel Lecture, 23 de maio de 1930. Nobel Foundation. Arquivado dende o orixinal (PDF) o 27 de setembro de 2007. Consultado o 30 de setembro de 2007.
- ↑ Warburg O, Christian W. (1936). "Pyridin, the hydrogen-transferring component of the fermentation enzymes (pyridine nucleotide)". Biochemische Zeitschrift 287: 291.
- ↑ Elvehjem CA, Madden RJ, Strong FM, Woolley DW. (1938). "The isolation and identification of the anti-black tongue factor" (PDF). J. Biol. Chem. 123 (1): 137–49. Arquivado dende o orixinal (PDF) o 26 de marzo de 2009. Consultado o 15 de agosto de 2011.
- ↑ Axelrod AE, Madden RJ, Elvehjem CA (1939). "The effect of a nicotinic acid deficiency upon the coenzyme I content of animal tissues" (PDF). J. Biol. Chem. 131 (1): 85–93. Arquivado dende o orixinal (PDF) o 26 de marzo de 2009. Consultado o 15 de agosto de 2011.
- ↑ Kornberg, A. (1948). "The participation of inorganic pyrophosphate in the reversible enzymatic synthesis of diphosphopyridine nucleotide" (PDF). J. Biol. Chem. 176 (3): 1475–76. Arquivado dende o orixinal (PDF) o 26 de marzo de 2009. Consultado o 15 de agosto de 2011.
- ↑ Friedkin M, Lehninger AL. (1 de abril de 1949). "Esterification of inorganic phosphate coupled to electron transport between dihydrodiphosphopyridine nucleotide and oxygen". J. Biol. Chem. 178 (2): 611–23.
- ↑ Preiss J, Handler P. (1 de agosto de 1958). "Biosynthesis of diphosphopyridine nucleotide. I. Identification of intermediates". J. Biol. Chem. 233 (2): 488–92. PMID 13563526. Arquivado dende o orixinal o 14 de decembro de 2007. Consultado o 15 de agosto de 2011.
- ↑ Preiss J, Handler P. (1 de agosto de 1958). "Biosynthesis of diphosphopyridine nucleotide. II. Enzymatic aspects". J. Biol. Chem. 233 (2): 493–500. PMID 13563527. Arquivado dende o orixinal o 13 de abril de 2020. Consultado o 15 de agosto de 2011.
- ↑ Chambon P, Weill JD, Mandel P (1963). "Nicotinamide mononucleotide activation of new DNA-dependent polyadenylic acid synthesizing nuclear enzyme". Biochem. Biophys. Res. Commun. 11: 39–43. PMID 14019961. doi:10.1016/0006-291X(63)90024-X.
- ↑ Clapper DL, Walseth TF, Dargie PJ, Lee HC (15 de xullo de 1987). "Pyridine nucleotide metabolites stimulate calcium release from sea urchin egg microsomes desensitized to inositol trisphosphate". J. Biol. Chem. 262 (20): 9561–8. PMID 3496336. Arquivado dende o orixinal o 14 de decembro de 2007. Consultado o 15 de agosto de 2011.
- ↑ Imai S, Armstrong CM, Kaeberlein M, Guarente L (2000). "Transcriptional silencing and longevity protein Sir2 is an NAD-dependent histone deacetylase". Nature 403 (6771): 795–800. PMID 10693811. doi:10.1038/35001622.