Mutación por desprazamento do marco de lectura

Unha mutación por desprazamento do marco de lectura (ou por cambio da pauta de lectura) é unha mutación causada por indeis (insercións ou delecións) de certo número de nucleótidos na secuencia de ADN que non é divisible por tres. Debido a que a expresión xénica se realiza por medio de tripletes ou codóns, unha inserción ou deleción deste tipo pode cambiar o marco de lectura (o agrupamento de codóns), tendo como resultado unha tradución de proteínas completemente diferente da orixinal. Canto máis ao principio da secuencia ocorra a deleción ou inserción, máis se altera a proteína.[1] Unha mutación de desprazamento do marco de lectura non é o mesmo que un polimorfismo dun único nucleótido, no cal se substitúe un nucleótido, en lugar de inserirse ou eliminarse un. Unha mutación por desprazamento de marco de lectura causará en xeral que os codóns que están despois da mutación codifiquen diferentes aminoácidos. A mutación por desprazamento do marco de lectura tamén altera o primeiro codón de terminación ou stop ("UAA", "UGA" ou "UAG") que se encontra na secuencia, pero tamén podería orixinar un codón de terminación novo situado antes do normal. O polipéptido que se crea podería ser anormalmente curto ou anormalmente longo e moi probablemente non será funcional.[2]
As mutacións por desprazamento do marco de lectura aparecen en graves doenzas xenéticas como a enfermidade de Tay-Sachs; incrementan a susceptibilidade a certos cancros e clases de hipercolesterolemia familiar. En 1997,[3] unha mutación de desprazamento do marco delectura foi asociada á resistencia á infección polo retrovirus VIH. Propúxose que estas mutacións son unha fonte de novidade biolóxica, que explicaría, segundo algúns autores, a creación da nailonase; porén, esta interpretación é discutible. Un estudo feito por Negoro et al (2006) [4] atopou que era improbable que unha mutación deste tipo fose a causa, senón que posiblemente unha susbtitución de dous aminoácidos no sitio activo dunha esterase ancestral tivo como resultado a aparición da nailonase.
Introdución
[editar | editar a fonte]A información que contén o ADN determina a función dunha proteína nas células de todos os organismos. A transcrición e tradución fan que esta información se comunique para fabricar proteínas. Porén, un erro na lectura desta comunicación pode causar que o funcionamento da proteína non sexa correcto e finalmente cause unha doenza, incluso se a célula ten unha serie de medidas correctoras.
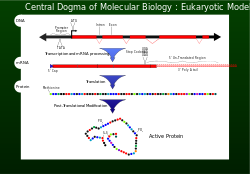
O dogma central
[editar | editar a fonte]- Artigo principal: Dogma central da bioloxía molecular.
En 1956 Francis Crick describiu o fluxo de información xenética desde o ADN a unha secuencia específica de aminoácidos para fabricar unha proteína denominándoo dogma central da bioloxía molecular.[1] Para que unha célula funcione correctamente, necesítase que as proteínas se produzan adecuadamente para desempeñaren as súas actividades estruturais e catalíticas. Para asegurar que o xenoma transmite satisfactoriamente a información, a célula incorpora mecanismos de corrección de probas como exonucleases e sistemas de reparación de discordancias durante a replicación do ADN .[1]
Transcrición e tradución
[editar | editar a fonte]- Artigos principais: Transcrición (xenética) e Tradución de proteínas.
Despois da replicación do ADN, a lectura dunha sección seleccionada de información xenética lévase a cabo por medio da transcrición.[1] Os nucleótidos que conteñen a información xenética están agora nunha febra molde mensaxeira chamada ARNm. O ARNm únese a unha subunidade dun ribosoma e interacciona cun ARNr. A información xenética transportada nos codóns do ARNm é agora lida (decodificada) polos anticodóns do ARNt. A medida que se le cada codón (triplete), os aminoácidos se enlazan formando a cadea polipeptídica ata que se chega ao codón de finalización (UAG, UGA ou UAA). Nese momento, o polipéptido (proteína) está sintetizada e é liberada.[1] De cada 1000 aminoácidos incorporados á proteína, normalmente só un é incorrecto. Esta fidelidade de recoñecemento dos codóns, indica a importancia de manter o marco de lectura correcto, o cal é asegurado por un axeitado apareamento de bases no sitio A do ribosoma, a actividade de hidrólise do GTP do factor EF-Tu, unha forma de estabilidade cinética e un mecanismo de corrección de probas unha vez que se libera EF-Tu.[1]
O desprazamento do marco de lectura pode tamén ocorrer durante a tradución de retrovirus na célula, producindo diferentes proteínas a partir de marcos de lectura abertos solapantes, como as proteínas retrovirais gag-pol-env. Isto é bastante común en virus e tamén ocorre en bacterias e lévedos (Farabaugh, 1996). A transcriptase inversa, a diferenza do que fai a ARN polimerase II, pénsase que é unha poderosa causa da aparición de mutacións por desprazamento do marco de lectura. En experimentos só do 3 ao 13% de todo este tipo de mutacións ocorre por culpa da ARN polimerase II. En procariotas a taxa de erro que induce mutacións de dedsprazamento do marco de lectura é só de entre 0,0001 e 0,00001.[5]
Hai varios procesos biolóxicos que contribúen a evitar estas mutacións. As mutacións inversas prodúcense cando a secuencia mutada cambia outra vez á secuencia orixinal de tipo silvestre. Outra posibilidade para corrixir a mutación é o uso dunha mutación supresora. Isto compensa o efecto da mutación orixinal ao crear unha segunda mutación, que despraza a secuencia que fai que se lean os aminoácidos correctos. Tamén se pode usar un ARN guía para inserir ou eliminar uridina no ARNm despois da transcrición, isto permite que se lea o marco de lectura correcto.[1]
Importancia do codón ou triplete
[editar | editar a fonte]- Artigo principal: Código xenético.

Un codón é un conxunto de tres nucleótidos, un triplete que codifica un determinado aminoácido. O primeiro codón estabiliza o marco de lectura. A secuencia do esqueleto de aminoácidos dunha proteína defínese polos sucesivos tripletes contiguos.[6] Os codóns son a clave da tradución da información xenética para a síntese de proteínas. O marco de lectura establécese cando empeza a tradución do ARNm e mantense segundo se vai lendo un triplete tras outro. A lectura do código xenético está sometida a tres regras. Primeiro, os codóns lense en dirección 5' a 3'. Segundo, os codóns non se solapan e a mensaxe non tren ocos. A última regra, que xa se mencionou antes, é que a mensaxe se traduce nun marco de lectura fixo.[1]
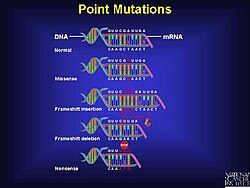
Mecanismo
[editar | editar a fonte]As mutacións de desprazamento do marco de lectura poden ocorrer aleatoriamente ou ser causadas por un estímulo externo. A detección de mutacións de desprazamento do marco de lectura poden ocorrer por varios métodos diferentes. Os desprazamentos de marco son só un tipo de mutación que pode orixinar proteínas incompletas ou incorrectas, pero explican unha significativa porcentaxe de erros no ADN.
Xenético ou ambiental
[editar | editar a fonte]- Artigo principal: Mutación.
As mutacións xenéticas a nivel de bases nucleotídicas poden ser inducidas por factores ambientais. Realizouse un estudo ambiental sobre a produción de mutacións por desprazamento de marco de electura inducidos pola luz UV polas ADN polimerases deficientes na actividade exonuclease 3′ → 5′. No estudo a secuencia normal foi cambiada para estudar os marcos de lectura. Os encimas pol I Kf de Escherichia coli e a ADN polimerase mutante de T7 desprovistos da actividade exonuclease 3′ → 5′ producen reversións inducidas pola luz UV a maior frecuencia que as súas equivalentes exonucleases funcionais. Os datos indican que a perda de actividade de corrección de probas incrementa a frecuencia de desprazamentos de marcos de lectura inducidos pola luz UV.[7]
Detección
[editar | editar a fonte]Fluorescencia
[editar | editar a fonte]Os efectos de bases veciñas e da estrutura secundaria para detectar a frecuencia de mutacións por desprazamento do marco de lectura foron investigadas en profundidade usando fluorescencia. O ADN etiquetado fluorescentemente, por medio de análogos de bases, permite estudar os cambios locais da secuencia de ADN.[8] Os estudos sobre os efectos da lonxitude da febra cebadora revelan que se observa unha mestura de equilibrio de catro conformacións de hibridación cando as bass do molde fan un bucle que forma unha protuberancia, é dicir, unha estrutura flanqueada a ambos os lados por ADN dúplex. En contraste, observouse unha estrutura de dobre bucle cunha conformación de ADN non usual no seu bordo de augas abaixo cando as bases extruídas estaban posicionadas na unión cebador-molde, o que mostra que os aliñamentos incorrectos poden ser modificados pola estrutura secundaria do ADN veciño.[9]
Secuenciación
[editar | editar a fonte]
A secuenciación de Sanger e a pirosecuenciación son dous métodos que se utilizaron para detectar mutacións por desprazamento do marco de lectura; porén, é probable que os datos xerados non sexan da máxima calidade. Aínda así, identificáronse 1,96 millóns de indeis por secuenciación de Sanger que non se solapan con outras bases de datos. Cando se observa unha mutación por despramento do marco de lectura compárase cos datos da Human Genome Mutation Database (HGMD) para determinar se a mutación ten un efecto prexudicial. Isto faise examinando catro características. Primeiro, a proporción entre o ADN afectado e conservado, segundo, a localización da mutación en relación ao transcrito, terceiro, a proporción de aminoácidos conservados e afectados e finalmente a distancia do indel ao final do exón.[10]
A secuenciación masivamente paralela é un novo método que se pode utilizar para detectar mutacións. Usando este método, poden secuenciarse ata 17 xigabases á vez, a diferenza dos rangos máis limitados que permite a secuenciación de Sanger de só aproximadamente 1 quilobase. Disponse de varias tecnoloxías para realizar este test e esta sendo considerado para usalo en aplicacións clínicas.[11] Cando se fan tests para diferentes carcinomas, os métodos correntes só permiten examinar un xene á vez. A secuenciación masivamene paralela pode testar varios cancros causantes de mutacións á vez a diferenza de diversos tests específicos.[12] Un experimento para determinar a exactitude deste método de secuenciación máis moderno para 21 xenes e non tivo resultados de falsos positivos para mutacións por desprazamento do marco de lectura.[13]
Diagnose
[editar | editar a fonte]Nos Estados Unidos a patente (5.958.684) de 1999 de Leeuwen, detalla os métodos e reactivos para a diagnose de doenzas causadas por ou asociadas cun xene que ten unha mutación somática que deu lugar a unha mutación por desprazamento da pauta de lectura. Os métodos requiren proporcionar un tecido ou mostra fluída e realizar unha análise xénica para mutacións por desprazamento do marco de lectura ou unha proteína xerada por este tipo de mutación. A secuencia de nucleótidos do xene sospeitoso é proporcionada por secuencias xénicas publicadas ou por clonación e secuencición do xene sospeitoso. A secuencia de aminoácidos codificada polo xene predícese despois.[14]
Frecuencia
[editar | editar a fonte]As mutacións ocorren malia as regras que gobernan o código xenético e os diversos mecanismos presentes nunha célula para asegurar a transferencia correcta da información xenética durante o proceso da replicación do ADN e durante a tradución; as mutacións por desprazamento do marco de lectura non son o único tipo. Hai polo menos outros dous tipos de mutacións puntuais recoñecidas, como son as mutacións con cambio de sentido e as mutacións sen sentido.[1] Unha mutación por desprazamento do marco de lectura pode cambiar drasticamente a capacidade de codificación (información xenetica) da mensaxe.[1] Pequenas insercións ou delecións (as de menos de 20 pares de bases) constitúen o 24% das mutacións que se manifestan en doenzas xenéticas actualmente recoñecidas.[10]
As mutacións por desprazamento do marco de lectura son máis comúns en rexións do ADN con repeticións. Unha razón para isto é o escorregamento do encima polimerase nas rexións repetidas, que permite que as mutacións entren na secuencia.[15] Poden realizarse experimentos para determinar a frecencia de mutacións por desprazamento do marco de lectura ao engadir ou eliminar un número de nucleótidos preestablecido. Fixéronse experimentos engadindo catro pares de bases, chamados experimentos +4, pero un equipo da Universidade Emory examinou a diferenza en frecuencia de mutación ao engadir ou eliminar un par de bases. Demostrouse que non había diferenzas na frecuencia entre a adición e eliminación de pares de bases. Hai, non obstante, unha diferenza no resultado final da proteína.[15]
A enfermidade de Huntington é un dos nove trastornos de reiteración de codón causado por mutacións de expansión da poliglutamina que inclúe a ataxia espinocerebelar (SCA) 1, 2, 6, 7 e 3, a atrofia muscular espinobulbar e a atrofia dentato-rubro-pálido-luisiana. Debe haber unha ligazón entre as doenzas causadas por mutacions de expansión da poliglutamina e da polialanina, xa que o marco despraza o produto do xene SCA3 orixinal que codifica CAG/poliglutaminas a GCA/polialaninas. O escorregamento ribosómico durante a tradución da proteína SCA3 propúxose como o mecanismo resultante do desprazamente desde o marco que codifica a poliglutamina ao da polialanina. Unha deleción de dinucleótidos ou a inserción dun só nucleótido sen o tracto de poliglutamina do exón 1 da huntingtina desprazaría a repetición CAG, marco codificante da poliglutamina, en +1 (desprazamento de marco de +1), a GCA, marco que codifica a polialanina e introduce un novo epítopo no C-terminal do exón 1 da hungtintina (APAAAPAATRPGCG).[16]
Enfermidades
[editar | editar a fonte]Varias enfermidades presentan mutacións por desprazamento do marco de lectura como polo menos parte da súa causa. Coñecer as mutacións prevalentes pode tamén axudar á diagnose da enfermidades. Actualmente hai intentos de usar mutacións por desprazamento do marco de lectura como beneficiosas no tratamento de doenzas, cambiando o marco de lectura dos aminoácidos.
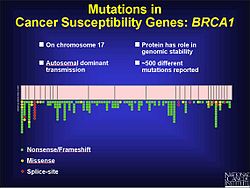

Cancro
[editar | editar a fonte]- Artigo principal: Cancro.
As mutacións por desprazamento do marco de lectura son un factor no cancro colorrectal e noutros cancros con inestabilidade de microsatélite. Como se indicou antes, estas mutacións é máis probable que ocorran nunha rexión con secuencia repetida. Cando a reparación de discordancias no ADN non arranxa a adición ou deleción de bases, é máis probable que estas mutacións sexan patoxénicas. Isto pode deberse en parte a que o tumor non recibe ordes de parar de crecer. Experimentos feitos en lévedos e bacterias axudan a mostrar as características dos microsatélites que poden contribuír a unha reparación de discordancias no ADN defectuosa. Estes inclúen a lonxitude do microsatélite, a composición do material xenético e o grao de pureza das repeticións. Baseándose en resultados experimentais os microsatélites máis longos teñen unha taxa maior de mutacións por desprazamento do marco de lectura. O ADN flanqueante pode tamén contribuír a mutacións deste tipo.[17] No cancro de próstata unha mutación por desprazamento do marco de lectura cambia o marco de lectura aberto (ORF) e impide a apoptose. Isto leva a un crecemento non regulado do tumor. Aínda que hai tres factores ambientais que contribúen á progresión do cancro de próstata, hai tamén un compoñente xenético. Durante a testaxe de rexións codificantes para identificar mutacións, descubríronse 116 variantes xenéticas, incluíndo 61 mutacións por desprazamento do marco de lectura.[18] Hai unhas 500 mutacións no cromosoma 17 que parecen xogar un papel no desenvolvemento dos cancros de mama e ovario no xene BRCA1, algunhas das cales son desprazamentos do marco de lectura.[19]
Enfermidade de Crohn
[editar | editar a fonte]A enfermidade de Crohn está asociada co xene NOD2. A mutación é unha inserción dunha citosina na posición 3020. Isto orixina un codón de terminación prematuro, acurtando a proteína que se supón que se transcribe. Cando a proteína pode formarse normalmente, responde aos lipopolisacáridos bacterianos, e a mutación 3020insC impide que a proteína sexa sensible.[20]
Fibrose quística
[editar | editar a fonte]A fibrose quística é unha doenza baseada en mutacións no xene do regulador de condutancia transmembrana da fibrose quística (CFTR). Identificáronse unhas 1500 mutacións, mais non todas causan a doenza.[21] A maioría dos casos de fibrose quística son o resultado da mutación ∆F508, que elimina o aminoácido. Dúas mutacións por desprazamento do marco de lectura son interesantes no diagnóstico da fibrose quística, CF1213delT e CF1154-insTC. Ambas as mutacións aparecen comunmente en tándem con polo menos outra mutación máis. Ambas as dúas causan unha pequena diminución no funcionamento dos pulmóns e aparecen nun 1% dos pacientes testados. Estas mutacións foron identificadas por secuenciación de Sanger.[22]
VIH
[editar | editar a fonte]- Artigo principal: VIH/SIDA.
O CCR5 é un dos cofactores para a entrada na célula asociados co VIH, máis frecuentemente implicados nas cepas indutoras de non sicitio, é máis frecuente en pacientes de VIH non avanzado que en pacientes de SIDA. Unha deleción de 32 pares de bases no CCR5 foi identificado como unha mutación que impide a probabilidade de infección por VIH. Esta rexión no marco de lectura aberto (ORF) contén unha mutación por desprazamento do marco de lectura orixinando un codon de terminación prematuro. Isto orixina a perda da función do correceptor de VIH in vitro. O CCR5-1 é considerado o tipo silvestre e CCR5-2 o alelo mutante. As persoas heterocigotas para esta mutación do CCR5 eran menos susceptibles ao desenvolvemento da infección por VIH. Nun estudo, entre persoas cunha grande exposición ao virus VIH, non houbo ningún homocigoto para a mutación CCR5 que dese positivo no test do VIH.[3]
Enfermidade de Tay–Sachs
[editar | editar a fonte]A enfermidade de Tay-Sachs é unha doenza mortal que afecta o sistema nervioso central. Dáse máis frecuentemente en bebés e nenos pequenos. A progresión da enfermidade empeza xa no útero, pero os síntomas non aparecen ata aproximadamente os 6 meses de idades. Non ten curación coñecida.[23] As mutacións no xene da β-hexosaminidase A (Hex A) afectan o comezo desta doenza, e describíronse 78 mutacións de diferentes tipos relacionadas, 67 das cales se sabe que causan a doenza. A maioría das mutacións observadas (65/78) son substitucións dun só nucleótido ou SNPs, 11 delas delecións, 1 grande e 10 pequenas e 2 insercións. 8 das mutacións observadas son desprazamentos de marcos de lectura, 6 delecións e 2 insercións. Unha inserción de 4 bases no exón 11 detéctase no 80% dos casos de Tay-Sachs que se dan na poboación xudía asquenací. As mutacións por desprazamento de marco de lectura orixinan un codón de finalización prematuro, que se sabe desempeña un papel no desenvolvemento da enfermidade en bebés. O atraso no comezo da enfermidade parece estar causado por 4 mutacións distintas, unha é unha deleción de 3 pares de bases.[24]
Síndrome de Smith–Magenis
[editar | editar a fonte]A síndrome de Smith-Magenis é unha complexa síndrome que causa deficiencia intelectual, trastornos do sono, problemas de comportamento e diversas anomalías craniofaciais, esqueléticas e viscerais. A mioría dos casos desta doenza débense a unha deleción común de ~3,5 Mb que comprende o xene do factor de transcrición RAI1 (ácido retinoico inducido 1). Outros casos ilustran a variabilidade do fenotipo desta síndrome que non se vira previamente na mutación de RAI1, incluíndo a perda de audición, ausencia de comportamentos autoabusivos, e atrasos globais leves. A secuenciación de RAI1 revelou unha mutación dun tracto heptamérico C (CCCCCCC) no exón 3 que tiña como resultado mutacións por desprazamento do marco de lectura. Das sete mutacións de cambio de marco das que se informou en tractos poli C de RAI1, catro casos (~57%) ocorrían neste tracto heptamérico C. Os resultados indican que este tracto heptamédico C é un punto quente de recombinación preferencial de insercións/delecións (indeis dun só nucleótido) e, por tanto unha diana primaria para a análise en pacientes que se sospeita que teñen mutacións en RAI1.[25]
Cardiomiopatía hipertrófica
[editar | editar a fonte]A cardiopatía hipertrófica é a causa máis común de morte súbita en xente nova, incluíndo atletas adestrados, e é causada por mutacións en xenes que codifican proteíns do sarcómero cardíaco. As mutacións no xene da troponina C (TNNC1) son unha rara causa xenética da cardiomiopatía hipertrófica. Un estudo recente indicou que unha mutación por desprazamento do marco de lectura (c.363dupG ou p.Gln122AlafsX30) na troponina C era a causa de cardiomiopatía hipertrófica (e da morte cardíaca súbita) en machos de 19 anos de idade.[26]
Tratamentos
[editar | editar a fonte]Atopar unha curación para as doenzas causadas por mutacións por desprazamento do marco de lectura é difícil, a pesar de que se investiga moito. Un exemplo é unha inmunodeficiencia primaria, unha condición herdada que pode orixinar un incremento de infeccións. Hai 120 xenes e 150 mutacións que xogan un papel nas inmunodeficiencias primarias. O tratamento estándar é actualmente a terapia xénica, pero este é un tratamento de alto risco e pode a miúdo orixinar outras enfermidades, como a leucemia. Entre os procedementos de terapia xénica están modificar a proteína de fusión nuclease dedo de zinc, clivando ambos os extremos da mutación, o que a elimina da secuencia. O salto de exón mediado por oligonucleótidos antisentido é outra posibilidade para a distrofia muscular de Duchenne. Este proceso permite satarse a mutación para que o resto da secuencia permanza dentro do marco e a funcionalidade da proteína siga intacta. Porén, isto non cura a doenza, só trata os síntomas e só é práctica en proteínas estruturais ou outros xenes repetitivos. Unha terceira forma de reparala é o mosaicismo reversor, que ocorre de forma natural creando unha mutación inversa ou unha mutación nun segundo sitio que corrixe o marco de lectura. Esta reversión pode ocorrer por recombinación intraxénica, conversión xénica mitótica, escorregamento do ADN nun segundo sitio ou reversión específica de sitio. Isto é posible en varias doenzas, como a inmunodeficiencia combinada grave ligada ao X, a síndrome de Wiskott–Aldrich e a síndrome de Bloom. Non hai fármacos nin outros medios farmacoxenómicos que poidan axudar aos pacientes de inmunodeficiencia primaria.[27]
Unha patente europea (EP1369126A1) de 2003 de Bork rexistra un método usado para a prevención de cancros e para o tratamento curativo de cancros e precancros como os tumores esporádicos por unha deficiente reparación de discordancias no ADN e os tumores asociados a cancro colorrectal non poliposo hereditario. A idea é utilizar inmunoterapia con mesturas combinatorias de péptidos derivados de mutacións por desprazamento do marco de lectura específicos de tumores para provocar a resposta de células T citotóxicas dirixidas especificamente contra as células tumorais.[28]
Notas
[editar | editar a fonte]- ↑ 1,00 1,01 1,02 1,03 1,04 1,05 1,06 1,07 1,08 1,09 Losick, Richard; Watson, James D.; Baker, Tania A.; Bell, Stephen; Gann, Alexander; Levine, Michael W. (2008). Molecular biology of the gene (6th ed.). San Francisco: Pearson/Benjamin Cummings. ISBN 978-0-8053-9592-1.
- ↑ "DNA Is Constantly Changing through the Process of Mutation". Nature. Consultado o 17 May 2019.
- ↑ 3,0 3,1 Zimmerman PA, Buckler-White A, Alkhatib G, Spalding T, Kubofcik J, Combadiere C, Weissman D, Cohen O, Rubbert A, Lam G, Vaccarezza M, Kennedy PE, Kumaraswami V, Giorgi JV, Detels R, Hunter J, Chopek M, Berger EA, Fauci AS, Nutman TB, Murphy PM (January 1997). "Inherited resistance to HIV-1 conferred by an inactivating mutation in CC chemokine receptor 5: studies in populations with contrasting clinical phenotypes, defined racial background, and quantified risk.". Molecular Medicine (Cambridge, Mass.) 3 (1): 23–36. PMC 2230106. PMID 9132277.
- ↑ Negoro S, Ohki T, Shibata N, Mizuno N, Wakitani Y, Tsurukame J, Matsumoto K, Kawamoto I, Takeo M, Higuchi Y (November 2005). "X-ray crystallographic analysis of 6-aminohexanoate-dimer hydrolase: molecular basis for the birth of a nylon oligomer-degrading enzyme". J Biol Chem 280 (47): 39644–52. PMID 16162506. doi:10.1074/jbc.m505946200.
- ↑ Zhang, J (August 2004). "Host RNA polymerase II makes minimal contributions to retroviral frame-shift mutations.". The Journal of General Virology 85 (Pt 8): 2389–95. PMID 15269381. doi:10.1099/vir.0.80081-0.
- ↑ Cox, Michael; Nelson, David R.; Lehninger, Albert L (2008). Lehninger principles of biochemistry. San Francisco: W.H. Freeman. ISBN 978-0-7167-7108-1.
- ↑ Sagher, Daphna; Turkington, Edith; Acharya, Sonia; Strauss, Bernard (July 1994). "Production of UV-induced Frameshift Mutations in Vitro by DNA Polymerases Deficient in 3′ → 5′ Exonuclease Activity". Journal of Molecular Biology 240 (3): 226–242. PMID 8028006. doi:10.1006/jmbi.1994.1437.
- ↑ Johnson, Neil P.; Walter A. Baase; Peter H. von Hippel (March 2004). "Low-energy circular dichroism of 2-aminopurine dinucleotide as a probe of local conformation of DNA and RNA". Proc Natl Acad Sci U S A 101 (10): 3426–31. PMC 373478. PMID 14993592. doi:10.1073/pnas.0400591101.
- ↑ Baase, Walter A.; Davis Jose; Benjamin C. Ponedel; Peter H. von Hippel; Neil P. Johnson (2009). "DNA models of trinucleotide frameshift deletions: the formation of loops and bulges at the primer–template junction". Nucleic Acids Research 37 (5): 1682–9. PMC 2655659. PMID 19155277. doi:10.1093/nar/gkn1042.
- ↑ 10,0 10,1 Hu, J; Ng, PC (9 February 2012). "Predicting the effects of frameshifting indels.". Genome Biology 13 (2): R9. PMC 3334572. PMID 22322200. doi:10.1186/gb-2012-13-2-r9.
- ↑ Tucker, Tracy; Marra, Marco; Friedman, Jan M. (2009). "Massively Parallel Sequencing: The Next Big Thing in Genetic Medicine". The American Journal of Human Genetics 85 (2): 142–154. PMC 2725244. PMID 19679224. doi:10.1016/j.ajhg.2009.06.022.
- ↑ Walsh, T.; Casadei, S.; Lee, M. K.; Pennil, C. C.; Nord, A. S.; Thornton, A. M.; Roeb, W.; Agnew, K. J.; Stray, S. M.; Wickramanayake, A.; Norquist, B.; Pennington, K. P.; Garcia, R. L.; King, M.-C.; Swisher, E. M. (2011). "From the Cover: Mutations in 12 genes for inherited ovarian, fallopian tube, and peritoneal carcinoma identified by massively parallel sequencing". Proc Natl Acad Sci U S A 108 (44): 18032–7. PMC 3207658. PMID 22006311. doi:10.1073/pnas.1115052108.
- ↑ Walsh, T.; Lee, M. K.; Casadei, S.; Thornton, A. M.; Stray, S. M.; Pennil, C.; Nord, A. S.; Mandell, J. B.; Swisher, E. M.; King, M.-C. (2010). "Detection of inherited mutations for breast and ovarian cancer using genomic capture and massively parallel sequencing". Proc Natl Acad Sci U S A 107 (28): 12629–33. PMC 2906584. PMID 20616022. doi:10.1073/pnas.1007983107.
- ↑ US Patent 5,958,684 (September 28, 1999) "Diagnosis of Neurodegenerative Disease" by Leeuwen et al
- ↑ 15,0 15,1 Harfe, BD; Jinks-Robertson, S (July 1999). "Removal of frameshift intermediates by mismatch repair proteins in Saccharomyces cerevisiae.". Molecular and Cellular Biology 19 (7): 4766–73. PMC 84275. PMID 10373526. doi:10.1128/MCB.19.7.4766.
- ↑ Davies, J E; Rubinsztein, D C (2006). "Polyalanine and polyserine frameshift products in Huntington's disease". Journal of Medical Genetics 43 (11): 893–896. PMC 2563184. PMID 16801344. doi:10.1136/jmg.2006.044222.
- ↑ Schmoldt, A; Benthe, HF; Haberland, G (1 September 1975). "Digitoxin metabolism by rat liver microsomes". Biochemical Pharmacology 24 (17): 1639–41. PMID 10. doi:10.1016/0006-2952(75)90094-5.
- ↑ Xu, XiaoLin; Zhu, KaiChang; Liu, Feng; Wang, Yue; Shen, JianGuo; Jin, Jizhong; Wang, Zhong; Chen, Lin; Li, Jiadong; Xu, Min (May 2013). "Identification of somatic mutations in human prostate cancer by RNA-Seq". Gene 519 (2): 343–7. PMID 23434521. doi:10.1016/j.gene.2013.01.046.
- ↑ "Cancer Genomics". National Cancer Institute at the National Institute of Health. Arquivado dende o orixinal o 18 de marzo de 2013. Consultado o 24 March 2013.
- ↑ Ogura Y, Bonen DK, Inohara N, Nicolae DL, Chen FF, Ramos R, Britton H, Moran T, Karaliuskas R, Duerr RH, Achkar JP, Brant SR, Bayless TM, Kirschner BS, Hanauer SB, Nuñez G, Cho JH (May 31, 2001). "A frameshift mutation in NOD2 associated with susceptibility to Crohn's disease." (PDF). Nature 411 (6837): 603–6. PMID 11385577. doi:10.1038/35079114. hdl:2027.42/62856.
- ↑ Farrell PM, Rosenstein BJ, White TB, Accurso FJ, Castellani C, Cutting GR, Durie PR, Legrys VA, Massie J, Parad RB, Rock MJ, Campbell PW (2008). "Guidelines for Diagnosis of Cystic Fibrosis in Newborns through Older Adults: Cystic Fibrosis Foundation Consensus Report". The Journal of Pediatrics 153 (2): S4–S14. PMC 2810958. PMID 18639722. doi:10.1016/j.jpeds.2008.05.005.
- ↑ Iannuzzi, MC; Stern, RC; Collins, FS; Hon, CT; Hidaka, N; Strong, T; Becker, L; Drumm, ML; White, MB; Gerrard, B (February 1991). "Two frameshift mutations in the cystic fibrosis gene.". American Journal of Human Genetics 48 (2): 227–31. PMC 1683026. PMID 1990834.
- ↑ "Learning About Tay-Sachs Disease". National Human Genome Research Institute. Consultado o 24 March 2013.
- ↑ Myerowitz, R (1997). "Tay-Sachs disease-causing mutations and neutral polymorphisms in the Hex A gene.". Human Mutation 9 (3): 195–208. PMID 9090523. doi:10.1002/(SICI)1098-1004(1997)9:3<195::AID-HUMU1>3.0.CO;2-7.
- ↑ Truong, Hoa T; Dudding, Tracy; Blanchard, Christopher L.; Elsea, Sarah H (2010). "Frameshift mutation hotspot identified in Smith-Magenis syndrome: case report and review of literature". BMC Medical Genetics 11 (1): 142. PMC 2964533. PMID 20932317. doi:10.1186/1471-2350-11-142.
- ↑ Chung WK, Kitner C, Maron BJ (June 2011). "Novel frameshift mutation in Troponin C ( TNNC1) associated with hypertrophic cardiomyopathy and sudden death". Cardiol Young 21 (3): 345–8. PMID 21262074. doi:10.1017/S1047951110001927.
- ↑ Hu, Hailiang; Gatti, Richard A (2008). "New approaches to treatment of primary immunodeficiencies: fixing mutations with chemicals". Current Opinion in Allergy and Clinical Immunology 8 (6): 540–6. PMC 2686128. PMID 18978469. doi:10.1097/ACI.0b013e328314b63b.
- ↑ European Patent [1] (December 10, 2003) "Use of coding microsatellite region frameshift mutation-derived peptides for treating cancer" by Bork et al
Véxase tamén
[editar | editar a fonte]| Wikimedia Commons ten máis contidos multimedia na categoría: Mutación por desprazamento do marco de lectura |
Outros artigos
[editar | editar a fonte]- Desprazamento do marco de lectura traducional ou ribosómico
- Codón
- Marco de lectura
- Enfermidade de Crohn
- Enfermidade de Tay-Sachs
Bibliografía
[editar | editar a fonte]- Farabaugh PJ (1996). "Programmed translational frameshifting". Annu. Rev. Genet. 30 (1): 507–28. PMC 239420. PMID 8982463. doi:10.1146/annurev.genet.30.1.507.
- Lewis, Ricki (2005). Human Genetics: Concepts and Applications (6th ed.). Boston MA: McGraw Hill. pp. 227–8. ISBN 978-0-07-111156-0.
- "Nylonase Enzymes". 20 April 2004. Consultado o 2 June 2009.
Ligazóns externas
[editar | editar a fonte]- Frameshift Mutation Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.
- NCBI dbSNP database — "un repositorio central para polimorfismos por substitucións nucleotídicas dunha base e delecións curtas e insercións"
- Wise2 - aliñamentos de proteínas con secuencias de ADN que permiten desprazamentos de marco de lectura e intróns
- FastY - base de datos para comparar unha secuencia de ADN cunha secuencia de proteínas, que permite ocos e desprazamentos de marco de lectura
- Path Arquivado 19 de xullo de 2011 en Wayback Machine. - ferramenta que compara dúas proteínas con desprazamento de marco de lectura (principio da retrotradución)
- HGMD - Human Genome Mutation Database
