Asgard (arqueas)
| Asgard (arqueas) | |||||||
|---|---|---|---|---|---|---|---|
| Clasificación científica | |||||||
| |||||||
 Distribución global de secuencias ensambladas metaxenomicamente de arqueas Asgard
| |||||||
| Phyla | |||||||
|
ver texto | |||||||
| Sinonimia | |||||||
| |||||||
Asgard ou Asgardarchaeota[2] é un superfilo proposto que comprende un grupo de arqueas que contén proteínas sinatura eucariotas.[3] Parece que os eucariotas, o dominio que contén os animais, plantas, fungos e protistas, xurdiron das Asgard,[4] nunha rama que contiña as Heimdallarchaeota.[5] Isto apoia a clasificación por medio do sistema de dous dominios en vez do sistema de tres dominios.[6][7]
Descubrimento e nomenclatura[editar | editar a fonte]
No verán de 2010 analizáronse sedimentos dunha mostra de núcleo de gravidade tomada no rift da dorsal de Knipovich no océano Glacial Ártico, preto do sitio da cheminea hidrotermal chamada Castelo de Loki. Sometéronse a análies metaxenómicas horizontes sedimentarios específicos nos que previamente se sabía que contiñan unha grande abundancia de novas liñaxes de arqueas.[8][9] En 2015 un equipo da Universidade de Uppsala propuxo o filo Lokiarchaeota baseándose en análises filoxenéticas feitas usando un conxunto de xenes codificantes de proteínas altamente conservadas.[10] O grupo nomeouse polo nome do deus nórdico que cambia de forma Loki, en alusión ao complexo de chemineas hidrotermais do cal se obtivo a primeira mostra xenómica.[11] O Loki da mitoloxía/relixión foi descrito como "unha figura abraiantemente complexa, confusa e ambivalente que foi o catalizador de incontables controversias eruditas non resoltas",[12] análogo ao papel das Lokiarchaeota nas discusións sobre a orixe dos eucariotas.[10][13]
En 2016 un equipo da Universidade de Texas descubriu os Thorarchaeota en mostras tomadas do río White Oak de Carolina do Norte, nomeados en referencia a Thor, outro deus nórdico.[14] Mostras tomadas no Castelo de Loki, Parque Nacional de Yellowstone, Baía de Aarhus, un acuífero preto do río Colorado, a lagoa Radiata de Nova Zelandia, chemineas hidrotermais preto da illa de Taketomi no Xapón, e no estuario do río White Oak nos Estados Unidos contiñan especies de Odinarchaeota e Heimdallarchaeota;[3] e seguindo a convención de nomenclatura baseada en deuses nórdicos, estes grupos foron nomeados Odín e Heimdallr, respectivamente. Os investigadores nomearon en consecuencia o superfilo que contiña estes organismos "Asgard", que era o fogar dos deuses na mitoloxía/relixión nórdica.[3] Cultiváronse dous espécimes de Lokiarchaeota, o que permitiu obter unha visión detallada da súa morfoloxía e características como o citoesqueleto.[15]
Descrición[editar | editar a fonte]
Proteínas[editar | editar a fonte]
Os membros de Asgard codifican moitas proteínas sinatura eucariotas, incluíndo novas GTPases, proteínas remodeladoras da membrana como ESCRT e SNF7, un sistema modificador de ubiquitina, e homólogos da vía da N-glicosilación.[3]
As arqueas Asgard teñen un citoesqueleto de actina regulado, e profilinas e xelsolinas que usan para interaccionar coas actinas eucariotas.[16][17] Ademais, a tubulina das arqueas Asgard das Odinarchaeota (OdinTubulina) que viven en ambientes hidrotermais foi identificada como unha tubulina xenuína. A OdinTubulina forma protómeros e protofilamentos moi similares aos microtúbulos eucariotas, se ben se ensambla formando sistemas anulares semellantes aos de FtsZ, o que indica que a OdinTubulina pode representar un intermediario evolutivo entre FtsZ e as tubulinas formadoras de microtúbulos.[18] Tamén parecen formar vesículas cando se ollan con microscopio electrónico crioxénico. Algunhas poden ter unha capa S con dominio PKD.[19] Tamén comparten cos eucariotas a expansión ES39 da molécula de ARNr da subunidade maior do ribosoma.[20] As agrupacións de xenes ou operóns que codifican proteínas ribosómicas adoitan estar menos conservados na súa organización no grupo Asgard que noutras arqueas, o que suxire que a orde dos xenes codificantes de proteínas ribosómicas pode seguir a filoxenia.[21]
Metabolismo[editar | editar a fonte]
-
Vías metabólicas das arqueas Asgard, que varían por filos.[22]
-
Vías metbólicas das arqueas Asgard, que varían segundo o ambiente.[22]
As arqueas Asgard son xeralmente anaerobios obrigados, aínda que os Kariarchaeota, Gerdarchaeota e Hodarchaeota poden ser aerobios facultativos.[23] Teñen unha vía de Wood–Ljungdahl e realizan a glicólise. Algúns membros poden ser autótrofos, heterótrofos, ou fotótrofos usando heliorrodopsina.[22] Un membro, o Candidatus Prometheoarchaeum syntrophicum, é sintrófico cunha proteobacteria redutora do xofre e unha arquea metanóxena.[19]
A RuBisCO que teñen non fixa o carbono, pero probblemente a usan para o rescate de nucleósidos.[22]
Ecoloxía[editar | editar a fonte]
As Asgard están amplamente distribuídas polo mundo, tanto xeograficamente coma en hábitat. Moitos dos clados coñecidos están restrinxidos aos sedimentos, mentres que as Lokiarchaeota, Thorarchaeota e outros clados ocupan hábitats moi diferentes. A salinidade e profundidade son importantes impulsores ecolóxicos para a maioría das arqueas Asgard. Outros hábitats inclúen o corpo de animais, a rizosfera das plantas, os sedimentos non salinos e solos, a superficie do mar, e as augas doces. Ademais, as Asgard están asociadas con outros organismos.[24]
Características similares ás eucariotas en subdivisións[editar | editar a fonte]
En 2017 atopouse que as especies do filo Heimdallarchaeota tiñan colas de histonas N-terminais como as dos nucleosomas, unha característica que previamente se pensaba que era exclusivamente eucariota. Outros dous filos arqueanos, ambos os dous fóra de Asgard, teñen tamén este tipo de colas, como se descubriu en 2018.[25]
En xaneiro de 2020 atopouse o Candidatus Prometheoarchaeum syntrophicum, un membro de Lokiarcheota, que establecía unha alimentación cruzada con dúas especies bacterianas. Debuxando unha analoxía coa simbioxénese, considéranse estas relacións unha posible ligazón entre os microorganismos procariotas simples e os microorganismos eucariotas complexos que ocoreu hai aproximadamente dous mil millóns de anos.[26][19]
Filoxenia[editar | editar a fonte]
As relacións filoxenéticas das arqueas Asgard estudáronas varios equipos de investigadores no século XXI.[5][4][27][23] Obtivéronse resultados variados, por exemplo usando 53 proteínas marcadoras da Genome Taxonomy Database.[28][29][30] En 2023 Eme, Tamarit, Caceres e colegas informaron que os Eukaryota se orixinaron profundamente dentro de Asgard, como irmáns das Hodarchaeales ambos dentro de Heimdallarchaeia.[31]
| Proteoarchaeota |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Taxonomía[editar | editar a fonte]
- Véxase tamén: Eucarioxénese.
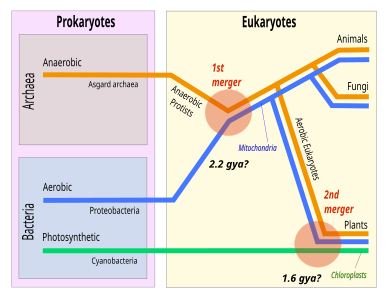
No escenario representado, os Eukaryota aparecen profundos na árbore das Asgard. Un escenario favorecido é a sintrofia, onde un organismo depende da alimentación do outro. Unha α-proteobacteria incorporouse para acabar formando as mitocondrias.[33] En cultivo, as arqueas Asgard existentes establecen varias dependencias sintróficas.[34] Gregory Fournier e Anthony Poole propuxeron que Asgard forma parte da "árbore dos eucariotas", formando un superfilo que chamaron "Eukaryomorpha", definido por "caraceres derivados compartidos" (proteínas sinatura eucariotas).[35]
A taxonomía é insegura e os nomes dos filos son, por tanto, algo especulativos. A lista de filos está baseada na List of Prokaryotic names with Standing in Nomenclature (LPSN)[36] e no National Center for Biotechnology Information (NCBI).[37]
- Filo Baldrarchaeota Caceres 2019
- Filo Borrarchaeota Liu et al. 2021
- Filo Freyrarchaeota corrig. Caceres 2019
- Filo Friggarchaeota Caceres 2019
- Filo Gefionarchaeota Caceres 2019
- Filo Gerdarchaeota Cai et al. 2020
- Filo Heimdallarchaeota Zaremba-Niedzwiedzka et al. 2017
- Filo Helarchaeota Seitz et al. 2019
- Filo Hermodarchaeota Liu et al. 2021
- Filo Hodarchaeota Liu et al. 2021
- Filo Idunnarchaeota Caceres 2019
- Filo Kariarchaeota Liu et al. 2021
- Filo Lokiarchaeota Spang et al. 2015
- Filo Njordarchaeota Xie et al. 2022
- Filo Odinarchaeota Zaremba-Niedzwiedzka et al. 2017
- Filo Sifarchaeota Farag et al. 2020
- Filo Sigynarchaeota Xie et al. 2022
- Filo Thorarchaeota Baker 2015
- Filo Tyrarchaeota Xie et al. 2022
- Filo Wukongarchaeota Liu et al. 2021
Elementos xenómicos[editar | editar a fonte]
Virus[editar | editar a fonte]
Descubríronse varios grupos no nivel de familia de virus asociados con arqueas Asgard usando metaxenómica.[38][39][40] Os virus foron asignados a hóspedes dos grupos Lokiarchaeia, Thorarchaeia, Odinarchaeia e Helarchaeia usando concordancias do espazador CRISPR cos correspondentes protoespazadores dentro dos xenomas virais. Dous grupos de virus (chamados 'verdandivirus') están relacionados con virus arqueanos e bacterianos da clase Caudoviricetes, é dicir, virus con cápside icosaédrica e colas helicoidais;[38][40] outros dous grupos (chamados 'skuldvirus') están relacionados distantemente cos virus sen cola arqueanos e bacterianos con cápsidas icosaédricas do realm (~dominio) Varidnaviria;[38][39] e o terceiro grupo de virus (chamados wyrdviruses) está relacionado con virus específicos de arqueas con partículas víricas con forma de limón (familia Halspiviridae).[38][39] Os virus identificáronse en sedimentos do mar profundo[38][40] e nunha fonte termal terrestre do Parque Nacional de Yellowstone.[39] Todos estes virus presentan unha similitude de secuencia moi baixa a outros virus coñecidos, pero están relacionadas xeralmente con virus procariotas previamente descritos,[41] sen ningunha afinidade significativa con virus de eucariotas.[42][38]
Elementos xenéticos móbiles[editar | editar a fonte]
Ademais dos virus, descubríronse varios grupos de elementos xenéticos móbiles con concordancia co espazador CRISPR, que están asociados con arqueas Asgard das liñaxes Lokiarchaeia, Thorarchaeia e Heimdallarchaeia.[38][43] Estes elementos móbiles non codifican proteínas distintivas virais recoñecibles e poderían representar tipos novos de virus ou plásmidos.
Notas[editar | editar a fonte]
- ↑ Fournier, G.P.; Poole, A.M. (2018). "A Briefly Argued Case That Asgard Archaea Are Part of the Eukaryote Tree". Frontiers in Microbiology 9: 1896. PMC 6104171. PMID 30158917. doi:10.3389/fmicb.2018.01896.
- ↑ Da Cunha, Violette; Gaia, Morgan; Gadelle, Daniele; et al. (xuño de 2017). "Lokiarchaea are close relatives of Euryarchaeota, not bridging the gap between prokaryotes and eukaryotes". PLOS Genetics 13 (6): e1006810. PMC 5484517. PMID 28604769. doi:10.1371/journal.pgen.1006810.
- ↑ 3,0 3,1 3,2 3,3 Zaremba-Niedzwiedzka, Katarzyna; Caceres, Eva F.; Saw, Jimmy H.; et al. (xaneiro de 2017). "Asgard archaea illuminate the origin of eukaryotic cellular complexity". Nature 541 (7637): 353–358. Bibcode:2017Natur.541..353Z. OSTI 1580084. PMID 28077874. doi:10.1038/nature21031.
- ↑ 4,0 4,1 Eme, Laura; Spang, Anja; Lombard, Jonathan; Stairs, Courtney W.; Ettema, Thijs J. G. (novembro de 2017). "Archaea and the origin of eukaryotes". Nature Reviews. Microbiology 15 (12): 711–723. PMID 29123225. doi:10.1038/nrmicro.2017.133.
- ↑ 5,0 5,1 Williams, Tom A.; Cox, Cymon J.; Foster, Peter G.; Szöllősi, Gergely J.; Embley, T. Martin (xaneiro de 2020). "Phylogenomics provides robust support for a two-domains tree of life". Nature Ecology & Evolution 4 (1): 138–147. PMC 6942926. PMID 31819234. doi:10.1038/s41559-019-1040-x.
- ↑ Nobs, Stephanie-Jane; MacLeod, Fraser I.; Wong, Hon Lun; Burns, Brendan P. (maio de 2022). "Eukarya the chimera: eukaryotes, a secondary innovation of the two domains of life?". Trends in Microbiology 30 (5): 421–431. PMID 34863611. doi:10.1016/j.tim.2021.11.003.
- ↑ Doolittle, W. Ford (febreiro de 2020). "Evolution: Two Domains of Life or Three?". Current Biology 30 (4): R177–R179. PMID 32097647. doi:10.1016/j.cub.2020.01.010.
- ↑ Jørgensen, Steffen Leth; Hannisdal, Bjarte; Lanzen, Anders; et al. (outubro de 2012). "Correlating microbial community profiles with geochemical data in highly stratified sediments from the Arctic Mid-Ocean Ridge". PNAS 109 (42): E2846–E2855. PMC 3479504. PMID 23027979. doi:10.1073/pnas.1207574109.
- ↑ Jørgensen, Steffen Leth; Thorseth, Ingunn H.; Pedersen, Rolf B.; et al. (4 de outubro de 2013). "Quantitative and phylogenetic study of the Deep Sea Archaeal Group in sediments of the Arctic mid-ocean spreading ridge". Frontiers in Microbiology 4: 299. PMC 3790079. PMID 24109477. doi:10.3389/fmicb.2013.00299.
- ↑ 10,0 10,1 Spang, Anja; Saw, Jimmy H.; Jørgensen, Steffen L.; et al. (maio de 2015). "Complex archaea that bridge the gap between prokaryotes and eukaryotes". Nature 521 (7551): 173–179. Bibcode:2015Natur.521..173S. PMC 4444528. PMID 25945739. doi:10.1038/nature14447.
- ↑ Yong, Ed. "Break in the Search for the Origin of Complex Life". The Atlantic. Consultado o 2018-03-21.
- ↑ von Schnurbein, Stefanie (novembro de 2000). "The Function of Loki in Snorri Sturluson's "Edda"". History of Religions 40 (2): 109–124. doi:10.1086/463618.
- ↑ Spang, Anja; Eme, Laura; Saw, Jimmy H.; et al. (marzo de 2018). "Asgard archaea are the closest prokaryotic relatives of eukaryotes". PLOS Genetics 14 (3): e1007080. PMC 5875740. PMID 29596421. doi:10.1371/journal.pgen.1007080.
- ↑ Seitz, Kiley W.; Lazar, Cassandre S.; Hinrichs, Kai-Uwe; et al. (xullo de 2016). "Genomic reconstruction of a novel, deeply branched sediment archaeal phylum with pathways for acetogenesis and sulfur reduction". The ISME Journal 10 (7): 1696–1705. PMC 4918440. PMID 26824177. doi:10.1038/ismej.2015.233.
- ↑ Rodrigues-Oliveira, Thiago; Wollweber, Florian; Ponce-Toledo, Rafael I.; et al. (2023-01-12). "Actin cytoskeleton and complex cell architecture in an Asgard archaeon". Nature 613 (7943): 332–339. Bibcode:2023Natur.613..332R. ISSN 0028-0836. PMC 9834061. PMID 36544020. doi:10.1038/s41586-022-05550-y.
- ↑ Akıl, Caner; Robinson, Robert C. (outubro de 2018). "Genomes of Asgard archaea encode profilins that regulate actin". Nature 562 (7727): 439–443. Bibcode:2018Natur.562..439A. PMID 30283132. doi:10.1038/s41586-018-0548-6.
- ↑ Akıl, Caner; Tran, Linh T.; Orhant-Prioux, Magali; Baskaran, Yohendran; Manser, Edward; Blanchoin, Laurent; Robinson, Robert C. (agosto de 2020). "Insights into the evolution of regulated actin dynamics via characterization of primitive gelsolin/cofilin proteins from Asgard archaea". Proceedings of the National Academy of Sciences of the United States of America 117 (33): 19904–19913. Bibcode:2020PNAS..11719904A. PMC 7444086. PMID 32747565. doi:10.1073/pnas.2009167117.
- ↑ Akıl, Caner; Ali, Samson; Tran, Linh T.; et al. (marzo de 2022). "Structure and dynamics of Odinarchaeota tubulin and the implications for eukaryotic microtubule evolution". Science Advances 8 (12): eabm2225. Bibcode:2022SciA....8M2225A. PMC 8956254. PMID 35333570. doi:10.1126/sciadv.abm2225.
- ↑ 19,0 19,1 19,2 Imachi, Hiroyuki; Nobu, Masaru K.; Nakahara, Nozomi; et al. (xaneiro de 2020). "Isolation of an archaeon at the prokaryote-eukaryote interface". Nature 577 (7791): 519–525. Bibcode:2020Natur.577..519I. PMC 7015854. PMID 31942073. doi:10.1038/s41586-019-1916-6.
- ↑ Penev, Petar I.; Fakhretaha-Aval, Sara; Patel, Vaishnavi J.; et al. (outubro de 2020). "Supersized Ribosomal RNA Expansion Segments in Asgard Archaea". Genome Biology and Evolution 12 (10): 1694–1710. PMC 7594248. PMID 32785681. doi:10.1093/gbe/evaa170.
- ↑ Tirumalai, Madhan R.; Raghavan, Sivaraman V.; Kutty, Layla A.; Song, Eric L.; Fox, George E. (outubro de 2023). "Ribosomal Protein Cluster Organization in Asgard Archaea". Archaea (Hindawi) 2023: 16. doi:10.1155/2023/5512414.
- ↑ 22,0 22,1 22,2 22,3 MacLeod, Fraser; Kindler, Gareth S.; Wong, Hon Lun; Chen, Ray; Burns, Brendan P. (2019). "Asgard archaea: Diversity, function, and evolutionary implications in a range of microbiomes". AIMS Microbiology 5 (1): 48–61. PMC 6646929. PMID 31384702. doi:10.3934/microbiol.2019.1.48.
- ↑ 23,0 23,1 Liu, Yang; Makarova, Kira S.; Huang, Wen-Cong; et al. (2020). "Expanding diversity of Asgard archaea and the elusive ancestry of eukaryotes". bioRxiv. doi:10.1101/2020.10.19.343400.
- ↑ Cai, Mingwei; Richter-Heitmann, Tim; Yin, Xiuran; et al. (2021). "Ecological features and global distribution of Asgard archaea". Science of the Total Environment 758: 143581. Bibcode:2021ScTEn.758n3581C. ISSN 0048-9697. PMID 33223169. doi:10.1016/j.scitotenv.2020.143581.
- ↑ Henneman, Bram; van Emmerik, Clara; van Ingen, Hugo; Dame, Remus T. (setembro de 2018). "Structure and function of archaeal histones". PLOS Genetics 14 (9): e1007582. Bibcode:2018BpJ...114..446H. PMC 6136690. PMID 30212449. doi:10.1371/journal.pgen.1007582.
- ↑ Zimmer, Carl (15 de xaneiro de 2020). "This Strange Microbe May Mark One of Life's Great Leaps - A organism living in ocean muck offers clues to the origins of the complex cells of all animals and plants.". The New York Times. Consultado o 16 de xaneiro de 2020.
- ↑ Liu, Yang; Makarova, Kira S.; Huang, Wen-Cong; Wolf, Yuri I.; Nikolskaya, Anastasia; Zhang, Xinxu; et al. (maio de 2021). "Expanded diversity of Asgard archaea and their relationships with eukaryotes". Nature 593 (7860): 553–557. Bibcode:2021Natur.593..553L. PMID 33911286. doi:10.1038/s41586-021-03494-3.
- ↑ "GTDB release 08-RS214". Genome Taxonomy Database. Consultado o 10 de maio de 2023.
- ↑ "ar53_r214.sp_label". Genome Taxonomy Database. Consultado o 10 de maio de 2023.
- ↑ "Taxon History". Genome Taxonomy Database. Consultado o 10 de maio de 2023.
- ↑ Eme, Laura; Tamarit, Daniel; Caceres, Eva F.; et al. (2023-06-14). "Inference and reconstruction of the heimdallarchaeial ancestry of eukaryotes". Nature 618 (7967): 992–999. Bibcode:2023Natur.618..992E. PMC 10307638. PMID 37316666. doi:10.1038/s41586-023-06186-2.
- ↑ Latorre, A.; Durban, A.; Moya, A.; Pereto, J. (2011). "The role of symbiosis in eukaryotic evolution". En Gargaud, M.; López-Garcìa, P.; Martin H. Origins and Evolution of Life: An astrobiological perspective. Cambridge: Cambridge University Press. pp. 326–339. ISBN 978-0-521-76131-4. Arquivado dende o orixinal o 24 de marzo de 2019. Consultado o 27 de agosto de 2017.
- ↑ López-García, Purificación; Moreira, David (xullo de 2019). "Eukaryogenesis, a syntrophy affair". Nature Microbiology 4 (7): 1068–1070. PMC 6684364. PMID 31222170. doi:10.1038/s41564-019-0495-5.
- ↑ Rodrigues-Oliveira, Thiago; Wollweber, Florian; Ponce-Toledo, Rafael I.; Xu, Jingwei; Rittmann, Simon K.-M. R.; Klingl, Andreas; Pilhofer, Martin; Schleper, Christa (xaneiro de 2023). "Actin cytoskeleton and complex cell architecture in an Asgard archaeon". Nature 613 (7943): 332–339. Bibcode:2023Natur.613..332R. ISSN 1476-4687. PMC 9834061. PMID 36544020. doi:10.1038/s41586-022-05550-y.
- ↑ Fournier, Gregory P.; Poole, Anthony M. (2018-08-15). "A Briefly Argued Case That Asgard Archaea Are Part of the Eukaryote Tree". Frontiers in Microbiology 9: 1896. ISSN 1664-302X. PMC 6104171. PMID 30158917. doi:10.3389/fmicb.2018.01896.
- ↑ Euzéby, J.P. "Superphylum "Asgardarchaeota"". List of Prokaryotic names with Standing in Nomenclature (LPSN). Consultado o 2021-06-27.
- ↑ "Asgard group". National Center for Biotechnology Information (NCBI) taxonomy database. Consultado o 2021-03-20.
- ↑ 38,0 38,1 38,2 38,3 38,4 38,5 38,6 Medvedeva, S.; Sun, J.; Yutin, N.; Koonin, Eugene V.; Nunoura, T.; Rinke, C.; Krupovic, M. (xullo de 2022). "Three families of Asgard archaeal viruses identified in metagenome-assembled genomes". Nature Microbiology 7 (7): 962–973. PMID 35760839. doi:10.1038/s41564-022-01144-6.
- ↑ 39,0 39,1 39,2 39,3 Tamarit, D.; Caceres, E.F.; Krupovic, M.; Nijland, R.; Eme, L.; Robinson, N.P.; Ettema, T.J.G. (xullo de 2022). "A closed Candidatus Odinarchaeum chromosome exposes Asgard archaeal viruses". Nature Microbiology 7 (7): 948–952. PMC 9246712. PMID 35760836. doi:10.1038/s41564-022-01122-y.
- ↑ 40,0 40,1 40,2 Rambo IM, Langwig MV, Leão P, De Anda V, Baker BJ. Genomes of six viruses that infect Asgard archaea from deep-sea sediments. Nat Microbiol. 2022 xullo;7(7):953-961. doi: 10.1038/s41564-022-01150-8. Epub 2022 xuño 27. PMID 35760837.
- ↑ Prangishvili, D.; Bamford, D.H.; Forterre, P.; Iranzo, J.; Koonin, Eugene V.; Krupovic, M. (novembro de 2017). "The enigmatic archaeal virosphere". Nature Reviews. Microbiology 15 (12): 724–739. PMID 29123227. doi:10.1038/nrmicro.2017.125.
- ↑ Alarcón-Schumacher, T.; Erdmann, S. (xullo de 2022). "A trove of Asgard archaeal viruses". Nature Microbiology 7 (7): 931–932. PMID 35760838. doi:10.1038/s41564-022-01148-2.
- ↑ Wu, F.; Speth, D.R.; Philosof, A.; et al. (febreiro de 2022). "Unique mobile elements and scalable gene flow at the prokaryote-eukaryote boundary revealed by circularized Asgard archaea genomes". Nature Microbiology 7 (2): 200–212. PMC 8813620. PMID 35027677. doi:10.1038/s41564-021-01039-y.
Véxase tamén[editar | editar a fonte]
Ligazóns externas[editar | editar a fonte]
- Traci Watson: The trickster microbes that are shaking up the tree of life, en: Nature, 14 de maio de 2019

![Vías metabólicas das arqueas Asgard, que varían por filos.[22]](http://upload.wikimedia.org/wikipedia/commons/thumb/7/7f/Asgard_archaea_Phyla_%28cropped%29.png/634px-Asgard_archaea_Phyla_%28cropped%29.png)
![Vías metbólicas das arqueas Asgard, que varían segundo o ambiente.[22]](http://upload.wikimedia.org/wikipedia/commons/thumb/a/aa/Asgard_archaea_in_various_environments_%28cropped%29.png/638px-Asgard_archaea_in_various_environments_%28cropped%29.png)

