Indel sinatura conservado
Os indeis sinatura conservados ou insercións e delecións sinatura conservados (abreviados como CSI, polas súas siglas en inglés) son marcadores moleculares que se encontran nas secuencias de proteínas, moi importantes para establecer as relacións filoxenéticas entre distintos grupos de seres vivos.[1][2] Os CSIs, orixínanse por raros cambios xenéticos, teñen xeralmente un tamaño definido e están flanqueados a ambos os lados por rexións conservadas, que aseguran a súa fiabilidade como marcadores filoxenéticos útiles. Aínda que os indeis poden ser insercións ou delecións arbitrarias, os CSIs defínense só como aqueles indeis das proteínas que están presentes dentro de rexións conservadas da proteína (noutro caso eses indeis non serían indeis sinatura conservados).[2][3][4][5]
Os CSIs que están restrinxidos a un clado particular ou grupo de especies, proporcionan xeralmente bos marcadores filoxenéticos sobre a descendencia evolutiva común.[2] Debido á rareza e natureza moi específica de tales cambios, é menos probable que se tivesen orixinado de forma independente por evolución converxente ou paralela (é dicir, homoplasia) ou sinapomorfía. Outros factores que adoitan ser confusos como as diferenzas nas taxas de evolución en diferentes sitios ou entre diferentes especies tampouco afectan xeralmente á interpretación dos CSIs.[2][3] Determinando a presenza ou ausencia de CSIs en especies fóra do grupo, pode inferirse se a forma ancestral do CSI foi unha inserción ou deleción e isto pode utilizarse para elaborar árbores enraizadas que indiquen as relacións filoxenéticas entre os organismos.[1][2]
A maioría dos CSIs que foron identificados mostran un alto valor preditivo e xeralmente manteñen a especificidade polos clados de especies identificados orixinalmente. Xa que logo, baseándose na súa presenza ou ausencia, sería posible identificar tanto especies coñecidas coma mesmo especies previamente descoñecidas que pertencen a estes grupos e viven en distintos ambientes.[3]
Tipos de CSIs[editar | editar a fonte]
CSIs específicos de grupo[editar | editar a fonte]

Os CSIs específicos de grupo son compartidos xeralmente por diferentes especies que pertencen a un determinado taxon (como xénero, familia, orde, clase, filo) pero non están presentes noutros grupos. Estes CSIs moi probablemente foron introducidos no antepasado do grupo de especies antes que que diverxisen os membros do taxon. Proporcionan un medio de tipo molecular para distinguir os membros dun determinado taxon do resto dos organismos.[2][5]
A figura 1 mostra un exemplo do CSI de 5 aminoácidos (aa) que se encontra en todas as especies do taxon X. Isto é unha característica distintiva dese taxon, xa que non se encontra noutras especies. Esta sinatura foi introducida probablemente no antepasado común de todas as especies do taxon. De modo similar outras sinaturas específicas de grupo (que non se mostran) poderían ser compartidas por A1 e A2 ou B1 e B2 etc., ou mesmo por X1 e X2 ou por X3 e X4 etc. Os grupos A, B, C, D e X, deste diagrama poderían corresponder a varios filos bacterianos ou eucarióticos.[6]
Os CSIs específicos de grupo foron utilizados no pasado para determinar as relacións filoxenéticas de varios filos bacterianos e de subgrupos dentro deles. Por exemplo unha inserción de 3 aminoácidos só era compartida por membros do filo Thermotogae na proteína ribosómica L7/L12 50S, nunha rexión moi conservada (de 82-124 aminoácidos). Esta non estaba presente en ningunha outra especie de bacteria e podía usarse para caracterizar a membros do filo Thermotogae distinguíndoos do resto das bacterias. Os CSIs específicos de grupo tamén se utilizaron para caracterizar os subgrupos dentro do filo Thermotogae.[7]
CSIs multigrupo ou de Liña Principal[editar | editar a fonte]

Os CSIs de Liña Principal son aqueles nos que se comparte unha inserción ou deleción por varios filos maiores, pero que está ausente noutros filos.[2]
A figura 2 mostra un exemplo de CSI de 5 aminoácidos encontrado nunha rexión conservada que está xeralmente presente nas especies que pertencen aos filos X, Y e Z, pero está ausente noutros filos (A, B e C). Esta sinatura indica unha relación específica entre os taxons X, Y e Z e tamén A, B e C. Baseándose na presenza ou ausencia dun indel deste tipo, en especies fóra do grupo (Archaea), pode inferirse se o indel é unha inserción ou unha deleción, e cal dos dous grupos, o grupo A, B, C ou o X, Y, Z, é o ancestral.[6]
Os CSIs de Liña Principal utilizáronse no pasado para determinar a relación filoxenética de varios filos bacterianos. O gran CSI duns 150-180 aminoácidos situado dentro da rexión conservada da xirase B (entre os aminoácidos 529-751), é xeralmente compartida por varias especies de Proteobacteria, Chlamydiales, Planctomycetes e Aquificales. Este CSI está ausente noutros filos procarióticos ancestrais como as Archaea.[8] Igualmente, un gran CSI duns 100 aminoácidos en homólogos da proteína RpoB (entre os aminoácidos 919-1058) está presente en varias especies de Proteobacteria, Bacteroidetes-Chlorobi, Chlamydiales, Planctomycetes e Aquificales. Este CSI está ausente noutros filos bacterianos ancestrais e nas Archaea.[9][10] En ambos os casos pode inferirse que os grupos que carecen deste CSI son ancestrais.
Estudos evolutivos baseados nos CSIs[editar | editar a fonte]


Un aspecto chave da filoxenia bacteriana é comprender como diferentes especies bacterianas están relacionadas unhas con outras e a súa orde de ramificación desde un antepasado común. Actualmente a maioría das árbores filoxenéticas están baseadas no ARNr 16S ou outros xenes ou proteínas. Estas árbores non sempre permiten resolver cuestións filoxenéticas fundamentais cun grao alto de certeza.[11][12][13][14][15] Con todo, nos últimos anos o descubrimento e análise de CSIs en moitas proteínas universalmente distribuídas axudou moito nesta tarefa. Os sucesos xenéticos que deron lugar a estes CSIs suponse que ocorreron en puntos de ramificación evolutiva importantes e os seus patróns de distribución de especies proporcionan valiosa información sobre a orde de ramificación e as interrelacións entre diferentes filos bacterianos.[1][2][7]
Thermotogae[editar | editar a fonte]
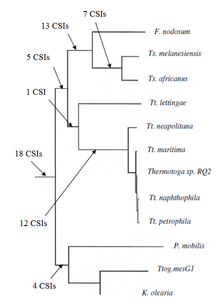
Recentemente caracterizáronse as reaccións filoxenéticas do grupo bacteriano Thermotogae baseándose no estudo de CSIs. Antes, non se coñecía ningún marcador molecular bioquímico que puidese servir para distinguir claramente as especies deste filo doutras bacterias. Descubríronse máis de 60 CSIs que eran específicos para o filo enteiro de Thermotogae ou aos seus diferentes subgrupos. Hai 18 CSIs que están presentes unicamente en varias especies de Thermotogae e proporcionan marcadores moleculares para o filo. Adicionalmente, hai moitos CSIs que son específicos de varios subgrupos de Thermotogae. 12 CSIs eran específicos dun clado formado por varias especies do xénero Thermotoga excepto T. lettingae. 14 CSIs eran específicos dun clado formado polos xéneros Fervidobacterium e Thermosiphon e 18 CSIs eran específicos do xénero Thermosiphon.[7]
Ultimamente informouse de 16 CSIs que son compartidos por algunhas ou todas as especies de Thermotogae ou algunhas especies doutros taxons como Archaea, Aquificae, Firmicutes, Proteobacteria, Deinococcus, Fusobacteria, Dictyoglomus, Chloroflexi e os eucariotas. A presenza compartida dalgúns destes CSIs podería deberse a unha transferencia lateral de xenes entre estes grupos. Con todo, o número de CSIs que son normalmente compartidos con outros taxons é moito menor que os que son específicos das Thermotogae e non mostran ningún patrón específico. Por tanto, non teñen un efecto significativo na distinción das Thermotogae.[7]
Arqueas[editar | editar a fonte]
As Crenarchaeota mesofílicas foron situadas recentemente nun novo filo de arqueas denominado Thaumarchaeota. Porén, hai moi poucos marcadores moleculares que poidan distinguir este grupo de arqueas do filo das Crenarchaeota. Realizouse un estudo filoxenético detallado utilizando CSIs para distinguir estes filos en termos moleculares. Atopouse que 6 CSIs só aparecían en varias Thaumarchaeota, como Cenarchaeum symbiosum, Nitrosopumilus maritimus e varias crenarqueotas mariñas non cultivadas. Outros 3 CSIs eran compartidos comunmente entre especies que pertencían a Thaumarchaeota e Crenarchaeota. Ademais, varios CSIs eran específicos de diferentes ordes de Crenarchaeota (3 CSIs de Sulfolobales, 5 CSIs de Thermoproteales, e 2 CSIs comúns de Sulfolobales e Desulfurococcales). As sinaturas descritas proporcionan novos medios de distinguir Crenarchaeota e Thaumarchaeota, ademais, poderían utilizarse como unha ferramenta para a clasificación e identificación de especies relacionadas.[16]
Pasteurellales[editar | editar a fonte]
Os membros da orde Pasteurellales distínguense actualmente baseándose principalmente na ramificación da súa árbore de ARNr 16S. Coñécense moi poucos marcadores moleculares cos que se poidan distinguir os membros desta orde doutras bacterias. Nun estudo de CSIs recente utilizado para dilucidar as relacións filoxenéticas nesta orde, descubríronse máis de 40 CSIs que eran compartidos só por todas ou a maioría das especies desta orde. Na orde formáronse dous clados principais: clado I e II. O clado I, que comprende a Aggregatibacter, Pasteurella, Actinobacillus succinogenes, Mannheimia succiniciproducens, Haemophilus influenzae e Haemophilus somnus, está apoiado por 13 CSIs. O clado II comprende Actinobacillus pleuropneumoniae, Actinobacillus minor, Haemophilus ducreyi, Mannheimia haemolytica e Haemophilus parasuis, e está apoiado por 9 CSIs. Baseándose nestes resultados propúxose que Pasteurellales, que está actualmente formado por unha soa familia, sexa dividido en polo menos dúas. Ademais, as sinaturas descritas proporcionarían novos métodos de identificar especies de Pasteurellales non descubertas.[17]
Gammaproteobacterias[editar | editar a fonte]
A clase Gammaproteobacteria é un dos grupos máis grandes de bacterias. Actualmente distínguese doutras bacterias só polas árbores filoxenéticas de ARNr 16S. Non se coñecen características moleculares únicas da clase ou dos seus diferentes subgrupos. Un detallado estudo de CSIs serviu opara coñecer mellor a filoxenia desta clase. Primeiramente, creouse unha árbore filoxenética baseada nas secuencias concatenadas de varias proteínas distribuídas universalmente. A orde de ramificación das diferentes ordes da clase Gammaproteobacteria (do máis recente ao primeiro que diverxiu) era: Enterobacteriales >Pasteurellales >Vibrionales, Aeromonadales >Alteromonadales >Oceanospirillales, Pseudomonadales >Chromatiales, Legionellales, Methylococcales, Xanthomonadales, Cardiobacteriales, Thiotrichales. Ademais, descubríronse 4 CSIs que eran únicos da maioría das especies da clase Gammaproteobacteria. Unha deleción de 2 aminoácidos na transformilase AICAR era compartida exclusivamente por todas as gammaproteobacterias excepto Francisella tularensis. Unha deleción de 4 aminoácidos na subunidade b da ARN polimerase e unha deleción 1 aminoácido na proteína ribosómica L16 só se atoparon en varias especies das ordes Enterobacteriales, Pasteurellales, Vibrionales, Aeromonadales e Alteromonadales, pero non se atoparon noutras gammaproteobacterias. Por último, unha deleción de 2 aminoácidos na leucil-ARNt sintetase estaba presente comunmente nas ordes superiores da clase Gammaproteobacteria e nalgúns membros da orde Oceanospirillales.[18] Outro estudo baseado en CSIs identificou 4 CSIs exclusivos da orde Xanthomonadales. En conxunto, estes dous feitos mostran que Xanthomonadales é un grupo monofilético, que é ancestral con respecto ás outras Gammaproteobacterias; o que indica que Xanthomonadales é unha subdivisión independente, e constitúe unha das liñaxes que antes se ramificou no clado das Gammaproteobacterias.[4][18]
Fungos[editar | editar a fonte]
As relacións filoxenéticas exactas entre plantas, animais e fungos non se comprende totalmente. Realizouse un pequeno estudo baseado en CSIs para intentar dilucidar esta relación. Utilizáronse catro CSIs para situar a animais e fungos xuntos como un grupo monofilético do que estaban excluídas as plantas. Estes atopáronse en dous proteínas celulares esenciais, o factor de elongación 1 alfa 1 da tradución eucariótica e a enolase. Porén, tradicionalmente a relación específica entre os fungos e os animais non foi admitida.[1]
Notas[editar | editar a fonte]
- ↑ 1,0 1,1 1,2 1,3 Baldauf, S. L. (1993). "Animals and Fungi are Each Other's Closest Relatives: Congruent Evidence from Multiple Proteins". Proceedings of the National Academy of Sciences 90 (24). PMC 48023. PMID 8265589. doi:10.1073/pnas.90.24.11558.
- ↑ 2,0 2,1 2,2 2,3 2,4 2,5 2,6 2,7 Gupta, Radhey S. (1998). "Protein Phylogenies and Signature Sequences: A Reappraisal of Evolutionary Relationships among Archaebacteria, Eubacteria, and Eukaryotes". Microbiology and Molecular Biology Reviews 62 (4): 1435–91. PMC 98952. PMID 9841678.
- ↑ 3,0 3,1 3,2 Gupta, Radhey S.; Griffiths, Emma (2002). "Critical Issues in Bacterial Phylogeny". Theoretical Population Biology 61 (4): 423–34. PMID 12167362. doi:10.1006/tpbi.2002.1589.
- ↑ 4,0 4,1 Cutiño-Jiménez, Ania M.; Martins-Pinheiro, Marinalva; Lima, Wanessa C.; Martín-Tornet, Alexander; Morales, Osleidys G.; Menck, Carlos F.M. (2010). "Evolutionary placement of Xanthomonadales based on conserved protein signature sequences". Molecular Phylogenetics and Evolution 54 (2): 524–34. PMID 19786109. doi:10.1016/j.ympev.2009.09.026.
- ↑ 5,0 5,1 Rokas, Antonis; Holland, Peter W.H. (2000). "Rare genomic changes as a tool for phylogenetics". Trends in Ecology & Evolution 15 (11). PMID 11050348. doi:10.1016/S0169-5347(00)01967-4.
- ↑ 6,0 6,1 Gupta, Radhey. "Conserved Inserts and Deletions in Protein Sequences". Bacterial Phylogeny. Gupta RS. Consultado o 2 April 2012.
- ↑ 7,0 7,1 7,2 7,3 Gupta, Radhey S.; Bhandari, Vaibhav (2011). "Phylogeny and molecular signatures for the phylum Thermotogae and its subgroups". Antonie van Leeuwenhoek 100 (1): 1–34. PMID 21503713. doi:10.1007/s10482-011-9576-z.
- ↑ Griffiths, E.; Gupta, R. S. (2007). "Phylogeny and shared conserved inserts in proteins provide evidence that Verrucomicrobia are the closest known free-living relatives of chlamydiae". Microbiology 153 (8). PMID 17660429. doi:10.1099/mic.0.2007/009118-0.
- ↑ Gupta, Radhey S. (2003). "Evolutionary relationships among photosynthetic bacteria". Photosynthesis Research 76 (1–3): 173–83. PMID 16228576. doi:10.1023/A:1024999314839.
- ↑ Griffiths, Emma; Gupta, Radhey S. (2004). "Signature sequences in diverse proteins provide evidence for the late divergence of the Order Aquificales" (PDF). International Microbiology 7 (1): 41–52. PMID 15179606. Arquivado dende o orixinal (PDF) o 14 de abril de 2012. Consultado o 06 de agosto de 2013.
- ↑ Brown, James R.; Douady, Christophe J.; Italia, Michael J.; Marshall, William E.; Stanhope, Michael J. (2001). "Universal trees based on large combined protein sequence data sets". Nature Genetics 28 (3): 281–5. PMID 11431701. doi:10.1038/90129.
- ↑ Cavalier-Smith, T (2002). "The neomuran origin of archaebacteria, the negibacterial root of the universal tree and bacterial megaclassification". International Journal of Systematic and Evolutionary Microbiology 52 (1): 7–76. PMID 11837318.
- ↑ Ciccarelli, F. D.; Doerks, T; Von Mering, C; Creevey, CJ; Snel, B; Bork, P (2006). "Toward Automatic Reconstruction of a Highly Resolved Tree of Life". Science 311 (5765): 1283–7. PMID 16513982. doi:10.1126/science.1123061.
- ↑ Daubin, V.; Gouy, M; Perrière, G (2002). "A Phylogenomic Approach to Bacterial Phylogeny: Evidence of a Core of Genes Sharing a Common History". Genome Research 12 (7): 1080–90. PMC 186629. PMID 12097345. doi:10.1101/gr.187002.
- ↑ Eisen, Jonathan A. (1995). "The RecA Protein as a Model Molecule for Molecular Systematic Studies of Bacteria: Comparison of Trees of RecAs and 16S rRNAs from the Same Species". Journal of Molecular Evolution 41 (6): 1105–23. PMC 3188426. PMID 8587109.
- ↑ Gupta, Radhey S.; Shami, Ali (2010). "Molecular signatures for the Crenarchaeota and the Thaumarchaeota". Antonie van Leeuwenhoek 99 (2): 133–57. PMID 20711675. doi:10.1007/s10482-010-9488-3.
- ↑ Naushad, Hafiz Sohail; Gupta, Radhey S. (2011). "Molecular signatures (conserved indels) in protein sequences that are specific for the order Pasteurellales and distinguish two of its main clades". Antonie van Leeuwenhoek 101 (1): 105–24. PMID 21830122. doi:10.1007/s10482-011-9628-4.
- ↑ 18,0 18,1 Gao, B.; Mohan, R.; Gupta, R. S. (2009). "Phylogenomics and protein signatures elucidating the evolutionary relationships among the Gammaproteobacteria". International Journal of Systematic and Evolutionary Microbiology 59 (2). PMID 19196760. doi:10.1099/ijs.0.002741-0.
