Vector de clonación
Un vector de clonación é un pequeno fragmento de ADN, tomado dun virus, un plásmido ou unha célula dun orgnismo superior, que pode ser mantido establemente nun organismo, no cal se pode inserir un fragmento de ADN alleo co obxectivo de clonalo.[1] O vector presenta características que permiten unha axeitada inserción no vector dun fragmento de ADN ou a súa eliminación, por exemplo tratando o vector e o ADN alleo cun encima de restrición que corta o ADN. Os fragmentos de ADN así xerados conteñen extremos romos ou con extemos coherentes ou "adhesivos" que sobresaen, e despois o ADN do vector e o ADN alleo con extremos compatibles poden volver a unirse por ligación. Unha vez que o fragmento de ADN foi clonado nun vector de clonación, pode ser despois subclonado noutro vector deseñado para un uso máis específico.
Hai moitos tipos de vectores de clonación, pero os utilizados máis frecuentemente son os plásmidos modificados por enxeñaría. A clonación xeralmente realízase utilizando a bacteria Escherichia coli, e os vectores de clonación en E. coli poden ser plásmidos, bacteriófagos (como o fago λ), cósmidos e cromosomas artificiais bacterianos (BAC). Porén, algúns ADN non poden manterse establemente en E. coli, como ocorre cos fragmentos de ADN moi longos, e nese caso poden usarse outros organismos como os lévedos. Os vectores de clonación en lévedos adoitan ser cromosomas artificiais de lévedos (YAC).
Características dun vector de clonación[editar | editar a fonte]
Todos os vectores comunmente usados en bioloxía molecular teñen unha serie de características claves necesarias para realizar a súa función, como un sitio de clonación adecuado e un marcador seleccionable. Outros poden ter características adicionais para o uso que se lle vai dar. Por razóns de facilidade e conveniencia a clonación adoita facerse con E. coli. Así, os vectores de clonación adoitan ter elementos necesarios para a súa propagación e mantemento en E. coli, como unha orixe de replicación (ori) adicional. A orixe de replicación ColE1 encóntrase en moitos plásmidos. Algúns vectores tamén inclúen elementos que lles permiten manterse noutro organismo ademais de en E. coli, e estes vectores denomínanse vectores lanzadeira.
Sitio de clonación[editar | editar a fonte]
Todos os vectores de clonación teñen características que permiten que se poida inserir convenientemente un xene no vector ou se poida eliminar del. Esta característica pode ser un sitio de clonación múltiple (MCS) ou polilinker, que contén moitos sitios de restrición. Os sitios de restrición no sitio de clonación múltiple son primeiramente clivados por encimas de restrición, despois lígase ao vector un xene diana amplificado por PCR que tamén foi dixerido co mesmo encima usando unha ADN ligase. A secuencia de ADN diana pode inserirse no vector nunha dirección específica se se desexa. Os sitios de restrición pode ser tamén usado para a subclonación noutro vector se é necesario.[2]
Outros vectores de clonación pode usarse unha topoisomerase en vez da ligase e pode facerse a clonación máis rapidamente sen necesidade de facer unha dixestion do vector ou inserto con encimas de restrición. Neste método chamado clonación TOPO un vector linearizado é activado unindo unha topoisomerase I aos seus extremos, e este vector "activado por TOPO" pode despois aceptar un produto de PCR ligando os extremos 5' do produto de PCR, liberando a topoisomerase e formando un vector circular no proceso.[3] Outro método de clonación que non usa a dixestión do ADN e a ligase realízase por recombinación do ADN, por exemplo como a usada no sistema de clonación Gateway.[4][5] O xene, unha vez clonado no vector de clonación (chamado clon de entrada neste método), pode ser convenientemente introducido en diversos vectores de expresión por recombinación.[6]
Marcador seleccionable[editar | editar a fonte]
O vector porta un marcador seleccionable para que se poidan seleccionar as células que foron transformadas positivamente. A resistencia a antibióticos é un marcador de uso frecuente, e un exemplo é o xene da beta-lactamase, que confire resistencia ao grupo de antibióticos beta-lactámicos da penicilina, como a ampicilina. Algúns vectores conteñen dous marcadores seleccionables, por exemplo o plásmido pACYC177 ten xenes para a resistencia á ampicilina e á kanamicina.[7] Os vectores lanzadeira designados para que se poidan manter en dous organismos diferentes poden tamén requirir dous marcadores seleccionables, aínda que algúns marcadores selectables como a resistencia ao zeocina e a higromicina B son efectivos en diferentes tipos celulares. Poden usarse tamén marcadores de selección auxotróficos que permiten que un organismo auxotrófico creza nun medio de crecemento mínimo; exemplos deles son LEU2 e URA3, que se utilizan coas correspondentes cepas auxotróficas de lévedos.[8]
Outro tipo de marcador seleccionable permite a selección positiva de plásmidos co xene clonado. Isto pode implicar o uso dun xene letal para a célula hóspede, como o da barnase,[9] Ccda,[10] e as toxinas parD/parE.[11][12] Este funciona normalmente distorsionando ou eliminando o xene letal durante o proceso de clonación, e os clons insatisfactorios, nos que o xene letal aínda permanece intacto matarían as células hóspede, polo que só se seleccionan os clons satisfactorios.
Xene reporteiro[editar | editar a fonte]
Os xenes reporteiro utilízanse nalgúns vectores de clonación para facilitar o cribado dos clons realizados con éxito usando as características destes xenes que permiten identificar doadamente os clons satisfactorios. Tales características presentes nos vectores de clonación poden ter un operón lacZα para a complementación na selección azul/branco, ou o xene marcador ou os xenes reporteiros en marco con e flanqueando o sitio de clonación múltiple para facilitar a produción de proteínas de fusión. Exemplos de elementos para a fusión que se poden utilizar para o cribado son a proteína fluorescente verde (GFP) e a luciferase.
Elementos para a expresión[editar | editar a fonte]
- Artigo principal: Vector de expresión.
Un vector de clonación non necesita conter elementos axeitados para a expresión dun xene diana clonado, como un promotor e un sitio de unión ao ribosoma (RBS), pero moitos contéñenos e poden funcionar como un vector de expresión. O ADN diana pode inserirse nun sitio que está baixo o control dun promotor particular necesario para a expresión dun xene diana no hóspede elixido. Onde está presente o promotor, a expresión do xene está estreitamente controlada e é inducible para que as proteínas só se produzan cando sexa necesario. Algúns promotores comunmente usados son o T7 e o operón lac. Cómpre a presenza dun promotor cando se usan as técnicas de cribado como a selección azul-branco.
Úsanse ás veces vectores de clonación sen promotor e sitio de unión ao ribosoma para a secuencia de ADN clonada, por exemplo cando se clonan xenes cuxos produtos son tóxicos para as células de E. coli. Son tamén innecesarios o promotor e o sitio de unión ao ribosoma para a secuencia de ADN clonado cando se fai primeiro unha biblioteca xenómica ou de ADNc de clons, xa que os xenes clonados son normalmente subclonados nun vector de expresión máis apropiado se a súa expresión é necesaria.
Algúns vectores son deseñados para a transcrición só con proteínas expresadas non heterólogas, por exemplo para a produción de ARNm in vitro. Estes vectores chámanse vectores de transcrición. Poden carecer das secuencias necesarias para a poliadenilación e terminación, polo que non poden ser utilizadas para a produción de proteínas.
Tipos de vectores de clonación[editar | editar a fonte]
Disponse dun gran número de vectores de clonación, e a elección do vector pode depender de diversos factores, como o tamaño do inserto, o número de copias e o método de clonación. Os insertos grandes non poden manterse establemente nun vector de clonación xeral, especialmente para aqueles cun alto número de copias, polo que clonar fragmentos grandes pode implicar o uso dun vector de clonación máis especializado.[13]
Plásmido[editar | editar a fonte]
- Artigo principal: Vector plásmido.
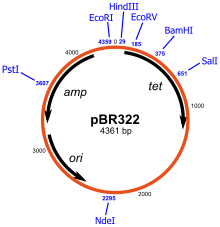
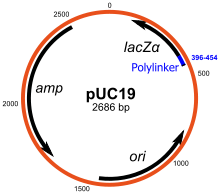
Os plásmidos son moléculas de ADN extracromosómicas circulares que se replican autonomamente. Son os vectores de clonación estándar e os máis usados. Os plásmidos máis xerais poden utilizarse para clonar insertos de ADN de ata 15 kb. Un dos primeiros vectores de clonación que foron comunmente utilizados foi o plásmido pBR322. Outros vectores de clonación inclúen os plásmidos da serie pUC e disponse dun gran número doutros vectores plásmidos de clonación. Moitos plásmidos teñen un alto número de copias, por exemplo o pUC19, que ten un número de copias de 500-700 copias por célula,[14] e ter un número alto de copias é útil xa que produce un maior rendemento de plásmidos recombinantes para a posterior manipulación. Porén os plásmidos de baixo número de copias poden usarse preferiblemente en certas circunstancias, por exemplo, cando as proteínas formadas a partir do xene clonado son tóxicas para a célula.[15]
Algúns plásmidos conteñen unha orixe de replicación de bacteriófago M13 e poden utilizarse para xerar ADN monocatenario. Estes denomínanse faxémidos, e exemplos son a serie pBluescript de vectores de clonación.
Bacteriófago[editar | editar a fonte]
Os bacteriófagos usados para a clonación son o fago λ e o fago M13. Hai unha cantidade máxima de ADN que se pode empaquetar dentro dun fago, que é de 53 kb, polo que para permitir que o ADN alleo se insira dento do ADN do fago, pode ser necesario que nos vectores de clonación fagos se eliminen algúns xenes do fago non esenciais, por exemplo os xenes para a lisoxenia, xa que no uso do fago λ como vector de clonación só está implicadoo ciclo lítico.[16] Hai dous tipos de vectores fago λ: vectores de inserción e vectores de substitución. Os vectores de inserción conteñen un sitio de clivaxe único no que se pode inserir ADN de 5–11 kb. Nos vectores de substitución (replacement vectors) os sitios de clivaxe flanquean unha rexión que contén xenes non esenciais para o ciclo lítico, e esta rexión pode eliminarse e substituírse polo inserto de ADN no proceso de clonación e así pode inserirse un ADN de maior tamaño, de 8–24 kb.[17]
Hai tamén un límite mínimo para o ADN que pode ser empaquetado nun fago e un vector de ADN que é demasiado pequeno non pode ser empaquetado axeitadamente dentro do fago. Esta propiedade pode utilizarse para a selección: un vector sen inserto pode ser demasiado pequeno, polo que só se poden seleccionar para a propagación os vectores con inserto.[18]
Cósmido[editar | editar a fonte]
Os cósmidos son plásmidos que incorporan un segmento de ADN de bacteriófago λ que ten o sitio de extremos cohesivos (cos) que conten elementos necesarios para o empaquetamento do ADN dentro das partículas λ. Utilízanse normalmente para clonar fragmentos de ADN grandes de entre 28 e 45 Kb.[13]
Cromosoma artificial bacteriano[editar | editar a fonte]
Os cromosomas artificiais bacterianos (BAC) teñen un tamaño de inserción de ata 350 kb. Os BAC mantéñense en E. coli cun número de copias de só 1 por célula.[17] Os BAC están baseados no plásmido F, pero hai outro tipo de cromosoma artificial chamado PAC que está baseado no fago P1.
Cromosoma artificial de lévedo[editar | editar a fonte]
Poden introducirse insertos de ata 3 000 kb en cromosomas artificiais de lévedos.
Cromosoma artificial humano[editar | editar a fonte]
Un cromosoma artificial humano pode ser potencialmente útil como vector de transferencia de xenes para entregar xenes en células humanas e unha ferramenta para estudos de expresión e determinar a función do cromosoma humano. Pode portar fragmentos de ADN moi grandes (non hai un límite superior de tamaño a efectos prácticos), por tanto, non existe o problema dunha capacidade de clonación limitada que teñen outros vectores e tamén evita unha posible mutaxénese insercional causada pola integración no cromosoma do hóspede dun vector viral.[19][20]
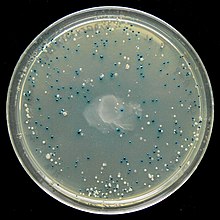
Cribado: exemplo de cribado azul/branco[editar | editar a fonte]
- Artigo principal: Cribado azul/branco.
Moitos vectores usados para propósitos xerais como pUC19 xeralmente inclúen un sistema para detectar a presenza dun fragmento de ADN clonado, baseado na perda dun fenotipo facilmente detectable. O de uso máis xeral é o xene de E. coli que codifica o encima β-galactosidase, cuxa actividade pode ser doadamente detectada pola capacidade deste encima de hidrolizar o substrato soluble incoloro X-gal (5-bromo-4-cloro-3-indolil-beta-d-galactósido) orixinando un produto azul insoluble (5,5'-dibromo-4,4'-dicloro índigo). Clonar un fragmento de ADN dentro da secuencia lacZα baseada no vector da β-galactosidase impide a produción dun encima activo. Se incluímos X-gal nas placas de ágar selectivas, as colonias tansformantes son xeralmne azuis no caso de que se trate dun vector sen ADN inserido e brancos no caso dun vector que conteña un fragmento de ADN clonado; estes últimos son os que interesan.
Notas[editar | editar a fonte]
- ↑ "Definition of cloning vector". Genome Dictionary. Consultado o 2012-10-18.
- ↑ Gorman; et al. (2015). "Adaptability and reproducibility of alternative splice and restriction sites in differential cloning vectors: a study and literature review". Journal of Biomolecular Technology 30 (19): 120–142.
- ↑ "The Technology Behind TOPO® Cloning". Invitrogen.
- ↑ Esposito D, Garvey LA, Chakiath CS (2009). Gateway cloning for protein expression. Methods in Molecular Biology 498. pp. 31–54. ISBN 978-1-58829-879-9. PMID 18988017. doi:10.1007/978-1-59745-196-3_3.
- ↑ "Cloning Methods - Recombination cloning systems". EMBL.
- ↑ "Gateway® Recombination Cloning Technology". Invitrogen.
- ↑ Nicola Casali; Andrew Preston (2003). E. coli plasmid vectors. Methods in Molecular Biology 235. p. 23. ISBN 978-1-58829-151-6.
- ↑ Romanos MA, Scorer CA, Clare JJ (1992). "Foreign gene expression in yeast: a review" (PDF). Yeast 8 (6): 423–88. PMID 1502852. doi:10.1002/yea.320080602.
- ↑ Yazynin SA, Deyev SM, Jucovic M, Hartley RW (1996). "A plasmid vector with positive selection and directional cloning based on a conditionally lethal gene". Gene 169 (1): 131–2. PMID 8635737. doi:10.1016/0378-1119(95)00814-4.
- ↑ Philippe Bernard (1996). "Positive Selection of Recombinant DNA by CcdB" (PDF). BioTechniques 21 (2): 320–323. PMID 8862819. doi:10.2144/96212pf01. Arquivado dende o orixinal (PDF) o 16 de maio de 2017. Consultado o 16 de decembro de 2018.
- ↑ Gabant P, Van Reeth T, Drèze PL, Faelen M, Szpirer C, Szpirer J (2000). "New positive selection system based on the parD (kis/kid) system of the R1 plasmid". BioTechniques 28 (4): 784–8. PMID 10769758.
- ↑ Kim HG, Kim HS, Hwang HJ, Chung SK, Lee JM, Chung DK (2004). "Construction of a pTOC-T vector using GST-ParE toxin for direct cloning and selection of PCR products". Biotechnology Letters 26 (21): 1659–63. PMID 15604816. doi:10.1007/s10529-004-3518-z.
- ↑ 13,0 13,1 Andrew Preston (2003-07-03). E. coli plasmid vectors (PDF). Methods in Molecular Biology 235. pp. 19–20. ISBN 978-1-58829-151-6.
- ↑ Nicola Casali; Andrew Preston (2003). E. Coli Plasmid Vectors: Methods and Applications. Humn Press. ISBN 978-1588291516.
- ↑ "Copy number". Genetics Institute, Inc. Arquivado dende o orixinal o 19 de abril de 2013. Consultado o 16 de decembro de 2018.
- ↑ B. R. Glick; J. J. Pasternak (2005). Molecular Biotechnology Principles and Applications of Recombinant DNA (3rd ed.). ASM Press. ISBN 9781555816124.
- ↑ 17,0 17,1 Andrew Preston (2003-07-03). E. coli plasmid vectors (PDF). Methods in Molecular Biology 235. pp. 21–22. ISBN 978-1-58829-151-6. Arquivado dende o orixinal (PDF) o 19 de febreiro de 2018. Consultado o 16 de decembro de 2018.
- ↑ TA Brown (2010-04-19). Gene Cloning and DNA Analysis: An Introduction. Wiley-Blackwell. p. 100. ISBN 978-1444334074.
- ↑ Kim JH, Kononenko A, Erliandri I, Kim TA, Nakano M, Iida Y, Barrett JC, Oshimura M, Masumoto H, Earnshaw WC, Larionov V, Kouprina N (Dec 13, 2011). "Human artificial chromosome (HAC) vector with a conditional centromere for correction of genetic deficiencies in human cells". Proc Natl Acad Sci U S A 108 (50): 20048–53. PMC 3250132. PMID 22123967. doi:10.1073/pnas.1114483108.
- ↑ Kouprina N, Earnshaw WC, Masumoto H, Larionov V (2013). "A new generation of human artificial chromosomes for functional genomics and gene therapy". Cellular and Molecular Life Sciences 70 (7): 1135–48. PMC 3522797. PMID 22907415. doi:10.1007/s00018-012-1113-3.
