Reacción en cadea da polimerase


A reacción en cadea da polimerase, coñecida como PCR (do inglés polymerase chain reaction), é unha técnica de bioloxía molecular desenvolvida en 1987 por Kary Mullis,[1] que ten como obxectivo obter un gran número de copias dun fragmento de ADN particular, partindo dunha cantidade mínima. En teoría abondaría partir dunha única copia dese fragmento orixinal, ou molde.
Esta técnica serve para amplificar un fragmento de ADN. A súa utilidade é que trala amplificación é moito máis fácil identificar cunha moi alta probabilidade, virus ou bacterias causantes dunha enfermidade, identificar persoas (cadáveres) ou facer investigacións científicas sobre o ADN amplificado. Estes usos derivados da amplificación fixeron que se convertise nunha técnica moi utilizada, o que supuxo o abaratamento do equipo necesario para levala a cabo.
Fundamento e importancia[editar | editar a fonte]
Esta técnica fundaméntase na propiedade natural das ADN polimerases de replicar fibras de ADN, para o cal se empregan ciclos de altas e baixas temperaturas alternadas para separar as dúas cadeas do ADN acabado de formar tras cada fase de replicación e, seguidamente, deixar que as fibras de ADN volvan a unirse para poder duplicalas novamente. A reacción en cadea da polimerase foi perfeccionada por Kary Mullis cando traballaba na Cetus Corporation en California, na década de 1980.
Inicialmente a técnica era lenta, xa que as polimerases se desnaturalizaban ao realizar os cambios de temperatura e era necesario engadir novas polimerases en cada ciclo. Como as temperaturas ás que funcionaba o ciclo (95 °C nas fases de desnaturalización do ADN) supoñen a inmediata desnaturalización das proteínas, empréganse ADN polimerases termoestables, extraídas de microorganismos adaptados a vivir a esas temperaturas, restritivas para a maioría dos seres vivos. Ditos microorganismos, xeralmente arqueas, son: Thermus aquaticus (Taq polimerase), Pyrococcus furiosus (Pfu polimerase), Thermococcus litoralis (Vent) e Thermus thermophilus (Tth). Xeralmente empréganse mesturas de polimerases moi procesivas (Taq) con outras con capacidade de realizar corrección de erros (Pfu, Vent).

Hoxe, todo o proceso da PCR está automatizado nun aparello chamado termociclador, que permite quentar e arrefriar os tubos de reacción para controlar a temperatura necesaria para cada fase da reacción. Moitos termocicladores modernos usan o efecto Peltier, que permite tanto quentar coma arrefriar os tubos simplemente invertendo a corrente eléctrica. Os tubos usados para PCR teñen unha parede moi fina, o que favorece unha boa condutividade térmica, permitindo que se acade rapidamente o equilibrio térmico. Case todos os termocicladores teñen un sistema que quenta a tapa de peche coa finalidade de evitar a condensación sobre os tubos de reacción. Os termocicladores máis antigos carecían deste sistema e solucionaban o problema da condensación cunha capa de aceite na parte superior da mestura de reacción ou cun pouco de cera dentro dos tubos.
En xeral, a PCR é unha técnica común e normalmente indispensable en laboratorios de investigación médica e biolóxica para unha gran variedade de aplicacións. Entre elas inclúense a clonación de ADN para a secuenciación, a filoxenia baseada en ADN, a análise funcional de xenes, o diagnóstico de trastornos hereditarios, a identificación de pegadas xenéticas (usada en técnicas forenses e nas probas de paternidade) e a detección e diagnóstico de enfermidades infecciosas.
Reactivos[editar | editar a fonte]

Para realizar a técnica cómpre o seguinte:[2]
- Os 4 desoxirribonucleósidos-trifosfato (dNTP), substratos para polimerizar o novo ADN.
- Dous cebadores ou iniciadores (en inglés, primers), oligonucleótidos que son complementarios a unha parte das dúas fibras do ADN, un cebador para unha das fibras e o outro para a outra. Son secuencias curtas, de entre seis e corenta nucleótidos, normalmente de dezaoito a vinte e dous, que permiten que a polimerase inicie a reacción. Deben estar enfrontados e a non moita distancia. Delimitan a zona de ADN a amplificar, é dicir, corresponden aos nucleótidos que definen os extremos da secuencia que se desexa replicar.
- Ións divalentes. Adoita usarse o magnesio (Mg2+), engadido xeralmente como cloruro de magnesio (MgCl2), ou algún outro catión divalente. Tamén se pode empregar manganeso (Mn2+), para mutaxénese de ADN mediante PCR, xa que altas concentracións de Mn2+ incrementan a taxa de erro durante a síntese de ADN. Actúan como cofactores da polimerase.
- Ións monovalentes, como o potasio.
- Unha solución tampón ou buffer que mantén o pH axeitado para o funcionamento da ADN polimerase.
- ADN polimerase ou mestura de distintas polimerases con temperatura óptima arredor de 70 °C (a máis común é a Taq polimerase).
- ADN molde, que contén a rexión de ADN que se vai amplificar.
- Termociclador, o aparello que mantén a temperatura necesaria en cada unha das fases que forman un ciclo.
Conceptos utilizados na PCR[editar | editar a fonte]
- Sensibilidade: refírese á cantidade mínima de ADN necesaria para que se produza a amplificación, é dicir, para obter unha banda. Relaciónase cos falsos negativos, xa que pode que unha mostra sexa en realidade positiva pero o resultado dea negativo porque non se amplificou por non ter unha cantidade de ADN dabondo.
- Especificidade: refírese á obtención dun só produto amplificado. Está determinada polos oligos e a especificidade con que se unen ao ADN molde. Desta forma, se os oligos teñen máis dun sitio ao que se poden unir aparecerá máis dun produto amplificado.
- Eficiencia: refírese á amplificación máxima que se pode obter nun número determinado de ciclos.
- Fidelidade: refírese aos erros que comete a ADN polimerase durante a amplificación. Este concepto é de especial importancia na secuenciación, pero noutros casos non é tan importante. Unha boa fidelidade permite evitar falsos positivos e falsos negativos.
Ciclo de amplificación[editar | editar a fonte]


O proceso da PCR polo xeral consiste en realizar unha serie de 20 a 35 cambios repetidos de temperatura chamados ciclos; cada ciclo adoita constar de 2-3 pasos a diferentes temperaturas. A PCR común realízase con ciclos que teñen tres pasos de temperatura. Os pasos de ciclo a miúdo están precedidos por un choque térmico (chamado "hold") a alta temperatura (> 90 °C), e seguido por outro ao final do proceso para a extensión do produto final ou o breve almacenamento. As temperaturas usadas e o tempo aplicado en cada ciclo dependen de moitos factores. Estes inclúen o encima usado para a síntese de ADN, a concentración de ións divalentes e dos dNTP na reacción, e a temperatura de unión dos cebadores, e a lonxitude do ADN que se desexa amplificar.[2]
Actualmente, case todos os termocicladores dan a opción de realizar a reacción da PCR coa chamada " tapa quente". Isto consiste en que o sistema do termociclador aplicará calor á parte de arriba do vial que contén a mestura de PCR. Ao comezo, os laboratorios que empezaron a usar os primeiros aparellos que se comercializaron e que non incluían este sistema tiñan que poñer unhas pingas de aceite dentro do vial. O obxectivo deste procedemento, igual que o da tapa quente, é evitar a condensación da mostra, xa que no tubo Eppendorf encóntranse dúas fases: líquido e gas. Ao condensar a mostra, pérdese volume da mestura. Porén, quentando a tapa ou poñendo as pingas de aceite evítase este proceso físico, conservando case intacto o volume da mostra.
Inicio[editar | editar a fonte]
Este paso consiste en levar a reacción ata unha temperatura de 94-96 °C (ou 98 °C se se está usando unha polimerase termoestable extrema), que se mantén durante 1-9 minutos. Isto só é necesario para ADN polimerases que requiran activación por calor.
Desnaturalización[editar | editar a fonte]
En primeiro lugar, desnaturalízase o ADN (sepáranse as dúas cadeas das que está formado). Este paso pode realizarse de diferentes xeitos, pero o máis habitual é o quentamento (a 94-95 °C) da mostra. A temperatura á cal se decide realizar a desnaturalización depende, por exemplo, da proporción de G+C que teña a cadea, e tamén da lonxitude da mesma. Outros métodos, raramente empregados na técnica da PCR, serían a adición de sales ou axentes químicos capaces de realizar a desnaturalización.
Aliñamento ou unión do cebador[editar | editar a fonte]
A continuación prodúcese a hibridación do cebador, é dicir, o cebador únese á súa secuencia complementaria no ADN molde. Isto chámase annealing. Para que se produza cómpre baixar a temperatura a 40-68 °C durante 20-40 segundos (dependendo do caso). As pontes de hidróxeno estables entre as cadeas de ADN (unión ADN-ADN) só se forman cando a secuencia do cebador é moi similar á secuencia do ADN molde. A polimerase únese ao híbrido entre a cadea molde e o cebador, e empeza a sintetizar ADN. Os cebadores actuarán como límites da rexión da molécula que vai ser amplificada.
Extensión ou elongación da cadea[editar | editar a fonte]
Actúa agora a ADN polimerase, tomando o ADN molde para sintetizar a cadea complementaria e partindo do cebador como soporte inicial necesario para a síntese de novo ADN. A polimerase sintetiza unha nova fibra de ADN complementaria á fibra molde engadindo os dNTP complementarios en dirección 5'→ 3', unindo o grupo 5'-fosfato dos dNTP co grupo 3'-hidroxilo do final da fibra de ADN en crecemento (a cal se estende). A temperatura para este paso depende da ADN polimerase que usemos. Para a Taq polimerase, a temperatura de máxima actividade está en 75-80 °C (normalmente 72 °C). O tempo de extensión depende tanto da ADN polimerase usada coma da lonxitude do fragmento de ADN que se vai amplificar. Hai unha regra comunmente usada: na súa temperatura óptima, a polimerase de ADN polimerizará mil bases por minuto.
Elongación final[editar | editar a fonte]
Fase única que se leva a cabo a unha temperatura de 70-74 °C durante 5-15 minutos tralo último ciclo de PCR. Con ela asegúrase que calquera ADN de cadea simple restante sexa totalmente ampliado.
Conservación[editar | editar a fonte]
Este é un paso que se realiza a 4-15 °C durante un tempo indefinido para conservar a reacción a curto prazo.
A PCR realízase normalmente cun volume de reacción de 15-100 μL, en pequenos tubos de 0,2-0,5 mL que se colocan no termociclador.
Para verificar que a PCR xerou o fragmento de ADN previsto, empréganse técnicas de electroforese, que separan os fragmentos de ADN xerados de acordo á súa carga, isto é, lonxitude, e, en menor medida e dependendo da matriz empregada, ao seu tamaño: tipicamente emprégase a electroforesis en xel de agarosa, para fragmentos grandes; en acrilamida, para os máis pequenos; e, de forma máis rápida e aplicable á PCR asociada a marcaxe fluorescente, a electroforese capilar.[3] O(s) tamaño(s) do(s) produto(s) da PCR está(n) determinados por un marcador de masa molecular de ADN, o cal contén fragmentos de ADN de tamaño coñecido, e que se fai correr no xel xunto cos produtos de PCR.
Optimización da PCR[editar | editar a fonte]
Na práctica, a PCR pode fallar por varias razóns, entre elas están:
- A PCR é unha técnica de gran sensibilidade, é dicir, necesita só unha mínima cantidade de ADN para obter un gran número de copias. Ademais, pode ser moi propensa a erros se se realiza en condicións inadecuadas de esterilidade, que conduzan á amplificación de ADN non correspondente á mostra a analizar (e, por tanto, a conclusións incertas). Deben tomarse unha serie de precaucións para evitar a contaminación con ADN estraño. Exemplos nos que é especialmente importante evitar a amplificación son a detección de enfermidades (para non diagnosticar erradamente aos pacientes) ou o diagnóstico de identidade e parentesco. Posibles precaucións a tomar son:
- Ter especial coidado durante os pasos previos á amplificación: recollida, envío, custodia e procesado de mostras.
- Limpeza exhaustiva e esterilización (lixivia, etanol, luz ultravioleta, psoralenos...) da superficie de traballo entre a realización dunha PCR e a seguinte.
- En laboratorios de diagnóstico xenético adoita haber áreas separadas de traballo: un laboratorio para a preparación da mestura nai para a PCR, outro para a adición do ADN a amplificar, outro onde se encontra a maquinaria para realizar a PCR e outro onde se abre e analiza a mostra xa amplificada.
- Cabinas de seguridade biolóxica para reducir vapores cargados de amplicóns de enfermidades.
- Protección adecuada dos operarios que manipulen as mostras e os instrumentos do laboratorio: luvas, bata, cubertas de zapatos, lentes de seguridade, pelo recollido... No diagnóstico molecular é conveniente que se cambien estes elementos ao pasar dunha zona de traballo a outra.
- Controis positivos e negativos.
- Tipificación xenética de todo o persoal do laboratorio. Así, en caso de atopar unha amplificación non agardada poderase comprobar se provén dun dos operarios.
- As técnicas de deseño de cebadores son importantes na mellora da obtención de produtos de PCR e para evitar a formación de produtos falsos. Algunhas consideracións ao deseñar estes cebadores son:
- Recoméndase usa cebadores de máis de 15 nucleótidos (18-30 nn) Normalmente, adoitan deseñarse de 20 nucleótidos. Os cebadores moi curtos fan que a PCR non sexa moi específica, e os que se excedan en lonxitude farán que se perda rendemento na reacción.
- Os cebadores ou primers non deben diferir en máis de 3 bases.
- A proporción entre bases nitroxenadas púricas e pirimidínicas dos dous oligos debería ser 1:1 (40-60% como moito), e que empecen e rematen cunha ou dúas bases púricas.
- Requírese unha distribución homoxénea dos catro nucleótidos na secuencia, evitando os poliT/A/G/C.
- A Tm (temperatura de fusión) dos oligos non pode diferir en máis de 5 °C.
- Os cebadores non deben incluír na súa secuencia rexións que sexan complementarias entre si, ou poderán formarse dímeros entre eles.
- Existe unha gran variedade de polimerases dispoñibles, e pódese elixir a que máis se axeite ás nosas necesidades. Por exemplo, algunhas teñen actividades inexistentes noutras, son máis eficientes ou funcionan en intervalos de temperatura nos cales outras se desnaturalizan ou perden funcionalidade. As máis habituais son a Taq e a Pfu.
- Os compoñentes do tampón de reacción deberán axustarse ás esixencias dos encimas utilizados.
- O termociclador debe poder reproducir correctamente os ciclos da PCR: para iso, diversas casas comerciais ofrecen termocicladores elaborados con materiais que fagan máis eficaces o desenvolvemento e o transcurso das distintas fases do proceso, ademais de diversas facilidades de utilización. Dentro do laboratorio, o seu uso, mantemento e situación son fundamentais.
Á parte de aspectos como a contaminación e algún fallo na hibridación de cebadores, pode haber outras complicacións que afecten á PCR, como son:
- Degradación do ADN: isto pode ocorrer cando se manipula a mostra en condicións subóptimas ou cando se fai unha autopsia, por exemplo, e dá lugar a unha ausencia de amplificación ou resultados parciais. Un caso particular é o "dropout" (~perda) de alelos, que se pode chegar a confundir con ser homocigoto para dito alelo.
- Inhibición da PCR: pode haber axentes que interfiran no correcto transcurso da PCR. Isto requirirá un procesamento adicional da mostra para eliminar estes compostos do medio antes de preparar a mestura de PCR. O sangue e o seme conteñen este tipo de inhibidores, polo que é necesario unha purificación previa.
- Modificación do ADN: as substancias como o formol (empregado en conservación de tecidos) ou a luz ultravioleta modifican o ADN, alterando así os resultados da amplificación.
- Artefactos: poden darse situacións como as bandas "stutter" (tatexo) ou "shadow" (sombra), que consisten en que a polimerase amplifica unha repetición de máis dun microsatélite ou repetición en tándem.
- Alelo fóra de patrón: un alelo amplificado pode non coincidir co patrón de picos da referencia. Isto é sinal de que algo foi mal durante a PCR.
- Mesturas: é posible que dúas mostras se mesturen dalgunha forma e o resultado sexa unha combinación do patrón de picos que cabe esperar trala amplificación de cada unha delas por separado. Isto e o caso anterior dificultan a interpretación dos resultados.
Tipos de PCR[editar | editar a fonte]
PCR aniñada[editar | editar a fonte]
Técnica moi sensible de PCR na que o produto dunha amplificación utilízase como molde para realizar unha segunda amplificación con cebadores que se sitúan dentro (están aniñados) da primeira secuencia amplificada, é dicir, cando se ten o primeiro amplicón poden unirse os cebadores e faise de novo unha amplificación dentro do amplicón inicial. Este tipo de PCR ten a vantaxe de ofrecer alta sensibilidade e especificidade. A especificidade aumenta porque como se trata da amplificación dun amplicón obtido previamente, os cebadores só van hibridar nun sitio dentro da molécula e o resultado será unha única banda. Así, evítanse posibles hibridacións inespecíficas dos cebadores. A desvantaxe desta técnica é que non permite cuantificar a mostra.
PCR de extensión solapada (mutaxénese)[editar | editar a fonte]
Introdúcense cambios de secuencia dentro de fragmentos (clonados) de ADN. Requírense dous cebadores mutaxénicos e outros dous. Amplifícase un fragmento 5' e un fragmento 3' que se solapan e levan ambos os dous a mutación. Úsanse os produtos noutra reacción para producir o ADN mutado de lonxitude completa.
PCR in situ[editar | editar a fonte]
A PCR in situ consiste nunha reacción de PCR en cortes histolóxicos ou células, onde os produtos xerados poden visualizarse no sitio de amplificación. Realízase sobre preparacións fixas nun portaobxectos. Na técnica da PCR in situ hai que realizar unha primeira amplificación do ADN diana e logo a detección mediante hibridación in situ convencional con sondas de ADN/ARN. Desta maneira poden detectarse cantidades pequenísimas de xenoma. Esta tecnoloxía é de gran capacidade para amplificar especificamente unha poboación de secuencias de menor representación.
PCR múltiple[editar | editar a fonte]
É unha PCR na cal se amplifica simultaneamente máis dunha secuencia.Para facelo hai que combinar dous ou máis pares de cebadores nun mesmo tubo, xunto co resto dos reactivos da reacción en cantidades suficientes, para amplificar simultaneamente varios segmentos de ADN. Vantaxes: información sobre varios locus nunha soa reacción, menor cantidade de molde para a análise, menor cantidade de reactivos, rápida construción de bases de datos. Desvantaxes: para levala a cabo axeitadamente e sen erros requírese unha coidadosa optimización do proceso.
PCR con transcriptase inversa (RT-PCR)[editar | editar a fonte]
- Artigo principal: PCR con transcriptase inversa.
É unha variante da PCR na que se usa ARN como molde inicial en vez de ADN, e emprega unha transcriptase inversa (como Tth) para realizar a síntese dun ADN complementario do ARN (ADNc). Deste xeito, o desenvolvemento inicial dunha RT-PCR sería:
- 1º paso: retrotranscrición a partir do ARN.
- 2º paso: amplificación a partir da primeira fibra de ADNc.
- 3º paso: PCR estándar.
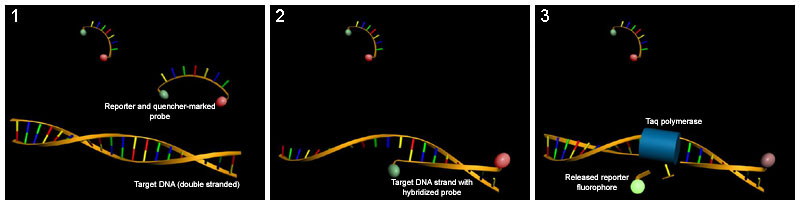
PCR en tempo real ou PCR cuantitativa (qPCR)[editar | editar a fonte]
- Artigo principal: PCR en tempo real.
Reacción de PCR que se caracteriza principalmente porque permite cuantificar a cantidade de ADN ou ARN presente na mostra orixinal, ou para identificar cunha moi alta probabilidade, mostras de ADN específicas a partir da súa temperatura de fusión (Tm).
Pódese dividir nas técnicas baseadas en fluorocromos non específicos e nas baseadas en sondas específicas.
Nas técnicas baseadas en fluorocromos o ADN, que multiplica a súa cantidade con cada ciclo, únese ao fluorocromo (xeralmente SYBR Green) producindo fluorescencia que se mide polo termociclador apto para PCR en tempo real. Permite cuantificar só unha secuencia por reacción pero ten a vantaxe de utilizar cebadores normais para a súa realización. É moito máis económica que a que usa sondas específicas.
As técnicas baseadas en sondas específicas utilizan unha sonda unida a dous fluorocromos que hibrida na zona intermedia entre o cebador directo (forward) e o inverso (reverse); cando a sonda está intacta, presenta unha transferencia enerxética de fluorescencia por resonancia (FRET). Dita FRET non se produce cando a sonda está danada e os dous fluorocromos están afastados, produto da actividade exonuclease 5'-3' da ADN polimerase. Isto permite monitorizar o cambio do patrón de fluorescencia e deducir o nivel de amplificación do xene.
A maioría destes inconvenientes solucionáronse coa introdución da PCR realizada en tempo real (qPCR), que elimina calquera proceso post-PCR, xa que monitoriza a progresión da amplificación no momento en que ocorre. A diferenza da PCR convencional (en punto final), que mide a acumulación do ADN ao final dun número predeterminado de ciclos, con qPCR isto faise durante o proceso de amplificación usando fluorescencia, de forma que o seu aumento é proporcional á cantidade de ADN formada. O proceso pódese automatizar doadamente usando un sistema que realice a amplificación (termociclador) e que á súa vez poida ler a fluorescencia. Existe unha ampla oferta de aparellos no mercado. A maioría poden traballar coas diversas opcións de marcado fluorescente e son "abertos", é dicir, permiten programar as condicións de amplificación e lectura de forma que o seu uso non queda limitado a uns reactivos determinados.
Outras variacións da PCR básica[editar | editar a fonte]
Ademais das técnicas de PCR básicas mencionadas existen tamén determinadas variacións, que son útiles para conseguir certos obxectivos. Son as seguintes:
- PCR específica de alelo: esta técnica de diagnóstico ou clonación utilízase para identificar ou utilizar os polimorfismos dunha soa base (SNPs).[4]
- PCR "assembly" (de ensamblaxe): consiste na síntese artificial de longas secuencias de ADN, realizando a PCR nun fondo de oligonucleótidos longos con secuencias solapantes curtas.[5]
- PCR asimétrica: usada para amplificar preferentemente unha cadea do ADN orixinal con respecto á outra.[6]
- PCR de colonias: mediante esta técnica, poden examinarse rapidamente colonias da bacteria Escherichia coli para construcións viables de vectores de ADN.
- Amplificación dependente de helicase: esta técnica é moi parecida á PCR convencional, pero nela emprégase o encima helicase e unha temperatura constante en lugar da ADN polimerase e os ciclos repetidos de hibridación-elongación..[7]
- PCR hot-start (inicio quente): esta técnica reduce a amplificación inespecífica durante as fases iniciais da PCR: mentres que a máquina atinxe a temperatura da primeira fase (uns 95 °C) pode que se produza a unión dos cebadores e haxa amplificación, xa que no camiño para chegar a eses 95º se pasa pola temperatura de annealing, que é máis baixa..[8][9][10]
- Para evitar isto, a PCR hot-start baséase en que a reacción comece cando a máquina xa estea a 95º, debido a que antes non están presentes a polimerase ou o cloruro de magnesio, o que se pode conseguir por medio de varias técnicas:
- - Engadir a polimerase ou o cloruro de magnesio tralo período de quentamento.
- - Separar por medio dunha capa de cera os distintos compoñentes da reacción. A cera funde ao acadar os 95º e é entón cando os compoñentes entran en contacto.
- - Anticorpos anti-polimerase que bloqueen a polimerase. Ao chegar aos 95º estes anticorpos inactívanse debido a que se desnaturalizan e a polimerase pode actuar.
- PCR específica de intersecuencia (ISSR): trátase dun método de PCR para o seu uso en pegada xenética, que amplifica rexións entre repeticións de secuencia simple para producir unha pegada xenética única de lonxitudes de fragmento amplificadas.[11]
- PCR inversa: é un método usado para poder realizar a PCR cando só se coñece unha secuencia interna. Moi útil na identificación de secuencias que flanquean insercións xenómicas.[12]
- PCR mediada por ligazón: este método usa pequenos linkers de ADN ligados ao ADN de interese e múltiples cebadores que hibridan eses linkers.[13]
- PCR específica de metilación (MSP): úsase para detectar metilacións en illas CpG do ADN xenómico.[14]
- Amplificación de sonda dependente de ligazón múltiple (Multiplex Ligation-dependent Probe Amplification ou MLPA): permite amplificar varias secuencias obxectivo cun único par de cebadores, evitando así as limitacións de resolución da PCR múltiple (multiplex).
- PCR cuantitativa: a PCR cuantitativa úsase para medir a cantidade dun produto de PCR. Na técnica clásica de PCR cuantifícase por aproximación con dilucións e amplificación de concentracións coñecidas da secuencia diana. Tamén se pode cuantificar por método competitivo: trabállase con concentracións crecentes coñecidas dun fragmento que pode ser amplificado polos mesmos oligos que a mostra de estudo, pero de menor tamaño que esta, de forma que cos resultados obtidos pode estimarse a concentración do competidor que é equivalente á da mostra de cantidade descoñecida. A cuantificación en PCR está optimizada na técnica de PCR en tempo real.
- PCR-TAIL: a PCR termal de entrelazado asimétrico úsase para illar unha secuencia descoñecida que está situada flanqueando unha secuencia coñecida.[15]
- PCR touchdown: trátase dunha variante da PCR que se emprega cando se descoñece a secuencia exacta dos extremos da secuencia a amplificar, de modo que se asume que pode existir algunha base desapareada no aliñamento cebador-secuencia. A súa finalidade é reducir o fondo non específico baixando gradualmente a temperatura de hibridación ao longo do progreso da PCR.[16]
- PAN-AC: este método usa condicións isotermas para a amplificación, e pode utilizarse en células vivas..[17][18]
- PCR dial-out: un método en paralelo para obter moléculas de ADN axeitadas para a síntese de xenes. Unha libraría complexa de moléculas de ADN modifícase con etiquetas de flanqueo únicas antes de facer unha secuenciación masivamente paralela. Os cebadores dirixidos ás etiquetas despois permiten a obtención de moléculas coas secuencias desexadas por PCR.[19]
- PCR minicebador (miniprimer): usa unha polimerase termoestable (S-Tbr) que pode facer a extensión desde curtos cebadores ("smalligos") de só 9 ou 10 nucleótidos. Este método permite que a PCR se centre en rexións de unión ao cebador máis pequenas, e utilízase para amplificar secuencias de ADN conservadas, como as dos xenes de ARNr 16S procarióticos (ou os de 18S eucarióticos).[20]
- PCR dixital (dPCR): utilizada para medir a cantidade dunha secuencia de ADN diana nunha mostra. A mostra de ADN dilúese moito para que despois execute moitas reaccións de PCR en paralelo, algunhas delas non reciben unha soa molécula do ADN diana. A concentración de ADN diana calcúlase usando a proporción de resultados negativos. De aí o nome de "PCR dixital".
- PCR en fase sólida: este termo ten varios significados, como amplificación Polony (na que as colonias de PCR se derivan nunha matriz de xel, por exemplo), PCR Bridge[21] (os cebadores son unidos covalentemente a unha superficie sólida), PCR de fase sólida convencional (na que se aplica PCR asimétrica en presenza dun soporte sólido que leva o cebador con secuencia complementaria a un dos cebadores acuosos) e a PCR de fase sólida amplificada[22] (na que a PCR de fase sólida convencional pode mellorarse empregando altas temperaturas de fusión (Tm) e un cebador de soporte sólido aniñado con aplicación opcional dun paso térmico para favorecer o cebado do soporte sólido).
- Fast Walking Universal: para a técnica do genome walking e pegada dactilar xenética[23][24][25][26]
- Ciclo térmico rápido para PCR: [27] A amplificación do ADN pode realizarse rapidamente, e completar 30 ciclos en menos de 30 minutos: ciclo térmico rápido. Normalmente considerábase que as fases de cada ciclo eran tres reaccións que ocorren en tres períodos separados e a tres temperaturas diferentes. Este paradigma do equilibrio secuencial non ten en conta que a temperatura da mostra non cambia instantaneamente, de feito, durante unha PCR a mostra está a maioría do tempo en temperaturas de transición. O ciclo térmico rápido aproveita a vantaxe da instantánea desnaturalización e hibridación, a cuxas temperaturas se debe chegar, pero non teñen que manterse. Ademais, para produtos curtos, a extensión pode levarse a cabo durante a transición á temperatura de extensión, e, xa que logo, tampouco é preciso mantela. Un ciclo rápido podería entón describirse como 94 °C, 0 seg; 55 °C, 0 seg e 72 °C, 0 seg. Este modelo non axuda, porque leva só a temperaturas extremas e non dá información sobre o que pasa entre elas. Polo contrario, no paradigma cinético para PCR de ciclo rápido descríbese completamente o historial de temperaturas da mostra. Considérase que a desnaturalización, hibridación e elongación ocorren nun rango de temperaturas e ademais poden solapar temporalmente. Hai termocicladores que permiten que un ciclo se complete en 20-60 segundos, polo que 30 ciclos tardan de 10 a 30 minutos.
- PCR in silico (en sílice): Non é unha PCR química senón as ferramentas computacionais utilizadas para calcular resultados de PCR teóricas utilizando un conxunto de cebadores dado (sondas para amplificar secuencias de ADN a partir dun xenoma secuenciado ou dun transcriptoma). Tamén se chama PCR dixital, virtual, electrónica, ou e-PCR.
Aplicacións[editar | editar a fonte]
A técnica da PCR ten multitude de aplicacións en ciencia básica como ferramenta de detección e/ou xeración de conxuntos de fragmentos de ADN de interese, e en ciencia aplicada, por exemplo en diagnóstico clínico.
Investigación[editar | editar a fonte]
A PCR convencional, emprégase como base para multitude de técnicas no laboratorio debido á súa robustez e rapidez. Deste modo, a PCR de punto final permite controlar e detectar os fragmentos de ADN de interese.

Unha aplicación da PCR de grande importancia é a clonación de secuencias de ADN en vectores, como poden ser os plásmidos. Para iso, empréganse cebadores que conteñen no seu extremo 5' unha curta secuencia que permite a interacción posterior con outra complementaria situada no vector de clonación a empregar. Por exemplo, pódese incluír unha diana de restrición en ditos cebadores, de modo que, e se esta non existía previamente no fragmento e é única no vector, poida efectuarse unha ligazón mediante a ADN ligase do fago T4 trala dixestión co encima de restrición apropiado de ambos os elementos. Outro método asimilable a esta vía é o emprego da recombinación dirixida; isto é, adáptase ao 5' dos cebadores unha secuencia que faculta a unha recombinase a recombinación dirixida cun vector dado.[28]
Medicina[editar | editar a fonte]
En medicina, a PCR utilízase fundamentalmente como ferramenta de diagnóstico:[29]
- Permite xenotipificar a especie ou especies que provocan un determinado cadro infeccioso: para iso, amplifícase unha zona do xenoma bacteriano cuxo produto de PCR posúa unhas características de tamaño ou temperatura de fusión que permitan identificalo de forma inequívoca. No caso de infeccións virais que implican a integración do xenoma do patóxeno no ADN do hospedador, como é o da infección por VIH, a PCR cuantitativa posibilita a determinación da carga viral existente e, por tanto, do estado da doenza.[30]
- A PCR tamén se pode usar en revisións médicas rutineiras, como nos servizos de doazón de sangue, para probas de rutina. por medio desta técnica poden detectarse infeccións perigosas no doante (como o VIH ou hepatite B) mentres aínda están no período de incubación. Dada a sensibilidade das probas de PCR pódense tomar mostras colectivas ou "pools" (por exemplo, 96 probas individuais). Se unha destas mostras colectivas dá positivo, tómanse a partir dela mostras progresivamente menores ata que se encontra a causante do positivo.
- O diagnóstico de enfermidades hereditarias presentes no xenoma é un proceso longo e complicado que pode acelerarse significativamente grazas á PCR. Cada un dos xenes proba poden amplificarse por medio dos seus correspondentes cebadores e posteriormente secuenciar para detectar a existencia de mutacións.
Paleontoloxía, antropoloxía biolóxica, e ciencias forenses[editar | editar a fonte]
Os eidos da paleontoloxía, antropoloxía biolóxica e a medicina e antropoloxía forense víronse enormemente beneficiados por esta técnica, xa que todas elas obteñen moitos datos dos restos ou pegadas dos seres vivos. Un dos materiais biolóxicos que máis información pode proporcionar é o ADN.
A relativa estabilidade deste permite que, aínda estando fragmentado, se conserve durante longos períodos se as condicións son axeitadas.[28] En certas ocasións as mostras intactas coas que se pode contar son extraordinariamente pequenas ou están deterioradas. A PCR soluciona ambos os problemas e fornece cantidades útiles para posteriores pasos de análise. En primeiro lugar aumenta a cantidade de material recuperado a partir de mostras escasas. Tamén debido á natureza da técnica e o seu propósito de amplificación de fragmentos pequenos, esta fragmentación non impide que este ADN poida ser empregado como molde para unha reacción de PCR.
- En paleontoloxía e antropoloxía a PCR permite recuperar as escasas cantidades de ADN que aínda non se degradaron. Algúns lugares nos que o ADN podería preservarse son a brea, as cinzas volcánicas, o ámbar, xeos históricos polares ou glaciarios, ambientes áridos, sedimentos, e nos cristais de apatita de restos de esqueleto,[31] polo que é posible caracterizar cadáveres, fósiles ou outros restos mediante a xenotipificación por análise de microsatélites ou mesmo de xenomas de taxons extinguidos, amplificados deste modo, como poden ser os realizados mediante o ADN xenómico do home de Neanderthal.[32] O propósito sería utilizar este ADN amplificado para posteriormente realizar estudos filoxenéticos ou etnográficos ou de poboacións comparando secuencias de ADN, ou o estudo das causas da separación evolutiva de dúas especies.
- Nas ciencias forenses emprégase para establecer a filiación dunha persoa ou para obter probas a partir de mostras mínimas deixadas polo autor dun crime como saliva, seme ou outros restos de tecidos.[33]
Agronomía e diversidade[editar | editar a fonte]
Igual a como se obteñen pegadas xenéticas de individuos concretos por PCR múltiplex no marco da xenética forense, existen métodos baseados na PCR que permiten distinguir entre grupos infraespecíficos de cultivos de interese agronómico; por exemplo, de cultivares.[34] Para iso, empréganse oligonucleótidos dun tamaño o suficientemente pequeno como para que ceben de forma relativamente inespecífica, aínda que sempre de tal forma que produzan un patrón de bandas discreto e interpretable. Así, a pauta obtida trala electroforese dos fragmentos tende a agrupar os individuos de maior semellanza, que posúen un comportamento similar.
Historia[editar | editar a fonte]
En 1971, un artigo publicado por Kleppe et al. en Journal of Molecular Biology describiu por primeira vez un método que usaba encimas para replicar unha secuencia pequena de ADN con cebadores in vitro.[35] Porén, este temperán exemplo do principio básico da PCR non recibiu moita atención, e a invención da reacción en cadea da polimerase en 1983 é xeralmente atribuída a Kary Mullis.[36][37] Mullis gañou o Premio Nobel polo seu traballo na PCR.
Algo moi importante na técnica da PCR é que a ADN polimerase que se use poida soportar as altas temperaturas de >90 °C necesarias para a separación das dúas fibras de ADN da dobre hélice tras cada ciclo de replicación. As ADN polimerases que se utilizaron orixinariamente (ver fragmento de Klenow) para os experimentos in vitro previos á PCR non podían aguantar esas altas temperaturas sen desnaturalizarse, polo que os primeiros procedementos para replicar o ADN eran moi ineficaces, longos e requirían grandes cantidades de ADN polimerase (había que engadir máis en cada ciclo).
O descubrimento en 1976 da Taq polimerase, unha ADN polimerase extraída da bacteria termófila Thermus aquaticus que habita en medios de moi alta temperatura (50-80 °C), eliminou os grandes inconvenientes do método da PCR. Esta ADN polimerase é estable a altas temperaturas, e permanece activa despois da desnaturalización do ADN, eliminando a necesidade de engadir á reacción nova polimerase tras cada ciclo. Este descubrimento permitiu automatizar o proceso, antes tan traballoso, acoplándoo ao uso do termociclador.
Ao mesmo tempo que se desenvolvía a PCR en 1983, Mullis traballaba en Emeryville, California (EE UU), para unha das primeiras empresas biotecnolóxicas, Cetus Corporation, onde era responsable de sintetizar cadeas curtas de ADN.[36][38]
Na revista Scientific American, Mullis resumiu o procedemento: "Comezando cunha única molécula do material xenético ADN, a PCR pode xerar 100.000 millóns de moléculas iguais nunha tarde. A reacción é doada de facer, non require máis que un tubo de ensaio, uns poucos reactivos simples e unha fonte de calor."[39] Foi premiado co Premio Nobel de Química en 1993 pola súa invención, e sete anos despois, el e os seus colegas do Cetus levaron á práctica a súa proposta. Porén, houbo controversias e diferentes versións sobre as contribucións intelectuais e prácticas doutros científicos ao traballo de Mullis, e sobre se el foi o inventor único do principio da PCR.
Guerras de patentes[editar | editar a fonte]
A técnica da PCR patentouna Cetus Corporation, onde Mullis traballaba cando a inventou en 1983. O encima Taq polimerase foi tamén patentado. Tiveron lugar varios preitos relacionados coa técnica, entre eles un preito fracasado iniciado por DuPont. A compañía farmacéutica Hoffmann-La Roche adquiriu os dereitos das patentes en 1992 e actualmente mantén as que aínda están protexidas.[40][41]
Notas[editar | editar a fonte]
- ↑ Bartlett J.M.S., Stirling D. (2003) A Short History of the Polymerase Chain Reaction. In: Methods Mol Biol. 226:3-6,DOI 10.1385/1-59259-384-4:3(en inglés)
- ↑ 2,0 2,1 Joseph Sambrook and David W. Russel (2001). Molecular Cloning: A Laboratory Manual (3rd ed. ed.). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 0-87969-576-5.
- ↑ Mathews, C. K.; Van Holde, K.E et Ahern, K.G (2003). "6". Bioquímica (3 ed.). pp. 204 e ss. ISBN 84-7892-053-2.
- ↑ Newton CR, Graham A, Heptinstall LE, Powell SJ, Summers C, Kalsheker N, Smith JC, and Markham AF (1989). "Analysis of any point mutation in DNA. The amplification refractory mutation system (ARMS)". Nucleic Acids Research 17 (7): 2503–2516. PMC 317639. PMID 2785681. doi:10.1093/nar/17.7.2503.
- ↑ Stemmer WP, Crameri A, Ha KD, Brennan TM, Heyneker HL (1995). "Single-step assembly of a gene and entire plasmid from large numbers of oligodeoxyribonucleotides". Gene 164 (1): 49–53. PMID 7590320. doi:10.1016/0378-1119(95)00511-4.
- ↑ Innis MA, Myambo KB, Gelfand DH, Brow MA. (1988). "DNA sequencing with Thermus aquaticus DNA polymerase and direct sequencing of polymerase chain reaction-amplified DNA". Proc Natl Acad Sci USA 85 (24): 9436–4940. PMC 282767. PMID 3200828. doi:10.1073/pnas.85.24.9436.
- ↑ Vincent,Myriam, Xu, Yan, and Kong, Huimin (2004). "Helicase-dependent isothermal DNA amplification". EMBO Reports 5 (8): 795–800. PMC 1249482. PMID 15247927. doi:10.1038/sj.embor.7400200.
- ↑ Q. Chou, M. Russell, D.E. Birch, J. Raymond and W. Bloch (1992). "Prevention of pre-PCR mis-priming and primer dimerization improves low-copy-number amplifications". Nucleic Acids Research 20 (7): 1717–1723. PMC 312262. PMID 1579465. doi:10.1093/nar/20.7.1717.
- ↑ Sharkey, D. J.; Scalice, E. R.; Christy, K. G.; Atwood, S. M.; Daiss, J. L. (1994). "Antibodies as Thermolabile Switches: High Temperature Triggering for the Polymerase Chain Reaction". Bio/Technology 12 (5): 506–509. doi:10.1038/nbt0594-506.
- ↑ Kellogg D E, Rybalkin I, Chen S, Mukhamedova N, Vlasik T, Siebert P D, Chenchik A. 1994. TaqStart Antibody: hot start PCR facilitated by a neutralizing monoclonal antibody directed against Taq DNA polymerase. BioTechniques 16 (6):1134-1137Kellogg, DE; Rybalkin, I; Chen, S; Mukhamedova, N; Vlasik, T; Siebert, PD; Chenchik, A (1994). "TaqStart Antibody: "hot start" PCR facilitated by a neutralizing monoclonal antibody directed against Taq DNA polymerase". BioTechniques 16 (6): 1134–7. PMID 8074881.
- ↑ E. Zietkiewicz, A. Rafalski, and D. Labuda (1994). "Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification". Genomics 20 (2): 176–83. PMID 8020964. doi:10.1006/geno.1994.1151.
- ↑ Ochman H, Gerber AS, Hartl DL (1988). "Genetic Applications of an Inverse Polymerase Chain Reaction". Genetics 120 (3): 621–623. PMC 1203539. PMID 2852134.
- ↑ Mueller PR, Wold B (1988). "In vivo footprinting of a muscle specific enhancer by ligation mediated PCR". Science 246 (4931): 780–786. PMID 2814500. doi:10.1126/science.2814500.
- ↑ Herman JG, Graff JR, Myöhänen S, Nelkin BD, Baylin SB (1996). "Methylation-specific PCR: a novel PCR assay for methylation status of CpG islands". Proc Natl Acad Sci USA 93 (13): 9821–9826. PMC 38513. PMID 8790415. doi:10.1073/pnas.93.18.9821.
- ↑ Y.G. Liu and R. F. Whittier (1995). "Thermal asymmetric interlaced PCR: automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking". Genomics 25 (3): 674–81. PMID 7759102. doi:10.1016/0888-7543(95)80010-J.
- ↑ Don RH, Cox PT, Wainwright BJ, Baker K, Mattick JS (1991). "'Touchdown' PCR to circumvent spurious priming during gene amplification". Nucl Acids Res 19 (14): 4008. PMC 328507. PMID 1861999. doi:10.1093/nar/19.14.4008.
- ↑ David, F. and Turlotte, E., (1998). "An Isothermal Amplification Method". C.R.Acad. Sci Paris, Life Science 321 (1): 909–914. doi:10.1016/S0764-4469(99)80005-5.
- ↑ Fabrice David (September–October 2002). "Utiliser les propriétés topologiques de l’ADN: une nouvelle arme contre les agents pathogènes" (PDF). Fusion. Arquivado dende o orixinal (PDF) o 28 de novembro de 2007. Consultado o 22 de xullo de 2013.
- ↑ Schwartz JJ, Lee C, Shendure J. (2012). "Accurate gene synthesis with tag-directed retrieval of sequence-verified DNA molecules". Nature Methods 9 (9): 913–915. PMC 3433648. PMID 22886093. doi:10.1038/nmeth.2137.
- ↑ Isenbarger TA, Finney M, Ríos-Velázquez C, Handelsman J, Ruvkun G (2008). "Miniprimer PCR, a New Lens for Viewing the Microbial World". Applied and Environmental Microbiology 74 (3): 840–9. PMC 2227730. PMID 18083877. doi:10.1128/AEM.01933-07.
- ↑ Bing, D. H., C. Boles, F. N. Rehman, M. Audeh, M. Belmarsh, B. Kelley, and C. P. Adams. (1996). "Bridge amplification: a solid phase PCR system for the amplification and detection of allelic differences in single copy genes". Genetic Identity Conference Proceedings, Seventh International Symposium on Human Identification. Arquivado dende o orixinal o 07 de maio de 2001. Consultado o 22 de xullo de 2013.
- ↑ Khan Z, Poetter K, Park DJ (2008). "Enhanced solid phase PCR: mechanisms to increase priming by solid support primers". Analytical Biochemistry 375 (2): 391–393. PMID 18267099. doi:10.1016/j.ab.2008.01.021.
- ↑ Myrick KV, Gelbart WM (2002). "Universal Fast Walking for direct and versatile determination of flanking sequence". Gene 284 (1–2): 125–131. PMID 11891053. doi:10.1016/S0378-1119(02)00384-0.
- ↑ "Park DJ Electronic Journal of Biotechnology (online). 15 August 2005, vol. 8, no. 2". Arquivado dende o orixinal o 16 de maio de 2008. Consultado o 22 de xullo de 2013.
- ↑ Park DJ (2005). "A new 5' terminal murine GAPDH exon identified using 5'RACE LaNe". Molecular Biotechnology 29 (1): 39–46. PMID 15668518. doi:10.1385/MB:29:1:39.
- ↑ Park DJ (2004). "3'RACE LaNe: a simple and rapid fully nested PCR method to determine 3'-terminal cDNA sequence". Biotechniques 36 (4): 586–588, 590. PMID 15088375.
- ↑ PCR Applications. Protocols for functional genomics. Capítulo 14. Edited by Michael A. Innis, David H. Gelfand, John J. Sninsky. ISBN 0-12-372186-5, Academic Press 1999
- ↑ 28,0 28,1 Watson, J, D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M. et Losick, R (2004). Benjamin Cummings, ed. Molecular Biology of the Gene (Fifth edition ed.). San Francisco. ISBN 0-321-22368-3.
- ↑ Coleman, WB e Tsongalis, GJ (2006). Molecular Diagnostics: For the Clinical Laboratorian. páx 47-56 e 65-74. Humana Press. ISBN 1-58829-356-4.
- ↑ Scott L. Butler, Mark S.T. Hansen & Frederic D. Bushman (2007). "A quantitative assay for HIV DNA integration in vivo". Nature Medicine (7): 631–634. doi:10.1038/87979.
- ↑ "Museo Victora de Melbourne sobre a preservación do ADN". Arquivado dende o orixinal o 24 de abril de 2016. Consultado o 16 de xullo de 2013.
- ↑ James P. Noonan,Graham Coop, Sridhar Kudaravalli, Doug Smith, Johannes Krause, Joe Alessi, Feng Chen, Darren Platt, Svante Pääbo, Jonathan K. Pritchard, Edward M. Rubin (17 de noviembre de 2006). "Sequencing and Analysis of Neanderthal Genomic DNA". Nature Medicine 314 (5802): 1113 – 1118. DOI: 10.1126/science.1131412.
- ↑ M., Butler, John. Forensic DNA typing : biology, technology, and genetics of STR markers. Elsevier, Acad. Press. ISBN 0121479528.
- ↑ Hu, Jinguo; Quiros, CarlosF. (1991-12). "Identification of broccoli and cauliflower cultivars with RAPD markers". Plant Cell Reports (en inglés) 10 (10). ISSN 0721-7714. doi:10.1007/BF00234583.
- ↑ Kleppe K, Ohtsuka E, Kleppe R, Molineux I, Khorana HG (1971). "Studies on polynucleotides. XCVI. Repair replications of short synthetic DNA's as catalyzed by DNA polymerases". J. Mol. Biol. 56: 341–361. Arquivado dende o orixinal o 07 de setembro de 2009. Consultado o 16 de xullo de 2013.
- ↑ 36,0 36,1 Mullis, Kary (1998). Dancing Naked in the Mind Field. New York: Pantheon Books. ISBN 0-679-44255-3.
- ↑ Rabinow, Paul (1996). University of Chicago Press, ed. Making PCR: A Story of Biotechnology. Chicago. ISBN 0-226-70146-8.
- ↑ Mullis, Kary (1998). Dancing Naked in the Mind Field. New York: Pantheon Books. p. 18. ISBN 0-679-44255-3.
- ↑ Mullis, Kary (1990). "The unusual origin of the polymerase chain reaction". Scientific American 262 (4): 56–61, 64–5.
- ↑ PR Newswire[Ligazón morta]
- ↑ Consellos sobre como sobrevivir á guerra das patentes de Taq Arquivado 27 de setembro de 2007 en Wayback Machine.: GEN Genetic Engineering News Biobusiness Channel: Artículos. 1 de mayo de 2006 (Vol. 26, No. 9).
Véxase tamén[editar | editar a fonte]
| Commons ten máis contidos multimedia sobre: Reacción en cadea da polimerase |
Outros artigos[editar | editar a fonte]
Bibliografía[editar | editar a fonte]
- Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, Mullis KB, et al.(1988) Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science 239: 487–491
- Griffiths, J.F. A.; et al. (2002). Genética. McGraw-Hill Interamericana. ISBN 84-486-0368-0.
- Coleman, WB e Tsongalis, GJ (2006). Molecular Diagnostics: For the Clinical Laboratorian. Humana Press. ISBN 1-58829-356-4. páxs. 47-56 e 65-74
- Butler, JM (2005). Forensic DNA Typing: Biology, Technology, and Genetics of STR. Academic Press pgs 63-84. ISBN 0-12-147952-8.
Ligazóns externas[editar | editar a fonte]
- Animación PCR
- Artigos sobre PCR
- Nova técnica máis eficiente en PCR
- Detalles da PCR en tempo real://www.gene-quantification.info
- Simulación online de reaccións de PCR con procariotas secuenciados.
- Exercicio online no que se deseñan e simulan experimentos de PCR e PCR-RFLP.
