Operón: Diferenzas entre revisións
| Liña 59: | Liña 59: | ||
Discovered in 1953 by [[Jacques Monod]] and colleagues, the trp operon in ''E. coli'' was the first repressible operon to be discovered. While the lac operon can be activated by a chemical ([[allolactose]]), the tryptophan (Trp) operon is inhibited by a chemical (tryptophan). This operon contains five structural genes: trp E, trp D, trp C, trp B, and trp A, which encodes [[tryptophan synthetase]]. It also contains a promoter which binds to RNA polymerase and an operator which blocks transcription when bound to the protein synthesized by the repressor gene (trp R) that binds to the operator. In the lac operon, lactose binds to the repressor protein and prevents it from repressing gene transcription, while in the trp operon, tryptophan binds to the repressor protein and enables it to repress gene transcription. Also unlike the lac operon, the trp operon contains a leader peptide and an [[attenuator (genetics)|attenuator]] sequence which allows for graded regulation.<ref>{{cite book |author=Cummings MS, Klug WS |title=Concepts of genetics |publisher=Pearson Education |location=Upper Saddle River, NJ |year=2006 |pages=394–402 |isbn=0-13-191833-8 |edition=8th}}</ref> This is an example of the [[corepressor (genetics)|corepressible]] model. |
Discovered in 1953 by [[Jacques Monod]] and colleagues, the trp operon in ''E. coli'' was the first repressible operon to be discovered. While the lac operon can be activated by a chemical ([[allolactose]]), the tryptophan (Trp) operon is inhibited by a chemical (tryptophan). This operon contains five structural genes: trp E, trp D, trp C, trp B, and trp A, which encodes [[tryptophan synthetase]]. It also contains a promoter which binds to RNA polymerase and an operator which blocks transcription when bound to the protein synthesized by the repressor gene (trp R) that binds to the operator. In the lac operon, lactose binds to the repressor protein and prevents it from repressing gene transcription, while in the trp operon, tryptophan binds to the repressor protein and enables it to repress gene transcription. Also unlike the lac operon, the trp operon contains a leader peptide and an [[attenuator (genetics)|attenuator]] sequence which allows for graded regulation.<ref>{{cite book |author=Cummings MS, Klug WS |title=Concepts of genetics |publisher=Pearson Education |location=Upper Saddle River, NJ |year=2006 |pages=394–402 |isbn=0-13-191833-8 |edition=8th}}</ref> This is an example of the [[corepressor (genetics)|corepressible]] model. |
||
== |
== Predición do número e organización de operóns == |
||
O número e organización de operóns foi estudado máis intensamente en ''[[E. coli]]''. Como resultado dos coñecementos adquiridos e dos coñecementos de xenómica, fixéronse predicións de operóns baseadas na secuencia xenómica dun organismo. |
O número e organización de operóns foi estudado máis intensamente en ''[[E. coli]]''. Como resultado dos coñecementos adquiridos e dos coñecementos de xenómica, fixéronse predicións de operóns baseadas na secuencia xenómica dun organismo. |
||
| Liña 65: | Liña 65: | ||
Un método de predición utiliza a distancia interxénica entre [[ORF|marcos de lectura]] como un preditor primario do número de operóns no xenoma. A separación simplemente cambia o marco e garante que a lectura é eficiente. Existen tramos máis longos onde os operóns empezan e acaban, a miúdo de ata 40–50 bases.<ref>Salgado, H.; Moreno-Hagelsieb, G.; Smith, T.; Collado-Vides, J. (2000). "Operons in Escherichia coli: Genomic analyses and predictions". Proceedings of the National Academy of Sciences 97 (12): 6652–6657. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.</ref> |
Un método de predición utiliza a distancia interxénica entre [[ORF|marcos de lectura]] como un preditor primario do número de operóns no xenoma. A separación simplemente cambia o marco e garante que a lectura é eficiente. Existen tramos máis longos onde os operóns empezan e acaban, a miúdo de ata 40–50 bases.<ref>Salgado, H.; Moreno-Hagelsieb, G.; Smith, T.; Collado-Vides, J. (2000). "Operons in Escherichia coli: Genomic analyses and predictions". Proceedings of the National Academy of Sciences 97 (12): 6652–6657. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.</ref> |
||
Un método alternativo para predicir operóns baséase en atopar ''clusters'' de xenes nos que a orde e orienración dos xenes está |
Un método alternativo para predicir operóns baséase en atopar ''clusters'' de xenes nos que a orde e orienración dos xenes está conservada en dous ou máis xenomas.<ref>Ermolaeva, M.; White, O.; Salzberg, S. (2001). "Prediction of operons in microbial genomes". Nucleic Acids Research 29 (5): 1216–1221. doi:10.1093/nar/29.5.1216. PMC 29727. PMID 11222772.</ref> |
||
A predición de operóns é mesmo máis exacta se consideramos o tipo funcional de moléculas. As bacterias teñen agrupados os seus marcos de lectura en unidades, secuestrados polas súa coimplicación na formación de complexos protícos, vías metabólicas comúns, ou compartición de substratos e transportadores. Así, unha predición exacta debería ter en conta todos estes datos, o cal é unha difícil tarefa. |
A predición de operóns é mesmo máis exacta se consideramos o tipo funcional de moléculas. As bacterias teñen agrupados os seus marcos de lectura en unidades, secuestrados polas súa coimplicación na formación de complexos protícos, vías metabólicas comúns, ou compartición de substratos e transportadores. Así, unha predición exacta debería ter en conta todos estes datos, o cal é unha difícil tarefa. |
||
Revisión como estaba o 15 de agosto de 2013 ás 21:06
Este artigo está a ser traducido ao galego por un usuario desta Wikipedia; por favor, non o edite. O usuario Miguelferig (conversa · contribucións) realizou a última edición na páxina hai 10 anos. Se o usuario non publica a tradución nun prazo de trinta días, procederase ó seu borrado rápido. |

En xenética, un operón é unha unidade funcional do ADN xenómico bacteriano ou máis raramente eucariótico ou vírico, que contén un grupo de xenes e rexións regulatorias, que están baixo o control dun só sinal regulatorio ou promotor.[1][2] Os xenes do operón son transcritos xuntos a ARNm e despois poden ser traducidos xuntos no citoplasma ou sofen primeiro un trans-splicing para crear ARNm monocistrónicos que se traducen por separado, é dicir, fórmanse varias moléculas de ARNm a partir do ARNm inicial grande, as cales codifican cada unha un só produto xénico (proteína). O resultado disto é que os xenes que contén o operón se expresan xuntos ou non se expresan en absoluto. Deben ser cotranscritos ou corregulados varios xenes para definir un operón.[3]
Inicialmente pensábase que os operóns existían só nos procariotas, pero despois atopáronse algúns en eucariotas e virus. Desdo o descubrimento dos primeiros operóns en eucariotas a principios da década de 1990,[4][5] acumuláronse máis evidencias que indican que os operóns son máis comúns do que previamente se pensaba.[6] En xeral, a expresión dos operóns procarióticos orixina a formación dun ARNm policistrónico, mentres que nos operóns eucariotas fórmanse ARNm monocistrónicos.
Tamén se atoparon operóns en virus, como os bacteriófagos.[7][8] Por exemplo, os fagos T7 teñen dous operóns, o primeiro codifica varios produtos incluíndo unha ARN polimerase de T7 especial, que pode unirse e transcribir o segundo operón, o cal contén un xene de lise, que se cre causa que a célula hóspede estoupe.[9]
Un operón contén un ou máis xenes estruturais, que se tanscriben xeralmente como un ARNm policistrónico (unha soa molécula de ARNm que codifica máis dunha proteína). Con todo, a definición de operón non require que o ARNm sexa necesariamente policistrónico, aínda que isto sexa o máis común.[10] Augas arriba dos xenes estruturais encóntrase unha secuencia promotora, que proporciona un sitio para que se una a ARN polimerase e inicie a transcrición. Preto do promotor hai unha sección do ADN denominada operador.
Historia
O termo "operón" foi proposto por primeira vez nun curto artigo publicado en 1960 pola Academia Francesa de Ciencias.[11] Neste documento explicábase a teoría xeral do operón. Esta teoría propoñia que todos os xenes están controlados por medio de operóns por un mecanismo regulatorio de retroalimentación simple, a represión. Máis tarde, descubriuse que a regulación de xenes é un proceso moito máis complicado. En realidade, non é posible falar dun mecanismos de regulación xeral, porque diferentes operóns teñen diferentes mecanismos de regulación. A pesar das modificacións que sufriu, o desenvolvemento deste concepto considérase un evento destacado na historia da bioloxía molecular. O primeiro operón que foi descrito foi o operón lac de Escherichia coli.[2] François Jacob, André Michel Lwoff e Jacques Monod foron galardoados co Premio Nobel de Medicina de 1965 polos seus descubrimentos sobre os operóns e a síntese vírica.
Introdución
Os operóns aparecen principalmente en procariotas pero tamén nalgúns eucariotas, como nematodos como Caenorhabditis elegans e a mosca ''Drosophila melanogaster''. Os xenes de ARNr xeralmente existen formando operóns, e foron atopados en moitos eucariotas incluíndo os cordados. Un operón está formado por varios xenes estruturais organizados baixo o control dun promotor común e regulados por un operador común. Defínese como un conxinto de xenes estruturais adxacentes, e sinais regulatorias adxacentes que afectan á transcrición dos xenes estruturais.[12] Os reguladores dun determinado operón, como son os represores, correpresores, e activadores, non están necesariamente codificados polo operón ao cal regulan. A localización e condicións dos reguladores, promotores, operadores e secuencias de ADN estruturais poden determinar os efectos de mutacións comúns.
Os operóns están relacionados cos regulóns, estimulóns e modulóns; pero mentres que os operons conteñen un conxunto de xenes regulados polo mesmo operador, os regulóns conteñen un conxunto de xenes que están regulados por unha soa proteína, e os estimulóns conteñen un conxunto de xenes regulados por un só estímulo celular. Segundo os seus autores o termo operón significa "operar".[13]
Estrutura xeral dun operón
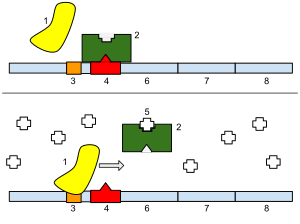
Un operón consra de 3 compoñentes básicos no ADN:
- Promotor. ë unha secuencia de nucleótidos que permite que se transcriba un xene. O promotor é recoñecido por unha ARN polimerase, a cal despois inicia a transcrición. Na síntese de ARN, os promotores indican que xenes deberían usarse para a formación do ARNm, e, por extensión, controlan as proteínas que produce a célula.
- Operador. É un segmento de ADN ao que se une un regulador. Defíniuse clasicamente no operón lac como un segmento entre o promotor e os xenes do operón.[14] Cando se une a el un represor, a proteína represora o que fai é empecer fisicamente á ARN polimerase para que esta non poida realizar a transcrición.
- Xenes estruturais. Son os xenes que son corregulados polo operón.
Ademais, poden conter un xene regulador. Este non sempre está incluído no operón, e consiste nun xene que se expresa constantemente, que codifica unha proteína represora. Este xene regulatorio non ten por que ser adxacente ou nin sequera próximo do operón.[15]
Regulación
O control dun operón é un tipo de regulación xénica que permite aos organismos regular a expresión de varios xenes dependendo das condicións ambientais. A regulación dun operón pode ser negativa ou positiva por indución ou por represión.[14]
O control negativo implica a unión dun represor ao operador, que impide a transcrición.
- Nos operóns inducibles negativos, a situación normal é que unha proteína represora reguladora estea unida sempre ao operador, o cal impide a transcrición dos xenes do operón. Se está presente unha molécula indutora, esta únese ao represor e cambia a súa conformación de modo que agora xa non pode permanecer unido ao operador. Isto permite a expresión do operón. Por tanto, son operóns que están sempre "apagados" a non ser que os "encenda" un indutor.
- Nos operóns represibles negativos, o normal é que a transcrición do operón sempre teñen lugar. Un xene regulador produce unha proteína represora, pero esta non pode unirse ao operador na súa conformación normal. Porén, certas moléculas chamadas correpresores únense á proteína represora, o que orixina un cambio conformacional no sitio activo. Isto activa á proteína represora, que xa se pode unir ao operador, impedindo a transcrición. Por tanto, o operón está sempre "encendido" a non ser que estean presentes á vez o represor e o correpresor.
Os operóns pode ser tamén controlados positivamente. No control positivo, unha proteína activadora estimula a transcrición ao unirse ao ADN (xeralmente nun sitio diferente do operador).
- Nos operóns inducibles positivos, as proteínas activadoras normalmente non poden unirse ao seu sitio no ADN. Pero cando se une un indutor á proteína activadora, esta experimenta un cambio conformacional que lle permite unirse ao ADN e activar a transcrición. Por tanto, o operón está "apagado" a non ser que estean presentes á vez o activador e o indutor.
- Nos operóns represibles positivos, as proteínas activadoras están unidas normalmente ao segmento de ADN específico para eles. Porén, cando un inhibidor se une ao activador, este xa non pode unirse ao ADN. Isto detén a activación e transcrición do sistema. Por tanto, o operón permanece "encendido" a non ser o "apague" o inhibidor.
The lac operon
- Artigo principal: lac operon.
The lac operon of the model bacterium Escherichia coli was the first operon to be discovered and provides a typical example of operon function. It consists of three adjacent structural genes, a promoter, a terminator, and an operator. The lac operon is regulated by several factors including the availability of glucose and lactose. This is an example of the derepressible (from above: negative inducible) model.
The trp operon
- Artigo principal: trp operon.
Discovered in 1953 by Jacques Monod and colleagues, the trp operon in E. coli was the first repressible operon to be discovered. While the lac operon can be activated by a chemical (allolactose), the tryptophan (Trp) operon is inhibited by a chemical (tryptophan). This operon contains five structural genes: trp E, trp D, trp C, trp B, and trp A, which encodes tryptophan synthetase. It also contains a promoter which binds to RNA polymerase and an operator which blocks transcription when bound to the protein synthesized by the repressor gene (trp R) that binds to the operator. In the lac operon, lactose binds to the repressor protein and prevents it from repressing gene transcription, while in the trp operon, tryptophan binds to the repressor protein and enables it to repress gene transcription. Also unlike the lac operon, the trp operon contains a leader peptide and an attenuator sequence which allows for graded regulation.[16] This is an example of the corepressible model.
Predición do número e organización de operóns
O número e organización de operóns foi estudado máis intensamente en E. coli. Como resultado dos coñecementos adquiridos e dos coñecementos de xenómica, fixéronse predicións de operóns baseadas na secuencia xenómica dun organismo.
Un método de predición utiliza a distancia interxénica entre marcos de lectura como un preditor primario do número de operóns no xenoma. A separación simplemente cambia o marco e garante que a lectura é eficiente. Existen tramos máis longos onde os operóns empezan e acaban, a miúdo de ata 40–50 bases.[17]
Un método alternativo para predicir operóns baséase en atopar clusters de xenes nos que a orde e orienración dos xenes está conservada en dous ou máis xenomas.[18]
A predición de operóns é mesmo máis exacta se consideramos o tipo funcional de moléculas. As bacterias teñen agrupados os seus marcos de lectura en unidades, secuestrados polas súa coimplicación na formación de complexos protícos, vías metabólicas comúns, ou compartición de substratos e transportadores. Así, unha predición exacta debería ter en conta todos estes datos, o cal é unha difícil tarefa.
O laboratorio de Pascale Cossart foi o primeiro que identificou experimentalmente todos os operóns dun microorganismo, Listeria monocytogenes. Os seus 517 operóns policistrónicos foron listados nun estudo de 2009 que describía os cambios globais na transcrición que tiñan lugar en L. monocytogenes baixo diferentes condicións.[19]
Notas
- ↑ Sadava, David; et al. (2009). Life: The Science of Biology (9th ed.). Macmillan. p. 349. ISBN 978-1-4292-1962-4.
- ↑ 2,0 2,1 Jacob, F.; Perrin, D.; Sanchez, C.; Monod, J. (Feb 1960). "Operon: a group of genes with the expression coordinated by an operator". Comptes rendus hebdomadaires des seances de l'Academie des sciences 250 (6): 1727–1729. doi:10.1016/j.crvi.2005.04.005. ISSN 0001-4036. PMID 14406329.
- ↑ Lodish, Harvey; Zipursky, Lawrence; Matsudaira, Paul; Baltimore, David; Darnel, James (2000). "Chapter 9: Molecular Definition of a Gene". Molecular Cell Biology. W. H. Freeman.
- ↑ Spieth, J.; Brooke, G.; Kuersten, S.; Lea, K.; Blumenthal, T. (1993). "Operons in C. Elegans: polycistronic mRNA precursors are processed by trans-splicing of SL2 to downstream coding regions". Cell 73 (3): 521–532. doi:10.1016/0092-8674(93)90139-H. PMID 8098272.
- ↑ Brogna, S.; Ashburner, M. (1997). "The Adh-related gene of Drosophila melanogaster is expressed as a functional dicistronic messenger RNA: Multigenic transcription in higher organisms". The EMBO Journal 16 (8): 2023–2031. doi:10.1093/emboj/16.8.2023. PMC 1169805. PMID 9155028.
- ↑ Blumenthal, T. (2004). "Operons in eukaryotes". Briefings in Functional Genomics and Proteomics 3 (3): 199–211. doi:10.1093/bfgp/3.3.199. PMID 15642184.
- ↑ "Definition of Operon". Medical Dictionary. MedicineNet.com. Consultado o 30 December 2012.
- ↑ "Displacements of Prohead Protease Genes in the Late Operons of Double-Stranded-DNA Bacteriophages". Journal of Bacteriology. 1. Consultado o 30 December 2012. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ "Bacteriophage Use Operons". Prokaryotic Gene Control. Dartmouth College. Consultado o 30 December 2012.
- ↑ Blumenthal, Thomas (2004). "Operons in Eukaryotes". Brief Funct Genomic Proteomic, 3 (3): 199–211. PMID 15642184. doi:10.1093/bfgp/3.3.199.
- ↑ Jacob, F.; Perrin, D.; Sanchez, C.; Monod, J. (Feb 1960). "Operon: a group of genes with the expression coordinated by an operator". Comptes rendus hebdomadaires des seances de l'Academie des sciences 250 (6): 1727–1729. doi:10.1016/j.crvi.2005.04.005. ISSN 0001-4036. PMID 14406329.
- ↑ Miller JH, Suzuki DT, Griffiths AJF, Lewontin RC, Wessler SR, Gelbart WM (2005). Introduction to genetic analysis (8th ed.). San Francisco: W.H. Freeman. p. 740. ISBN 0-7167-4939-4.
- ↑ Jacob, F. O. (2011). "The Birth of the Operon". Science 332 (6031): 767–767. doi:10.1126/science.1207943. PMID 21566161.
- ↑ 14,0 14,1 Lewin, Benjamin (1990). Genes IV (4th ed.). Oxford [Oxfordshire]: Oxford University Press. pp. 243–58. ISBN 0-19-854267-4.
- ↑ Mayer, Gene. "BACTERIOLOGY - CHAPTER NINE GENETIC REGULATORY MECHANISMS". Microbiology and Immunology Online. University of South Carolina School of Medicine. Consultado o 30 December 2012.
- ↑ Cummings MS, Klug WS (2006). Concepts of genetics (8th ed.). Upper Saddle River, NJ: Pearson Education. pp. 394–402. ISBN 0-13-191833-8.
- ↑ Salgado, H.; Moreno-Hagelsieb, G.; Smith, T.; Collado-Vides, J. (2000). "Operons in Escherichia coli: Genomic analyses and predictions". Proceedings of the National Academy of Sciences 97 (12): 6652–6657. doi:10.1073/pnas.110147297. PMC 18690. PMID 10823905.
- ↑ Ermolaeva, M.; White, O.; Salzberg, S. (2001). "Prediction of operons in microbial genomes". Nucleic Acids Research 29 (5): 1216–1221. doi:10.1093/nar/29.5.1216. PMC 29727. PMID 11222772.
- ↑ Toledo-Arana, A.; Dussurget, O.; Nikitas, G.; Sesto, N.; Guet-Revillet, H.; Balestrino, D.; Loh, E.; Gripenland, J. et al. (2009). "The Listeria transcriptional landscape from saprophytism to virulence". Nature 459 (7249): 950–956. doi:10.1038/nature08080. PMID 19448609.
