Pirosecuenciación
A pirosecuenciación é un método de secuenciación do ADN (é dicir, determinar a orde dos nucleótidos nunha molécula de ADN) baseándose no principio de "secuenciar por síntese", no cal a secuenciación realízase detectando o nucleótido incorporado por unha ADN polimerase. A pirosecuenciación depende da detección pola luz baseándose nunha reacción en cadea orixinada cando se libera pirofosfato. Precisamente, de aí vén o nome pirosecuenciación.
O principio da pirosecuenciación describírono primeiramente en 1993[1] Bertil Pettersson, Mathias Uhlen e Pål Nyren ao combinaren o método de secuenciación en fase sólida[2] usando boliñas magnéticas cubertas de estreptavidina cunha ADN polimerase recombinante que carecía de actividade exonuclease 3´a 5´ (a chamada corrección de probas) e a detección por luminescencia utilizando o encima luciferase de vagalume.[3] Engádese unha mestura de tres encimas (ADN polimerase, ATP sulfurilase e a luciferase de vagalume) e un nucleótido (en forma de nucleósido trifosfato, dNTP) a unha molécula de ADN monocatenario para secuenciala, e a incorporación do nucleótido (como nucleósido monofosfato) é detectada medindo a luz emitida. A intensidade da luz determina se se incorporaron 0, 1 ou máis nucleótidos, mostrando así cantos nucleótidos complementarios están presentes na febra molde. A mestura de nucleótidos é eliminada antes de que se engada a seguinte mestura de nucleótidos. Este proceso repítese con cada un dos catro nucleótidos ata que se determina a secuencia de ADN do molde monocatenario.
En 1998 Mostafa Ronaghi, Mathias Uhlen e Pål Nyren describiron un segundo método baseado en disolucións para pirosecuenciación.[4] Neste método alternativo, introdúcese un encima adicional, a apirase, para eliminar os nucleótidos que non son incorporados pola ADN polimerase. Isto permite que a mestura de encimas, que inclúe a ADN polimerase, a luciferase e a apirase, se engadan ao principio e se manteñan durante todo o procedemento, proporcionando así unha configuración simple axeitada para a automatización. Un instrumento automatizado baseado neste principio introduciuno no mercado ao ano seguinte a compañía Pyrosequencing.
Unha terceira variante microfluída do método de pirosecuenciación foi descrita en 2005[5] por Jonathan Rothberg e colaboradores na compañía 454 Life Sciences. Esta estratexia alternativa de pirosecuenciación estaba baseada no principio orixinal de ligar o ADN que se vai secuenciar a un soporte sólido e así mostraron que a secuenciación podía realizarse de maneira paralela usando un microarray microfabricado. Isto permitiu unha secuenciación de ADN de alto rendemento e introduciuse un instrumento automatizado no mercado. Este converteuse no primeiro instrumento de secuenciación de seguinte xeración que deu comezo unha nova era na investigación xenómica, cunha rápida baixada dos prezos da secuenciación do ADN posibilitando realizar secuenciacións de xenoma completo a custos asequibles.
Procedemento
[editar | editar a fonte]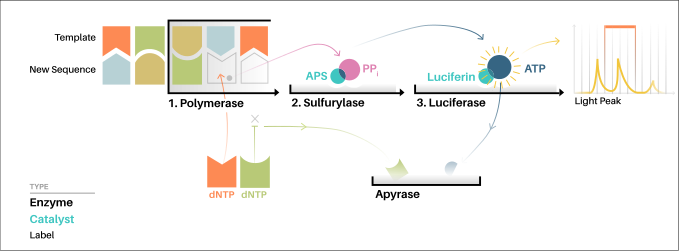
A "secuenciación por síntese" implica tomar un ADN monocatenario para secuencialo e despois sintetizar a súa cadea complementaria encimaticamente. O método de pirosecuenciación está baseado en detectar a actividade da ADN polimerase (un encima que sintetiza ADN) con outro encima quimioluminescente. Esencialmente, o método permite secuenciar un ADN monocatenario sintetizando a cadea complementaria sobre el, un par de bases á vez, e detectar que base é a que se está engadindo en cada paso. O ADN molde é inmóbil, e as solucións dos nucleótidos A, C, G e T vanse engadindo secuencialmente e son retiradas despois da reacción. Prodúcese luz soamente cando a solución de nucleótidos complementa a primeira base desapareada do molde. A secuencia de solucións que producen sinais de quimioluminescencia permite a determinación da secuencia do molde.[6]
Para a versión baseada en disolucións da pirosecuenciación, o molde de ADN monocatenario (ssDNA polas súas siglas en inglés) hibrídase a un cebador ou primer de secuenciación e incúbase cos encimas ADN polimerase, ATP sulfurilase, luciferase e apirase, e cos substratos adenosín 5´ fosfosulfato (APS) e luciferina.
- O segundo paso iníciase coa adición dun dos catro desoxinucleósidos trifosfato (dNTPs) (engádese dATPαS, que non é substrato da luciferase, en lugar de dATP para evitar ruído). A ADN polimerase incorpora os dNTPs complementarios correctos sobre o molde. Esta incorporación libera pirofosfato (PPi).
- A ATP sulfurilase converte o PPi en ATP en presenza de adenosín 5´ fosfosulfato. Este ATP actúa como un substrato para a conversión mediada pola luciferase da luciferina en oxiluciferina que xera luz visible en cantidades que son proporcionais á cantidade. A luz producida na reacción catalizada pola luciferase detéctaa unha cámara e analízase cun programa.
- Os nucleótidos non incorporados e o ATP son degradados pola apirase, e a reacción pode recomezar con outro nucleótido.
O proceso pode representarse coas seguintes ecuacións:
- PPi + APS → ATP + Sulfato (catalizado pola ATP-sulfurilase);
- ATP + luciferina + O
2 → AMP + PPi + oxiluciferina + CO
2 + hv (catalizada pola luciferase);
onde:
- PPi é pirofosfato
- APS é adenosín 5-fosfosulfato;
- ATP é adenosín trifosfato;
- O2 é unha molécula de oxíxeno;
- AMP é adenosín monofosfato;
- CO2 é dióxido de carbono;
- hv (hache ni) representa a luz.
Limitacións
[editar | editar a fonte]Actualmente a limitación do método é que as lonxitudes de cada lectura individual da secuencia de ADN están arredor dos 300-500 nucleótidos, máis curta que os 800-1000 que se poden obter con métodos de terminación de cadea (por exemplo, a secuenciación de Sanger). Isto pode facer o proceso de ensamblaxe do xenoma máis difícil, especialmente para secuencias que conteñen unha gran cantidade de ADN repetitivo. A falta de actividade de corrección de probas limita a exactitude deste método.
Comercialización
[editar | editar a fonte]A compañía Pyrosequencing AB de Uppsala, Suecia fundouse con capital de risco proporcionado por HealthCap para comercializar máquinas e reactivos para secuenciar tramos curtos de ADN usando a técnica de pirosecuenciación. Pyrosequencing AB estaba na lista da Bolsa de Valores de Estocolmo en 1999. Foi renomeada como Biotage en 2003.[7] A liña de negocio da pirosecuencación foi adquirida por Qiagen en 2008. A tecnoloxía de pirosecuenciación foille tamén autorizada a 454 Life Sciences, a cal desenvolveu unha tecnoloxía de pirosecuenciación baseada en arrays que xurdiu como unha plataforma para a secuenciación de ADN a grande escala, incluíndo secuenciación de xenomas e metaxenómica.
Roche anunciou en 2013 que non continuaría coa comercialización da plataforma 454 de secuenciación.[8]
Notas
[editar | editar a fonte]- ↑ Nyren, Pettersson and Uhlen (1993) “Solid Phase DNA Minisequencing by an Enzymatic Luminometric Inorganic Pyrophosphate Detection Assay” Analytical Biochemistry 208 (1), 171-175, https://doi.org/10.1006/abio.1993.1024
- ↑ Uhlen (1989) ”Magnetic separation of DNA” Nature 340: 733-4, https://doi.org/10.1038/340733a0
- ↑ Nyren and Lundin (1985) “Enzymatic method for continuous monitoring of inorganic pyrophosphate synthesis” Analytiocal Biochemistry 151 (2): 504-509. https://doi.org/10.1016/0003-2697(85)90211-8
- ↑ Ronaghi, Mostafa; Uhlén, Mathias; Nyrén, Pål (1998-07-17). "A Sequencing Method Based on Real-Time Pyrophosphate". Science (en inglés) 281 (5375): 363–365. PMID 9705713. doi:10.1126/science.281.5375.363.
- ↑ Marguiles et al (2005) “Genome sequencing in microfabricated high-density picolitre reactors” Nature 437, 376-380. https://doi.org/doi:10.1038/nature03959;
- ↑ QIAGEN. "Pyrosequencing Technology and Platform Overview". Consultado o 4 de agosto de 2017.
- ↑ Biotage. "Biotage History". www.biotage.com (en inglés). Consultado o 2022-09-19.
- ↑ Hollmer, Mark (17 de outubro de 2013). "Roche to close 454 Life Sciences as it reduces gene sequencing focus". Fierce Biotech.
Véxase tamén
[editar | editar a fonte]Bibliografía
[editar | editar a fonte]- Metzker M. (2005). "Emerging Technologies in DNA Sequencing". Genome Research 15 (12): 1767–76. PMID 16339375. doi:10.1101/gr.3770505.
