Fotoliase
| Fotoliase | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
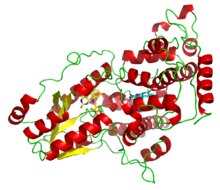 Unha deazaflavina fotoliase de Anacystis nidulans, que ilustra os dous cofactores captadores de luz: FADH− (amarelo) e 8-HDF (ciano). | |||||||||||
| Identificadores | |||||||||||
| Símbolo | FAD_binding_7 | ||||||||||
| Pfam | PF03441 | ||||||||||
| InterPro | IPR005101 | ||||||||||
| PROSITE | PDOC00331 | ||||||||||
| SCOPe | 1qnf / SUPFAM | ||||||||||
| |||||||||||
As fotoliases (EC 4.1.99.3) son encimas de reparación do ADN que reparan os danos causados pola exposición á luz ultravioleta. Este encima [1] necesita luz visible, preferentemente do extremo violeta/azul do espectro, e o mecanismo [1] que utiliza denomínase fotorreactivación.
A fotoliase é un encima filoxeneticamente antigo, que está presente e é funcional en moitas especies, desde as bacterias aos fungos, plantas e animais.[2] A fotoliase é especialmente importante para reparar os danos inducidos pola luz UV en plantas. Xenes de fotoliases perdéronse en humanos e outros mamíferos placentarios, que no seu lugar utilizan o mecanismo menos eficiente da reparación por escisión de nucleótidos.[3][4]
As fotoliases únense ás febras complementarias de ADN e rompen certos tipos de dímeros de pirimidina que se orixinan cando un par de bases de timina ou citosina da mesma febra do ADN quedan enlazadas covalentemente. Estes dímeros orixinan un 'vulto' na estrutura do ADN, denominado lesión. O enlace covalente máis común implica a formación dunha ponte de ciclobutano. As fotoliases teñen unha alta afinidade por estas lesión e únense reversiblemente a elas e convértenas de novo nas bases orixinais.

As fotoliases son flavoproteínas e conteñen dous cofactores captadores de luz. Todas as fotoliases conteñen o cofactor FADH− (reducido en dous electróns, semiquinona). Divídense en dúas clases baseándose no segundo cofactor que leven, que pode ser a pterina meteniltetrahidrofolato (MTHF) nas folato fotoliases ou a deazaflavina 8-hidroxi-7,8-didemetil-5-deazariboflavina (8-HDF) nas deazaflavina fotoliases. Aínda que só é necesario o FAD para a actividade catalítica, o segundo cofactor acelera significativamente a velocidade da reacción en condicións de baixa iluminación. O encima actúa por medio de transferencia de electróns, na cal o cofactor flavínico reducido FADH− é activado pola enerxía da luz e actúa como un doante de electróns para romper o dímero de pirimidina.[5]
Baseándose na similitude de secuencias as ADN fotoliases poden agruparse en dúas clases. A primeira clase contén encimas de bacterias grampositivas e gramnegativas, da arquea halófila Halobacterium halobium, e de fungos e plantas. Entre as proteínas que conteñen este dominio tamén están os criptocromos 1 e 2 da planta Arabidopsis thaliana, que son fotorreceptores de luz azul que funcionan como mediadores da expresión xénica inducida por luz azul e na modulación dos ritmos circadianos.
A rama de criptocromos denominada Cry-DASH (Criptocromo-Drosophila, Arabidopsis, Synechocystis, Humanos) pensábase anteriormente que non tiña actividade de reparación do ADN debido a que a súa actividade sobre o ADN bicatenario era desprezable. Mais o estudo publicado por A. Sancar e P. Selby proporcionou probas que indicaban que esta rama de criptocromos tiña actividade de fotoliase cun alto grao de especificidade polos dímeros de pirimidina ciclobutano no ADN de febra simple. O seu estudo mostrou que a VcCry1 de Vibrio cholerae, a X1Cry de Xenopus laevis, e a AtCry3 de Arabidopsis thaliana tiñan actividade de fotoliase sobre o ADN de febra simple irradiado con luz UV in vitro.[2]
Algunhas cremas protectoras solares inclúen a fotoliase entre os seus ingredientes, e salientan a súa acción reparadora sobre a pel danada polos raios UV.[6]
Proteínas humanas que conteñen este dominio
[editar | editar a fonte]Notas
[editar | editar a fonte]- ↑ 1,0 1,1 V. Thiagarajan; M. Byrdin; A.P.M. Eker; P. Müller; K. Brettel (2011). "Kinetics of cyclobutane thymine dimer splitting by DNA photolyase directly monitored in the UV". Proc. Natl. Acad. Sci. USA 108: 9402–9407. doi:10.1073/pnas.1101026108.
- ↑ 2,0 2,1 Selby, Christopher P.; Sancar, Aziz (21 November 2006). "A cryptochrome/photolyase class of enzymes with single-stranded DNA-specific photolyase activity". Proceedings of the National Academy of Sciences of the United States of America 103 (47): 17696–700. PMC 1621107. PMID 17062752. doi:10.1073/pnas.0607993103.
- ↑ Michael Lynch, José Ignacio Lucas-Lledó; Lynch, M. (19 February 2009). "Evolution of Mutation Rates: Phylogenomic Analysis of the Photolyase/Cryptochrome Family". Molecular Biology and Evolution 26 (5): 1143–1153. PMC 2668831. PMID 19228922. doi:10.1093/molbev/msp029.
- ↑ Tomohisa Kato Jr, Takeshi Todo, Hitoshi Ayaki, Kanji Ishizaki, Takashi Morita, Sankar Mitra and Mituo Ikenaga. Cloning of a marsupial DNA photolyase gene and the lack of related nucleotide sequences in placental mammals. Nucl. Acids Res. (1994) 22 (20): 4119-4124. doi: 10.1093/nar/22.20.4119. [1]
- ↑ Sancar A (2003). "Structure and function of DNA photolyase and cryptochrome blue-light photoreceptors". Chem Rev 103 (6): 2203–37. PMID 12797829. doi:10.1021/cr0204348.
- ↑ Kulms, Dagmar; Pöppelmann, Birgit; Yarosh, Daniel; Luger, Thomas A.; Krutmann, Jean; Schwarz, Thomas (1999). "Nuclear and cell membrane effects contribute independently to the induction of apoptosis in human cells exposed to UVB radiation". PNAS 96 (14): 7974–7979. PMC 22172. PMID 10393932. doi:10.1073/pnas.96.14.7974.
