Elemento PYLIS
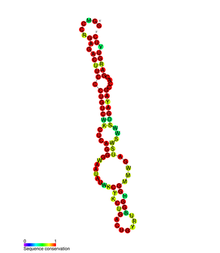
En bioloxía o elemento PYLIS ou secuencia PYLIS (do inglés pyrrolysine insertion sequence, secuencia de inserción da pirrolisina) é unha estrutura talo-bucle que aparece nalgunhas secuencias de ARNm dunhas poucas especies de procariotas (arqueas metanóxenas da familia das Methanosarcinaceae). Está ausente en eucariotas. Nas arqueas a secuencia PYLIS está situada inmediatamente despois do codón UAG en dirección ao extremo 3' do ARNm.
Propúxose que este motivo estrutural causaba que o codón de terminación UAG (ámbar), que serve normalmente para finalizar a síntese de proteínas, codificase para o aminoácido especial pirrolisina, que se incorporaría á secuencia de aminoácidos dalgúns encimas que interveñen no metabolismo do metano (monometilaminas metiltransferases) [1]. De todos modos, parece que o esencial para este cambio de función do codón UAG é que a bacteria teña os xenes PylT e PylS que codifican un ARNt especial e unha aminoacil ARNt sintetase especial, de modo que este elemento non funcionaría exactamente igual ca o elemento parecido SECIS para a selenocisteína.[2][3]
A secuencia PYLIS ten 86 nucleótidos e, aínda que contén estruturas talo-bucle, son un pouco máis complicadas ca o que se esperaba inicialmente, xa que consta de catro talos separados por tres bucles internos e acaba nun gran bucle no extremo. Este motivo está conservado na secuencia e o pregamento dos ARNm das mononometilamina metiltransferases das Methanosarcinaceae.
Notas
[editar | editar a fonte]- ↑ Théobald-Dietrich A, Giegé R, Rudinger-Thirion J (2005). "Evidence for the existence in mRNAs of a hairpin element responsible for ribosome dependent pyrrolysine insertion into proteins". Biochimie 87 (9-10): 813–7. PMID 16164991. doi:10.1016/j.biochi.2005.03.006.
- ↑ Michael Rother, Joseph A. Krzycki. Selenocysteine, Pyrrolysine, and the Unique Energy Metabolism of Methanogenic Archaea. Archaea. 2010; 2010: 453642. Published online 2010 August 17. doi: 10.1155/2010/453642 PMCID: PMC2933860. [1]
- ↑ Zhang Y, Baranov PV, Atkins JF, Gladyshev VN (2005). "Pyrrolysine and selenocysteine use dissimilar decoding strategies". J. Biol. Chem. 280 (21): 20740–51. PMID 15788401. doi:10.1074/jbc.M501458200.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]Bibliografía
[editar | editar a fonte]- Longstaff DG, Blight SK, Zhang L, Green-Church KB, Krzycki JA (2007). "In vivo contextual requirements for UAG translation as pyrrolysine". Mol. Microbiol. 63 (1): 229–41. PMID 17140411. doi:10.1111/j.1365-2958.2006.05500.x.
- Namy O, Zhou Y, Gundllapalli S; et al. (2007). "Adding pyrrolysine to the Escherichia coli genetic code". FEBS Lett. 581 (27): 5282–8. PMID 17967457. doi:10.1016/j.febslet.2007.10.022.
