ARN transferente-mensaxeiro: Diferenzas entre revisións
| Liña 50: | Liña 50: | ||
===Procesamento do ARNtm=== |
===Procesamento do ARNtm=== |
||
A maioría dos ARNtm transcríbense como precursores máis longos, que son procesados de forma similar ao ARNt. A clivaxe no extremo 5´ realízaa a [[ribonuclease P]] (RNase P).<ref name="Komine94" /> Poden participar moitas exonucleases no procesamento do extremo 3' do ARNtm, aínda que a [[RNase T]] e a [[RNase PH]] spn as máis efectivas.<ref>{{cite journal |author=Srivastava RA, Srivastava N, Apirion D |title=Characterization of the RNA processing enzyme RNase III from wild type and overexpressing Escherichia coli cells in processing natural RNA substrates |journal=Int. J. Biochem. |volume=24 |issue=5 |pages=737–49 |year=1992 |month=May |pmid=1375563 |doi= 10.1016/0020-711X(92)90007-N|url= }}</ref><ref>{{cite journal |author=Li Z, Pandit S, Deutscher MP |title=3' exoribonucleolytic trimming is a common feature of the maturation of small, stable RNAs in Escherichia coli |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=95 |issue=6 |pages=2856–61 |year=1998 |month=March |pmid=9501180 |pmc=19659 |doi= 10.1073/pnas.95.6.2856|url=http://www.pnas.org/cgi/pmidlookup?view=long&pmid=9501180 |accessdate=2010-07-14}}</ref> Dependendo da especie bacteriana, o extremo 3'-CCA é codificado ou é engadido pola [[ARNt nucleotidiltransferase]]. |
|||
Un procesamento similar nos sitios internos do ARNtm precursor permutado explica a súa separación física en dúas pezas. Os ARNtm de dúas pezas teñen dous extremos adicionais, cuxo procesamento debe tamén considerarse. Nas alfaproteobacterias, un extremo 5´ é o sitio de inicio non procesado da [[transcrición xenética|transcrición]].<ref>{{cite journal |author=Mao C, Bhardwaj K, Sharkady SM, ''et al.'' |title=Variations on the tmRNA gene |journal=RNA Biol |volume=6 |issue=4 |pages= 355–61|year=2009 |pmid=19617710 |doi= 10.4161/rna.6.4.9172|url=}}</ref> O extremo 3´ pode nalgúns casos ser o resultado dunha terminación independente de rho. |
|||
===Estruturas tridimensionais=== |
===Estruturas tridimensionais=== |
||
Revisión como estaba o 27 de novembro de 2013 ás 19:38
Este artigo está a ser traducido ao galego por un usuario desta Wikipedia; por favor, non o edite. O usuario Miguelferig (conversa · contribucións) realizou a última edición na páxina hai 10 anos. Se o usuario non publica a tradución nun prazo de trinta días, procederase ó seu borrado rápido. |
| ARN transferente-mensaxeiro | |
|---|---|
| Identificadores | |
| Rfam | RF00023 |
| Outros datos | |
| Estruturas PDB | PDBe |
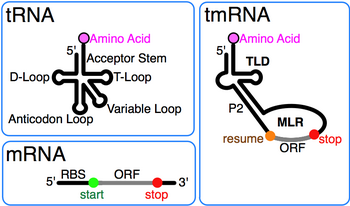
O ARN transferente-mensaxeiro, abreviado como ARNtm (en inglés tmRNA), e tamén chamado ARN 10Sa ou utilizando o seu nome xenético SsrA. é unha molécula de ARN bacteriano que presenta propiedades tanto de ARNt coma de ARNm. O ARNtm forma un complexo ribonucleoproteico (o RNPtm ou, en inglés, tmRNP) xunto coa proteína SmpB (pequena proteína B), o factor de elongación Tu (EF-Tu), e a proteína ribosómica S1. No chamada trans-tradución, o ARNtm e as súas proteínas asociadas únense aos ribosomas bacterianos que quedaron bloqueados en medio da síntese de proteínas, por exemplo cando se chega ao final dun ARN mensaxeiro que perdeu o seu codón de parada. O ARNtm é moi versátil: recicla os ribosomas que quedaron bloqueados, engade unha marca de indución á proteólise ao polipéptido inacabado, e facilita a degradación do ARNm anormal.[1] Na maioría das bacterias estas funcións lévanas a cabo ARNtm dunha soa peza. Noutras especies bacterianas, un xene ssrA permutado produce un ARNtm de dúas pezas, no cal dúas cadeas separadas de ARN se uniron por apareamento de bases.
Descubrimento dos ARNtm e traballos iniciais
O ARNtm foi inicialmente denominado ARN 10Sa porque unha fracción electroforética mixta de “10S” de ARN de Escherichia coli foi despois resolta como ARNtm e a RNase P (un ribocima de tamaño similar (10Sb).[2] A presenza de pseudouridina no ARN 10S mixto indicaba que o ARNtm tiña bases modificadas que se encontraban tamén no ARNt. A semellanza entre os extremos 3' do ARntm co talo-bucle T do ARNt foi recoñecida ao secuenciar o o ssrA de Mycobacterium tuberculosis.[3] Unha posterior comparación de secuencias indicou que o todo o dominio similar ao ARNt (TLD) formaba os extremos 5' e 3' do ARNtm, incluíndo o talo aceptor con elemntos como os atopados no ARNt da alanina, que promoven a súa aminoacilación pola alanina-ARNt ligase.[4] Tamén se encontraron diferenzas con respecto ao ARNt: o brazo anticodón non está presente nos ARNtm, e a rexión do brazo D é un bucle sen apareamentos de bases.
Estrutura do ARNtm
Estrutura secundaria dos ARNtm estándar dunha peza
A estrutura secundaria do ARNtm completo de E. coli foi dilucidada por análise de secuencia comparativo e structural probing.[5][6] As pares de bases de Watson-Crick e G-U identificáronse ao comparar as secuencias de ARNtm bacterianas utilizando métodos computacionais automatizados en combinación con procedementos de alineamento de secuencia manual.[7][8] A figura mostra o patrón de pares de bases deste ARNtm prototípico, que está organizado en 12 hélices apoiadas por evidencias filoxenéticas (tamén chamadas apareamentos P1 a P12), algúns divididos en segmentos helicoidais.
Unha característica salientable de todos os ARNtm é a conservación do dominio de tipo ARNt (TLD), composto das hélices 1, 12, e 2a (análogos, rspectivamente, do talo aceptor do ARNt, do talo T e do talo variable), que conteñen os extremos 5' monofosfato e o 3' CCA para a unión da alanina. A rexión de tipo ARNm (MLR) é no ARNtm estándar un grande bucle que contén pseudonós e unha secuencia codificante (CDS) para o péptido etiqueta, marcado polo codón de reinicio e o codón de parada. O péptido etiqueta codificado (que é o ANDENYALAA en E. coli) varía entre as bacterias, quizais dependendo do conxunto de proteases e adaptadores dispoñibles.[9]
Os ARNtm tipicamente conteñen catro pseudonós, un chamando pk1 situado corrnte arriba do péptido etiqueta CDS, e os outrso tres (de pk2 a pk4) situados corrente abaixo do CDS. As rexións pseudonó, aínda que están xeralmente conservadas, son evolutivamente plásticas. Por exemplo, nos ARNtm (dunha peza) de cianobacterias, o pk4 está substituído por dous pseudonós máis pequenos dispostos en tándem. Isto suxire que o pregamento do ARNtm fóra do TLD pode ser importante, yet a rexión pseudonó carece de residuos conservados e os pseudonós están entre as primeiras estruturas que se perden a medida que as secuencias de ssrA diverxen nos plastidios e liñaxes endosimbiónticas. O apareamento de bases na rexión dos tres pseudonós do ARNtm de E. coli é interrompida durante a trans-tradución.[7][10]
ARNtm de dúas pezas
Os ssrA permutados circularmente atopáronse en tres grandes liñaxes: i) en todas as [ alfaproteobacteria]]s e as primitivas mitocondrias dos protistas Jakobidae, ii) dous grupos separados de cianobacterias (Gloeobacter e un clado que contén a Prochlorococcus e a moitas Synechococcus), e iii) algúns membros das betaproteobacterias (Cupriavidus é algunhas Rhodocyclales).[11][12] Todas producen a mesma forma global de dúas pezas (pezas aceptora e codificante), equivalente á forma estándar nicked corrente abaixo do marco de lectura. Ningunha retén máis de dous pseudonós en comparación co ARNtm estándar de catro ou máis.
As Alphaproteobacteria teñen dúas secuencias sinatura: substitúen a secuencia típica do bucle T TΨCRANY pola secuencia GGCRGUA, e teñen a secuencia AACAGAA no bucle grande do pseudonó 3´-terminal. Nas mitocondrias perdeuse o MLR, e un grande re-permutation do ssrA mitocondrial orixinou un produto dunha soa peza no protista Jakoba libera.[13]
As cianobacterias son o mellor exemplo de evolución dun xene permutado a partir dun xene estándar, xa que presentan notables semellanzas na secuencia entre os dous tipos de xenes que aparecen en dúas cepas distintas de Synechococcus.
Procesamento do ARNtm
A maioría dos ARNtm transcríbense como precursores máis longos, que son procesados de forma similar ao ARNt. A clivaxe no extremo 5´ realízaa a ribonuclease P (RNase P).[4] Poden participar moitas exonucleases no procesamento do extremo 3' do ARNtm, aínda que a RNase T e a RNase PH spn as máis efectivas.[14][15] Dependendo da especie bacteriana, o extremo 3'-CCA é codificado ou é engadido pola ARNt nucleotidiltransferase.
Un procesamento similar nos sitios internos do ARNtm precursor permutado explica a súa separación física en dúas pezas. Os ARNtm de dúas pezas teñen dous extremos adicionais, cuxo procesamento debe tamén considerarse. Nas alfaproteobacterias, un extremo 5´ é o sitio de inicio non procesado da transcrición.[16] O extremo 3´ pode nalgúns casos ser o resultado dunha terminación independente de rho.
Estruturas tridimensionais


High-resolution structures of the complete tmRNA molecules are currently unavailable and may be difficult to obtain due the inherent flexibility of the MLR. In 2007, the crystal structure of the Thermus thermophilus TLD bound to the SmpB protein was obtained at 3 Å resolution. This structure shows that SmpB mimics the D stem and the anticodon of a canonical tRNA whereas helical section 2a of tmRNA corresponds to the variable arm of tRNA.[19] A cryo-electron microscopy study of tmRNA at an early stage of trans-translation shows the spatial relationship between the ribosome and the tmRNP (tmRNA bound to the EF-Tu protein). The TLD is located near the GTPase-associated center in the 50S ribosomal subunit; helix 5 and pseudoknots pk2 to pk4 form an arc around the beak of the 30S ribosomal subunit.[20]
Trans-tradución

Coding by tmRNA was discovered in 1995 when Simpson and coworkers overexpressed a mouse cytokine in E. coli and found several truncated cytokine-derived peptides each tagged at the carboxyl termini with the same 11-amino acid residue extension (A)ANDENYALAA. With the exception of the N-terminal alanine, which comes from the 3' end of tmRNA itself, this tag sequence was traced to a short open reading frame in E. coli tmRNA. Recognizing that the tag peptide confers proteolysis, the trans-translation model for tmRNA action was proposed.[21]
While details of the trans-translation mechanism are under investigation it is generally agreed that tmRNA first occupies the empty A site of the stalled ribosome. Subsequently, the ribosome moves from the 3' end of the truncated messenger RNA onto the resume codon of the MLR, followed by a slippage-prone stage from where translation continues normally until the in-frame tmRNA stop codon is encountered. Trans-translation is essential in some bacterial species, whereas other bacteria require tmRNA to survive when subjected to stressful growth conditions.[22] Depending on the organism, the tag peptide may be recognized by a variety of proteases or protease adapters.[9]
Elementos xenéticos móbiles e o xene do ARNtm
ssrA is both a target for some mobile DNAs and a passenger on others. It has been found interrupted by three types of mobile elements. By different strategies none of these disrupt gene function: group I introns remove themselves by self-splicing, rickettsial palindromic elements (RPEs) insert in innocuous sites, and integrase-encoding genomic islands split their target ssrA yet restore the split-off portion.[23][24][25][26]
Non-chromosomal ssrA was first detected in a genomic survey of mycobacteriophages (in 10% of the phages).[27] Other mobile elements including plasmids and genomic islands have been found bearing ssrA. One interesting case is Rhodobacter sphaeroides ATCC 17025, whose native tmRNA gene is disrupted by a genomic island; unlike all other genomic islands in tmRNA (or tRNA) genes this island has inactivated the native target gene without restoration, yet compensates by carrying its own tmRNA gene. A very unusual relative of ssrA is found in the lytic mycobacteriophage DS6A, that encodes little more than the TLD.
Notas
- ↑ Keiler KC (2008). "Biology of trans-translation". Annu. Rev. Microbiol. 62: 133–51. PMID 18557701. doi:10.1146/annurev.micro.62.081307.162948.
- ↑ Ray BK, Apirion D (1979). "Characterization of 10S RNA: a new stable rna molecule from Escherichia coli". Mol. Gen. Genet. 174 (1): 25–32. PMID 384159. doi:10.1007/BF00433301. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Tyagi JS, Kinger AK (1992). "Identification of the 10Sa RNA structural gene of Mycobacterium tuberculosis". Nucleic Acids Res. 20 (1): 138. PMC 310338. PMID 1371186. doi:10.1093/nar/20.1.138. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ 4,0 4,1 Komine Y, Kitabatake M, Yokogawa T, Nishikawa K, Inokuchi H (1994). "A tRNA-like structure is present in 10Sa RNA, a small stable RNA from Escherichia coli". Proc. Natl. Acad. Sci. U.S.A. 91 (20): 9223–7. PMC 44784. PMID 7524073. doi:10.1073/pnas.91.20.9223. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Williams KP, Bartel DP (1996). "Phylogenetic analysis of tmRNA secondary structure". RNA 2 (12): 1306–10. PMC 1369456. PMID 8972778. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Felden B, Himeno H, Muto A, McCutcheon JP, Atkins JF, Gesteland RF (1997). "Probing the structure of the Escherichia coli 10Sa RNA (tmRNA)". RNA 3 (1): 89–103. PMC 1369465. PMID 8990402. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ 7,0 7,1 Zwieb C, Wower I, Wower J (1999). "Comparative sequence analysis of tmRNA". Nucleic Acids Res 27 (10): 2063–71. PMC 148424. PMID 10219077. doi:10.1093/nar/27.10.2063.
- ↑ Andersen ES, Lind-Thomsen A, Knudsen B; et al. (2007). "Semiautomated improvement of RNA alignments". RNA 13 (11): 1850–9. PMC 2040093. PMID 17804647. doi:10.1261/rna.215407. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ 9,0 9,1 Gur E, Sauer RT (2008). "Evolution of the ssrA degradation tag in Mycoplasma: specificity switch to a different protease". Proc. Natl. Acad. Sci. U.S.A. 105 (42): 16113–8. PMC 2570983. PMID 18852454. doi:10.1073/pnas.0808802105. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Wower IK, Zwieb C, Wower J (2005). "Transfer-messenger RNA unfolds as it transits the ribosome". RNA 11 (5): 668–73. PMC 1370753. PMID 15811920. doi:10.1261/rna.7269305. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Keiler KC, Shapiro L, Williams KP (2000). "tmRNAs that encode proteolysis-inducing tags are found in all known bacterial genomes: A two-piece tmRNA functions in Caulobacter". Proc. Natl. Acad. Sci. U.S.A. 97 (14): 7778–83. PMC 16621. PMID 10884408. doi:10.1073/pnas.97.14.7778. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Sharkady SM, Williams KP (2004). "A third lineage with two-piece tmRNA". Nucleic Acids Res. 32 (15): 4531–8. PMC 516066. PMID 15326226. doi:10.1093/nar/gkh795. Consultado o 2010-07-14.
- ↑ Jacob Y, Seif E, Paquet PO, Lang BF (2004). "Loss of the mRNA-like region in mitochondrial tmRNAs of jakobids". RNA 10 (4): 605–14. PMC 1370551. PMID 15037770. doi:10.1261/rna.5227904. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Srivastava RA, Srivastava N, Apirion D (1992). "Characterization of the RNA processing enzyme RNase III from wild type and overexpressing Escherichia coli cells in processing natural RNA substrates". Int. J. Biochem. 24 (5): 737–49. PMID 1375563. doi:10.1016/0020-711X(92)90007-N. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Li Z, Pandit S, Deutscher MP (1998). "3' exoribonucleolytic trimming is a common feature of the maturation of small, stable RNAs in Escherichia coli". Proc. Natl. Acad. Sci. U.S.A. 95 (6): 2856–61. PMC 19659. PMID 9501180. doi:10.1073/pnas.95.6.2856. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Mao C, Bhardwaj K, Sharkady SM; et al. (2009). "Variations on the tmRNA gene". RNA Biol 6 (4): 355–61. PMID 19617710. doi:10.4161/rna.6.4.9172.
- ↑ Someya T, Nameki N, Hosoi H; et al. (2003). "Solution structure of a tmRNA-binding protein, SmpB, from Thermus thermophilus". FEBS Lett. 535 (1–3): 94–100. PMID 12560085. doi:10.1016/S0014-5793(02)03880-2. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Bessho Y, Shibata R, Sekine S; et al. (2007). "Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA". Proc. Natl. Acad. Sci. U.S.A. 104 (20): 8293–8. PMC 1895943. PMID 17488812. doi:10.1073/pnas.0700402104. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Bessho Y, Shibata R, Sekine S; et al. (2007). "Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA". Proc. Natl. Acad. Sci. U.S.A. 104 (20): 8293–8. PMC 1895943. PMID 17488812. doi:10.1073/pnas.0700402104.
- ↑ Valle M, Gillet R, Kaur S, Henne A, Ramakrishnan V, Frank J (2003). "Visualizing tmRNA entry into a stalled ribosome". Science 300 (5616): 127–30. PMID 12677067. doi:10.1126/science.1081798. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Keiler KC, Waller PR, Sauer RT (1996). "Role of a peptide tagging system in degradation of proteins synthesized from damaged messenger RNA". Science 271 (5251): 990–3. PMID 8584937. doi:10.1126/science.271.5251.990. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Thibonnier M, Thiberge JM, De Reuse H (2008). Ahmed, Niyaz, ed. "Trans-translation in Helicobacter pylori: essentiality of ribosome rescue and requirement of protein tagging for stress resistance and competence". PLoS ONE 3 (11): e3810. PMC 2584231. PMID 19043582. doi:10.1371/journal.pone.0003810. Consultado o 2010-07-14.
- ↑ Kirby JE, Trempy JE, Gottesman S (1994). "Excision of a P4-like cryptic prophage leads to Alp protease expression in Escherichia coli". J. Bacteriol. 176 (7): 2068–81. PMC 205313. PMID 7511583. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Williams KP (2002). "The tmRNA Website: invasion by an intron". Nucleic Acids Res. 30 (1): 179–82. PMC 99078. PMID 11752287. doi:10.1093/nar/30.1.179. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Dwyer DS (2001). "Selfish DNA and the origin of genes". Science 291 (5502): 252–3. PMID 11253208. doi:10.1126/science.291.5502.252. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Williams KP (2003). "Traffic at the tmRNA gene". J. Bacteriol. 185 (3): 1059–70. PMC 142792. PMID 12533482. doi:10.1128/JB.185.3.1059-1070.2003. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda) - ↑ Hatfull GF, Pedulla ML, Jacobs-Sera D; et al. (2006). "Exploring the mycobacteriophage metaproteome: phage genomics as an educational platform". PLoS Genet. 2 (6): e92. PMC 1475703. PMID 16789831. doi:10.1371/journal.pgen.0020092. Consultado o 2010-07-14. Parámetro descoñecido
|month=ignorado (Axuda)
Véxase tamén
Outras lecturas
- Hong, S. J.; Tran, Q. A.; Keiler, K. C. (2005). "Cell cycle-regulated degradation of tmRNA is controlled by RNase R and SmpB". Molecular Microbiology 57 (2): 565–575. doi:10.1111/j.1365-2958.2005.04709.x. PMC 3776457. PMID 15978085.
