Luciferase
| Monooxixenase similar á luciferase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura da flavoproteína non fluorescente de Photobacterium leiognathi.[1] | |||||||||
| Identificadores | |||||||||
| Símbolo | Bac_luciferase | ||||||||
| Pfam | PF00296 | ||||||||
| InterPro | IPR016048 | ||||||||
| PROSITE | PDOC00397 | ||||||||
| SCOPe | 1nfp / SUPFAM | ||||||||
| |||||||||

| |
| Estrutura da luciferase do vagalume Photinus pyralis. | |
Luciferase do vagalume
| |
| Identificadores | |
| Símbolo | ? |
| PDB | 1LCI Máis estruturas |
| UniProt | P08659 |
| Outros datos | |
| Número EC | 1.13.12.7 |
| Dominio catalítico da luciferase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
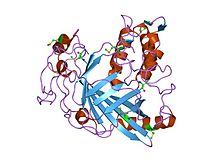 Estrutura cristalina dun dominio da luciferase do dinoflaxelado Lingulodinium polyedrum. | |||||||||
| Identificadores | |||||||||
| Símbolo | Luciferase_cat | ||||||||
| Pfam | PF10285 | ||||||||
| InterPro | IPR018804 | ||||||||
| |||||||||
| Luciferase/dominio N-terminal LBP | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Identificadores | |||||||||
| Símbolo | Luciferase_N | ||||||||
| Pfam | PF05295 | ||||||||
| InterPro | IPR007959 | ||||||||
| |||||||||
| Dominio en feixe helicoidal da luciferase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Estrutura cristalina dun dominio da luciferase do dinoflaxelado Lingulodinium polyedrum. | |||||||||
| Identificadores | |||||||||
| Símbolo | Luciferase_3H | ||||||||
| Pfam | PF10284 | ||||||||
| InterPro | IPR018475 | ||||||||
| |||||||||
A luciferase é o termo xenérico co que se denomina unha clase de encimas oxidativos que os seres vivos utilizan para producir bioluminescencia e que son distintos da fotoproteína. O exemplo de máis sona é a luciferase dos vagalumes (número EC 1.13.12.7).[2] A "luciferase de vagalume" usada como reactivo de laboratorio xeralmente é a luciferase da especie Photinus pyralis, aínda que existen tamén luciferases recombinantes doutras especies de vagalumes dispoñibles comercialmente. O nome deriva do latín lucem ferre, "levar luz", coa terminación ase propia dos encimas.
Reacción química[editar | editar a fonte]
A reacción química catalizada pola luciferase do vagalume ten lugar en dous pasos:
- luciferina + ATP → luciferil adenilato + PPi
- luciferil adenilato + O2 → oxiluciferina + AMP + luz
Emítese luz porque a reacción forma oxiluciferina nun estado excitado electrónico. A reacción libera un fotón de luz cando a oxiluciferina volve ao estado fundamental.
O luciferil adenilato pode participar adicionalmente nunha reacción co O2 na que se forma peróxido de hidróxeno e deshidroluciferil-AMP. Aproximadamente o 20% do intermediato luciferil adenilato é oxidado por esta vía.[3]
A reacción catalizada pola luciferase bacteriana é tamén un proceso oxidativo:
Na reacción un flavín mononucleótido reducido oxida un aldehido alifático de cadea longa a un ácido carboxílico alifático. A reacción forma un intermediato hidroxiflavina excitado, que é deshidratado para producir FMN e emitir luz verde-azulada.[4]
Case toda a entrada de enerxía na reacción transfórmase en luz. A reacción ten unha eficiencia entre o 80% [5] e o 90% [6]. Como comparación, unha lámpada de incandescencia corrente só converte un 10% da súa enerxía en luz,[7] e unha lámpada LED de 150 lumen por Watt (lm/W) converte o 20% da enerxía que consome en luz visible.[6]
Mecanismo[editar | editar a fonte]
A luciferase do vagalume xera luz a partir do seu substrato a luciferina nun proceso de múltiples fases. Primeiro, a D-luciferina é adenilada polo MgATP para formar luciferil adenilato e pirofosfato. Despois da activación polo ATP, o luciferil adenilato é oxidado polo oxíxeno molecular para formar un anel de dioxetanona. Unha reacción de descarboxilación orixina un estado excitado da oxiluciferina, que se tautomeriza entre as súas formas ceto e enol. A reacción finalmente emite luz cando a oxiluciferina torna ao seu estado fundamental.[8]

Estrutura da luciferase[editar | editar a fonte]
A estrutura proteica da luciferase de vagalume consta de dous dominios proteicos compactos: o dominio N-terminal e o C-terminal. O dominio N-terminal está composto de dúas follas β nunha estrutura αβαβα e un barril β. As dúas follas β están unha enriba da outra, e o barril β cobre a parte final das follas.[8]
O dominio C-terminal está conectado co N-terminal por unha zona bisagra flexible, que pode separar os dous dominios. A secuencia de aminoácidos da superficie de ambos os dominios que están un enfronte do outro é unha secuencia conservada nas luciferasas bacteriana e de vagalume, o que indica claramente que o sitio activo está localizado na fenda entre os dous dominios.[9]
Durante a reacción, a luciferase sofre un cambio conformacional e pasa á súa forma "pechada" na que os dous dominios se achegan e encerran entre eles o substrato. Isto asegura que a auga quedará excluída da reacción e non se hidrolizará o ATP ou o produto excitado electronicamente.[9]

Diferenzas espectrais en bioluminescencia[editar | editar a fonte]
A cor da bioluminescencia da luciferase pode variar entre o amarelo verdoso (λmax = 550 nm) ao vermello (λmax = 620).[10] Describíronse varios mecanismos que poden explicar como a estrutura da luciferase afecta ao espectro de emisión do fotón e á cor da luz emitida.
Un mecanismo proposto supón que a cor da luz emitida depende de se o produto está en forma ceto ou enol. O mecanismo suxire que a luz vermella a emite a forma ceto da oxiluciferina, e que a luz verdosa a emite a súa forma enol.[11][12] Porén, a 5,5-dimetiloxiluciferina emite luz verde mesmo cando está restrinxida á forma ceto porque non pode tautomerizarse.[13]
Outro mecanismo propón que a torsión do ángulo entre os aneis de benzotiazol e tiazol na oxiluciferina determina a cor da bioluminescencia. Esta explicación propón que unha forma planar cun ángulo de 0° entre os dous aneis corresponde a un estado de maior enerxía e emite unha luz verde de maior enerxía; pero cun ángulo de 90° a estrutura está nun estado de menor enerxía e emite unha luz vermella de menor enerxía.[14]
A explicación máis recente da cor de bioluminescencia examina o microambiente da oxiluciferina excitada. Os estudos realizados suxiren que as interaccións entre o produto no estado excitado e os residuos de aminoácidos das proximidades poden forzar á oxiluciferina a pasar a unha forma de maior enerxía, o que dá lugar á emisión de luz verde. Por exemplo, a Arg 218 establece interaccións electrostáticas con outros residuos próximos, impedindo a tautomerización á forma enol.[15] Igualmente, outros resultados indicaron que o microambiente da luciferase pode forzar á oxiluciferina a unha estrutura máis ríxida de alta enerxía, forzándoa a emitir unha luz verde de alta enerxía.[16]
Bifuncionalidade da luciferase[editar | editar a fonte]
A luciferase pode funcionar por dúas rutas diferentes: a vía bioluminescente e vía do CoA-ligase.[17] En ambas as dúas, a luciferase cataliza inicialmente unha reacción de adenilación con MgATP. Porén, na vía da CoA-ligase, o CoA pode desprazar o AMP para formar luciferil-CoA.
Dun modo similar a acilo graxo-CoA sintetase activa os ácidos graxos con ATP, o que vai seguido do desprazamento do AMP por CoA. Debido a estas actividades similares, a luciferase pode substituír a acilo graxo-CoA sintetase e converter ácidos graxos de cadea longa en acilos graxos-CoA por beta oxidación.[17]

Regulación[editar | editar a fonte]
A D-luciferina é o substrato da reacción de bioluminescencia da luciferase, entanto que a L-luciferina é o substrato para a actividade de luciferil-CoA sintetase. Ambas as reaccións son inhibidas polo enantiómero do substrato: a L-luciferina inhibe a vía da bioluminescencia e a D-luciferina inhibe a vía da CoA-ligase.[18] Isto mostra que a luciferase pode diferenciar perfectamente entre os dous isómeros D e L da luciferina.
A L-luciferina pode emitir unha luz feble mesmo se é un inhibidor competitivo da D-luciferina e da vía da bioluminescencia.[19] A luz emítese porque a vía da CoA sintase pode ser convertida á reacción bioluminescente ao hidrolizar por medio dunha esterase o produto final de novo a D-luciferina.[20]
A actividade de luciferase é inhibida adicionalmente pola oxiluciferina [21] e é activada alostericamente polo ATP. Cando o ATP se une aos dous sitios alostéricos do encima, increméntase a afinidade da luciferase para unirse ao ATP no seu sitio activo.[10]
Exemplos[editar | editar a fonte]
Varios organismos regulan a súa produción de luz utilizando diferentes luciferases en diversas reaccións emisoras de luz. A de maior sona é a dos vagalumes,[8] aínda que o encima existe en organismos tan diferentes como o cogomelo Omphalotus olearius e en moitas criaturas mariñas. Nos vagalumes requírese oxíxeno, que é subministrado por un tubo do abdome do aminal chamado traquea abdominal. As luciferases dos vagalumes (dos cales hai unhas 2000 especies) e dos insectos Elateroidea en xeral son o suficientemente diversas como para que sexan útiles en estudos de filoxenia molecular. A máis intensamente estudada é a luciferase do vagalume norteamericano Photinus pyralis, que funciona optimamente a un pH de 7,8.[22]
Tamén foi ben estudada a luciferase do cnidario Renilla reniformis. Neste organismo a luciferase (luciferina de Renilla 2-monooxixenase) está intimamente asociada coa proteína de unión á luciferina e cunha proteína fluorescente verde (GFP). O calcio desencadea a liberación da luciferina destes animais (coelenterazina) que estaba unida á proteína de unión á luciferina. O substrato queda despois dispoñible para a oxidación pola luciferase, onde será degradada a coelenteramida dando como resultado a liberación de enerxía. En ausencia de GFP, esta enerxía sería liberada como fotóns de luz azul (cun pico de emisión a unha lonxitude de onda de 482 nm). Porén, en vez diso, debido á GFP intimamente asociada, a emisión de enerxía pola luciferase combínase por medio de transferencia de enerxía de resonancia co fluoróforo da GFP, e é despois liberada como un fotón de luz verde (cun pico de emisión de 510 nm). A reacción catalizada é:[23]
- coelenterazina + O2 → coelenteramida + CO2 + fotón de luz
Foron identificadas recentemente novas luciferases que, a diferenza das de Renilla e do vagalume, son moléculas que se segregan fóra da célula de forma natural. Un deses exemplos é a luciferase de Metridia (MetLuc) que deriva do copépodo mariño Metridia longa. Este copépodo segrega unha luciferase codificada xeneticamente que é unha proteína de 24 kDa que contén un péptido sinal secretor N-terminal de 17 residuos de aminoácidos. Algúns dos beneficios de utilizar en diversos tipos de estudos unha molécula como a MetLuc son o seu protocolo que non prescisa a lise das células, que permite levar a cabo ensaios con células vivas e ensaios múltiples na mesma célula.[24]
Luciferase de dinoflaxelados[editar | editar a fonte]
A luciferase de dinoflaxelados é unha proteína multidominio específica dos dinoflaxelados, que consta dun dominio N-terminal, e tres dominios catalíticos, cada un dos cales está precedido dun dominio en feixe helicoidal. A estrutura do dominio catalítico da luciferase de dinoflaxelados xa foi resolto.[25] A parte central do dominio é un barril beta de 10 cadeas que é estruturalmente similar ás lipocalinas e á FABP.[25] O dominio N-terminal está conservado entre a luciferase dos dinoflaxelados e as proteínas de unión á luciferina (LBPs). Suxeriuse que esta rexión pode mediar unha interacción entre a LBP e a luciferase ou a súa asociación coa membrana vacuolar.[26] O dominio en feixe helicoidal ten unha estrutura con tres feixes helicoidais que leva catro importantes residuos de histidina que se cre xogan un papel na regulación por pH do encima.[25]
Aplicacións[editar | editar a fonte]
A luciferase pode producirse no laboratorio por técnicas de enxeñaría xenética para diversos propósitos. Os xenes das luciferases poden ser sintetizados e inseridos en organismos ou transfectados en células. O rato, o verme da seda, e as patacas son só algúns dos organismos que foron xa tratados con enxeñaría xenética para producir esta proteína.[27]
Na reacción da luciferase, emítese luz cando a luciferase actúa sobre o substrato luciferina apropiado. A emisión de fotóns pode detectarse por aparellos sensibles á luz como un luminómetro ou microscopios ópticos modificados. Isto permite a observación de procesos biolóxicos.[28]
En investigacións biolóxicas, a luciferase úsase frecuentemente como un reporter en ensaios da actividade transcricional en células que foron transfectadas cun material xenético preparado que contiña o xene da luciferase baixo o control dunha secuencia dun promotor xénico de interese.[29] Ademais, moléculas proluminescentes que son convertidas en luciferina coa actividade dun determinada encima poden urilizarse para detectar a actividade encimática en ensaios combinados ou de dous pasos da luciferase. Ditos substratos foron usados para detectar a actividade da caspase e o citocromo P450, entre outros.[28][29]
A luciferase pode tamén utilizarse para detectar o nivel de ATP celular en ensaios de viabilidade celular ou en ensaios de actividade de quinases.[29][30] A luciferase pode actuar como unha proteína sensora do ATP por medio da biotinilación. A biotinilación inmobiliza a luciferase na superficie celular ao unila a un complexo estreptavidina-biotina. Isto permite que a luciferase detecte o fluxo de ATP das células e indicará efectivamente a liberación en tempo real de ATP por medio da produción de bioluminescencia.[31] A luciferase pode ademais facerse máis sensible para a detección do ATP incrementando a intensidada da súa bioluminescencia por medio de modificacións xenéticas.[32]
A técnica de obtención de imaxes do animal completo (tanto in vivo coma, menos frecuentemente, ex vivo) é unha potente técnica para estudar poboacións celulares nos animais vivos, como nos ratos.[33] Poden prepararse por enxeñaría xenética diferentes tipos de células (por exemplo, células nai ou troncais da medula ósea ou células T) para que expresen unha luciferase, o que permitirá a súa visualización non invasiva no interior do animal vivo usando unha cámara CCD (charge-couple device). Esta técnica foi utilizada para seguir a evolución da tumoroxénese e a resposta dos tumores ao tratamento en modelos animais.[34][35] Porén, diversos factores ambientais e interferencias terapéuticas poden causar algunhas discrepancias entre a carga tumoral e a intensidade da bioluminescencia producida en relación cos cambios na actividade proliferativa. A intensidade do sinal medido por esta técnica in vivo pode depender de varios factores, como a absorción de D-luciferina a través do peritoneo, fluxo sanguíneo, permeabilidade da membrana celular, dispoñibilidade de cofactores, pH intracelular e transparencia do tecido supraxacente, ademais de da cantidade de luciferase.[36]
A luciferase pode utilizarse nos bancos de sangue para determinar se os glóbulos vermellos están empezando a descompoñerse. A luciferase é unha proteína sensible á calor que se usa en estudos sobre a desnaturalización de proteínas, comprobando as capacidades protectoras das proteínas de shock térmico. Os usos da polimerase continúan ampliándose cada ano.[37]
Notas[editar | editar a fonte]
- ↑ Moore SA, James MN (1995). "Structural refinement of the non-fluorescent flavoprotein from Photobacterium leiognathi at 1.60 A resolution". J. Mol. Biol. 249 (1): 195–214. PMID 7776372. doi:10.1006/jmbi.1995.0289.
- ↑ Gould SJ, Subramani S (1988). "Firefly luciferase as a tool in molecular and cell biology". Anal. Biochem. 175 (1): 5–13. PMID 3072883. doi:10.1016/0003-2697(88)90353-3.
- ↑ Fraga H, Fernandes D, Novotny J, Fontes R, Esteves da Silva JC (2006). "Firefly luciferase produces hydrogen peroxide as a coproduct in dehydroluciferyl adenylate formation". Chembiochem 7 (6): 929–35. PMID 16642538. doi:10.1002/cbic.200500443.
- ↑ Fisher AJ, Thompson TB, Thoden JB, Baldwin TO, Rayment I (1996). "The 1.5-A resolution crystal structure of bacterial luciferase in low salt conditions". J. Biol. Chem. 271 (36): 21956–68. PMID 8703001.
- ↑ Elizabeth Wilson (Jan 18, 1999). "What's That Stuff?". Chemical and Engineering News 77 (3): 65.
- ↑ 6,0 6,1 Vanessa Knivett (2009). "Lighting the way". EE times. Arquivado dende o orixinal o 05 de outubro de 2012. Consultado o 27 de xuño de 2012.
- ↑ General Electric TP-110, page 23, table.
- ↑ 8,0 8,1 8,2 8,3 Baldwin TO (1996). "Firefly luciferase: the structure is known, but the mystery remains". Structure 4 (3): 223–8. PMID 8805542. doi:10.1016/S0969-2126(96)00026-3.
- ↑ 9,0 9,1 9,2 Conti E, Franks NP, Brick P (1996). "Crystal structure of firefly luciferase throws light on a superfamily of adenylate-forming enzymes". Structure 4 (3): 287–98. PMID 8805533. doi:10.1016/S0969-2126(96)00033-0.
- ↑ 10,0 10,1 Ugarova NN (1989). "Luciferase of Luciola mingrelica fireflies. Kinetics and regulation mechanism". J. Biolumin. Chemilumin. 4 (1): 406–18. PMID 2801227. doi:10.1002/bio.1170040155.
- ↑ White EH, Rapaport E, Hopkins TA, Seliger HH (1969). "Chemi- and bioluminescence of firefly luciferin". J. Am. Chem. Soc. 91 (8): 2178–80. PMID 5784183.
- ↑ Fraga H (2008). "Firefly luminescence: a historical perspective and recent developments". Photochem. Photobiol. Sci. 7 (2): 146–58. PMID 18264582. doi:10.1039/b719181b.
- ↑ Branchini BR, Southworth TL, Murtiashaw MH; et al. (2004). "An alternative mechanism of bioluminescence color determination in firefly luciferase". Biochemistry 43 (23): 7255–62. PMID 15182171. doi:10.1021/bi036175d.
- ↑ McCapra F,Gilfoyle DJ, Young DW; et al. (1994). Bioluminescence and Chemiluminescence: Fundamentals and Applied.
- ↑ Nakatani N, Hasegawa JY, Nakatsuji H (2007). "Red light in chemiluminescence and yellow-green light in bioluminescence: color-tuning mechanism of firefly, Photinus pyralis, studied by the symmetry-adapted cluster-configuration interaction method". J. Am. Chem. Soc. 129 (28): 8756–65. PMID 17585760. doi:10.1021/ja0611691.
- ↑ Nakamura M, Niwa K, Maki S; et al. (2006). "Construction of a new firefly bioluminescence system using L-luciferin as substrate". Anal. Biochem. 47 (1): 1197–1200. doi:10.1016/j.tetlet.2005.12.033.
- ↑ 17,0 17,1 Oba Y, Ojika M, Inouye S (2003). "Firefly luciferase is a bifunctional enzyme: ATP-dependent monooxygenase and a long chain fatty acyl-CoA synthetase". FEBS Lett. 540 (1-3): 251–4. PMID 12681517. doi:10.1016/S0014-5793(03)00272-2.
- ↑ 18,0 18,1 Nakamura M, Maki S, Amano Y, Ohkita Y, Niwa K, Hirano T, Ohmiya Y, Niwa H (2005). "Firefly luciferase exhibits bimodal action depending on the luciferin chirality". Biochem. Biophys. Res. Commun. 331 (2): 471–5. PMID 15850783. doi:10.1016/j.bbrc.2005.03.202.
- ↑ Lembert N (1996). "Firefly luciferase can use L-luciferin to produce light". Biochem. J. 317 (1): 273–7. PMC 1217473. PMID 8694774.
- ↑ Nakamura M, Maki S, Amano Y; et al. (2005). "Firefly luciferase exhibits bimodal action depending on the luciferin chirality". Biochem. Biophys. Res. Commun. 331 (2): 471–5. PMID 15850783. doi:10.1016/j.bbrc.2005.03.202.
- ↑ Ribeiro C, Esteves da Silva JC (2008). "Kinetics of inhibition of firefly luciferase by oxyluciferin and dehydroluciferyl-adenylate". Photochem. Photobiol. Sci. 7 (9): 1085–90. PMID 18754056. doi:10.1039/b809935a.
- ↑ Steghens JP, Min KL, Bernengo JC (1998). "Firefly luciferase has two nucleotide binding sites: effect of nucleoside monophosphate and CoA on the light-emission spectra". Biochem. J. 336 (1): 109–13. PMC 1219848. PMID 9806891.
- ↑ Shimomura O (1985). "Bioluminescence in the sea: photoprotein systems". Symp. Soc. Exp. Biol. 39: 351–72. PMID 2871634.
- ↑ Baldwin TO; Lee, Jongsung; Jung, Eunsun; Kim, Sang-Cheol; Kang, Jung-Il; Lee, Jienny; Kim, Yong-Woo; Sung, Young Kwan; Kang, Hee-Kyoung (2009). "A cell-based system for screening hair growth-promoting agents.". Archives of Dermatological Research 301 (3): 381–385. PMID 19277688. doi:10.1007/s00403-009-0931-0.
- ↑ 25,0 25,1 25,2 Schultz LW, Liu L, Cegielski M, Hastings JW (2005). "Crystal structure of a pH-regulated luciferase catalyzing the bioluminescent oxidation of an open tetrapyrrole". Proc. Natl. Acad. Sci. U.S.A. 102 (5): 1378–83. PMC 547824. PMID 15665092. doi:10.1073/pnas.0409335102.
- ↑ Okamoto OK, Liu L, Robertson DL, Hastings JW (2001). "Members of a dinoflagellate luciferase gene family differ in synonymous substitution rates". Biochemistry 40 (51): 15862–8. PMID 11747464. doi:10.1021/bi011651q.
- ↑ Contag CH, Bachmann MH (2002). "Advances in in vivo bioluminescence imaging of gene expression". Annu Rev Biomed Eng 4: 235–60. PMID 12117758. doi:10.1146/annurev.bioeng.4.111901.093336.
- ↑ 28,0 28,1 "Introduction to Bioluminescence Assays". Promega Corporation. Arquivado dende o orixinal o 14 de agosto de 2010. Consultado o 2009-03-07.
- ↑ 29,0 29,1 29,2 Fan F, Wood KV (2007). "Bioluminescent assays for high-throughput screening". Assay Drug Dev Technol 5 (1): 127–36. PMID 17355205. doi:10.1089/adt.2006.053.
- ↑ Meisenheimer PL, O’Brien MA, Cali JJ (2008). "Luminogenic enzyme substrates: The basis for a new paradigm in assay design." (PDF). Promega Notes 100: 22–26. Arquivado dende o orixinal (PDF) o 06 de marzo de 2009. Consultado o 27 de xuño de 2012.
- ↑ Nakamura M, Mie M, Funabashi H, Yamamoto K, Ando J, Kobatake E (2006). "Cell-surface-localized ATP detection with immobilized firefly luciferase". Anal. Biochem. 352 (1): 61–7. PMID 16564487. doi:10.1016/j.ab.2006.02.019.
- ↑ Fujii H, Noda K, Asami Y, Kuroda A, Sakata M, Tokida A (2007). "Increase in bioluminescence intensity of firefly luciferase using genetic modification". Anal. Biochem. 366 (2): 131–6. PMID 17540326. doi:10.1016/j.ab.2007.04.018.
- ↑ Greer LF, Szalay AA (2002). "Imaging of light emission from the expression of luciferases in living cells and organisms: a review". Luminescence 17 (1): 43–74. PMID 11816060. doi:10.1002/bio.676.
- ↑ Lyons SK, Meuwissen R, Krimpenfort P, Berns A (2003). "The generation of a conditional reporter that enables bioluminescence imaging of Cre/loxP-dependent tumorigenesis in mice". Cancer Res. 63 (21): 7042–6. PMID 14612492.
- ↑ Becher OJ, Holland EC (2006). "Genetically engineered models have advantages over xenografts for preclinical studies". Cancer Res. 66 (7): 3355–8, discussion 3358–9. PMID 16585152. doi:10.1158/0008-5472.CAN-05-3827.
- ↑ Inoue Y., Tojo A., Sekine R., Soda Y., Kobayashi S., Nomura A., Izawa K., Kitamura T., Okubo T.; et al. (2006). "In vitro validation of bioluminescent monitoring of disease progression and therapeutic response in leukaemia model animals". European Journal of Nuclear Medicine & Molecular Imaging 33 (5): 557–565. doi:10.1007/s00259-005-0048-4.
- ↑ Massoud TF, Paulmurugan R, De A, Ray P, Gambhir SS (2007). "Reporter gene imaging of protein-protein interactions in living subjects". Curr. Opin. Biotechnol. 18 (1): 31–7. PMID 17254764. doi:10.1016/j.copbio.2007.01.007.
Véxase tamén[editar | editar a fonte]
Outros artigos[editar | editar a fonte]
Ligazóns externas[editar | editar a fonte]
- Trimmer B, Zayas R, Qazi S, Lewis S, Michel T, Dudzinski D, Aprille J, Lagace C (2001-06-28). "Firefly flashes and Nitric Oxide". Tufts University. Consultado o 2008-10-02.
- "Trends in development of reporter genes". reportergene.com. Consultado o 2009-03-07.
- "BL Web: Luciferin types". The Bioluminescence Web Page. University of California, Santa Barbara. Consultado o 2009-03-07.
- "Bioluminescence Reporters Protocols and Applications Guide". Protocols and applications. Promega Corporation. Arquivado dende o orixinal o 08 de agosto de 2010. Consultado o 2009-03-07.
- "BL Web: Luciferin types". ISCID Encyclopedia of Science and Philosophy. ISCID. Arquivado dende o orixinal o 21 de setembro de 2012. Consultado o 2010-04-20.
