Histona H1
| Histona H1 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||
| Identificadores | |||||||||||
| Símbolo | Linker_histone | ||||||||||
| Pfam | PF00538 | ||||||||||
| InterPro | IPR005818 | ||||||||||
| SMART | SM00526 | ||||||||||
| SCOPe | 1hst / SUPFAM | ||||||||||
| |||||||||||
A histona H1 é unha das cinco familias de proteínas histonas principais, que compoñen a cromatina das células eucarióticas. Aínda que a súa secuencia está moi conservada, é a histona máis variable entre especies. A histona H1 denomínase histona de unión ou enlace ou linker. Nas aves descubriuse a histona H5, que se considera unha isoforma da histona H1.
Estrutura[editar | editar a fonte]
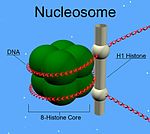
As proteínas H1 de metazoos presentan un dominio globular central e unha cola C-terminal longa e outra N-terminal curta. A histona H1 está implicada no empaquetamento das subestruturas que semellan "doas dun colar" da cromatina, chamadas nucleosomas, nunha estrutura de orde superior, da que non se coñecen todos os detalles.[1]
Función[editar | editar a fonte]
As funcións que se propuxeron para a H1 son: estabilizar as dúas espiras do ADN que rodean o nucleosoma, participar no espazado dos nucleosomas e a formación de estruturas de orde superior na cromatina, e probable implicación na regulación da expresión xénica.
A diferenza doutras histonas, a H1 non forma parte do núcleo (core) do nucleosoma, senón que se sitúa sobre dita estrutura, mantendo no seu sitio o ADN que se envolve arredor do nucleosoma. A cantidade de histona H1 é a metade ca a das outras histonas, de cada unha das cales hai dúas moléculas en cada nucleosoma. Ademais de unirse ao nucleosoma, a histona H1 únese ao ADN espazador (linker DNA) (tramo de aproximadamente 20-80 nucleótidos de lonxitude situado entre un nucleosoma e outro), que axuda a estabilizar a fibra cromatínica de 30 nm en zigzag.[2] Os estudos de fibras cromatínicas purificadas deron moita información sobre as histonas H1. A extracción iónica das histonas H1 da cromatina reconstituída ou nativa promove o seu despregamento en condicións hipotónicas de modo que a fibra cromatínica de 30 nm convértese nunha cadea de nucleosomas de tipo "colar de doas" de 10 nm de grosor. Por tanto, a histona H1 dalgunha maneira intervén na estabilización da cromatina de 30 nm.[3][4][5]
Non se sabe seguro se o que fai a H1 é promover a formación dunha fibra de cromatina de tipo solenoide de 30 nm, na cal se acurta o ADN espazador exposto, ou ben se simplemente promove un cambio no ángulo entre nucleosomas adxacentes, sen afectar á lonxitude do ADN espazador.[6] Os experimentos de dixestión con nucleases e ADN footprinting suxiren que o dominio globular da histona H1 se localiza preto da díade do nucleosoma, onde protexe aproximadamente 15-30 pares de bases adicionais de ADN.[7][8][9][10]
Ademais, os experimentos en cromatina reconstituída revelan un motivo talo característico na díade en presenza da histona H1.[11] Malia as lagoas que existen na nosa comprensión da estrutura, construíuse un modelo xeral no que o dominio globular da H1 pecha o nucleosoma ao enlazarse ao ADN que entra e ao que sae do nucleosoma, á vez que o dominio da cola se une ao ADN espazador e neutraliza a carga negativa deste.[6][9]
Realizáronse moitos experimentos para determinar a función da H1 en cromatina procesada purificada en condicións de baixa concentración salina, pero o papel da H1 in vivo é menos seguro. Os estudos celulares mostraron que a sobreexpresión de H1 pode orixinar unha morfoloxía nuclear e unha estrutura da cromatina anormais, e que a H1 pode servir tanto coma regulador positivo coma negativo da transcrición, dependendo do xene.[12][13][14] En extractos de ovos do anfibio Xenopus, a depleción da histona H1 causa que a lonxitude dos cromosomas mitóticos pase a ser o dobre do normal, mentres que a sobreexpresión da H1 causa que os cromosomas se hipercompacten formando unha masa inseparable.[15][16] Non se conseguiu un knockout completo para a H1 in vivo en organismos multicelulares debido á existencia de múltiples isoformas que poden estar presentes en varios clusters xénicos, pero conseguiuse a depleción de varias isoformas da histona H1 en diversos graos en organismos como o protozoo Tetrahymena, o verme nematodo Caenorhabditis elegans, a planta Arabidopsis, a mosca da froita e no rato, que orixinou varios defectos específicos de cada organismo na morfoloxía nuclear, estrutura da cromatina, metilación do ADN, e expresión de xenes específicos.[17][18][19]
Dinámica[editar | editar a fonte]
Unha gran sorpresa foi o descubrimento recente feito en experimentos de fotoblanqueo (phtobleaching) de que as histonas H1 son un compoñente da cromatina moito máis dinámico ca as histonas do núcleo do nucleosoma. A histona H1 está constantemente separándose da comatina e volvéndose a unir a outras rexións cromatínicas.[20][21]
É difícil comprender como unha proteína dinámica pode ser un compoñente estrutural da cromatina, pero suxeriuse que o equilibrio de estado estacionario no núcleo favorece claramente a asociación entre a H1 e a cromatina, o que significa que malia a súa dinámica, a gran maioría da H1 en calquera momento dado está unida á cromatina.[22]
Parece que son precisos factores citoplásmicos para o intercambio dinámico da histona H1 na cromatina, pero non foron aínda identificados.[23] A dinámica da H1 pode ser mediada en certa medida por O-glicosilación e fosforilación. A O-glicosilación da H1 pode promover a condensación e compactación da cromatina. A fosforilación durante a interfase sábese que fai diminuír a afinidade da H1 pola cromatina e pode promover a descondensación da cromatina e a transcrición activa. Porén, durante a mitose a fosforilación incrementa a afinidade da H1 polos cromosomas e, por tanto, promove a condensación cromosómica mitótica (véxase máis abaixo condensación mitótica por CDK1).[16]
Isoformas[editar | editar a fonte]
A familia da H1 nos animais comprende moitas isoformas da H1 que poden expresarse en tecidos diferentes ou solapados e distintos estados do desenvolvemento dun mesmo organismo. A razón da existencia destas múltiples isoformas non está aínda clara, pero tanto a súa conservación evolutiva desde o ourizo de mar aos humanos coma as significativas diferenzas nas súas secuencias de aminoácidos suxiren que non son funcionalmente equivalentes.[24][25][26] Unha isoforma é a histona H5, que se encontra só nos eritrocitos de aves, os cales, a diferenza dos de mamíferos, teñen núcleo celular. Outra isoforma é a H1M ovocítica/cigótica (tamén chamada B4 ou H1foo), que se encontra en ourizos de mar, ras, ratos, e humanos, que é substituída no embrión polas isoformas somáticas H1A-E, e H10, a cal lembra á H5.[26][27][28][29] A H1M, a pesar de ter máis cargas negativas ca as isoformas somáticas, únese con alta afinidade aos cromosomas mitóticos nos extractos de ovos de Xenopus.[16]
Fosforilación mitótica por CDK1[editar | editar a fonte]
Na mitose, as isoformas somáticas da H1 sofren fosforilación en moitos sitios de consenso da quinase dependente de ciclina 1 (CDK1), que introducen na molécula moitas cargas negativas. A fosforilación incrementa a afinidade da H1 polos cromosomas mitóticos en extractos de ovos e embrións de Xenopus, como se determinou por recuperación por fluorescencia despois de fotoblanqueo (photobleaching) de tipos salvaxes de H1 contra mutantes non fosforilables e de punto fosfomimético. Estes aumentos na afinidade de unión parecían compensar a dilución citoplasmática da H1 que se daba nas células mitóticas despois da disgregación da envoltura nuclear, ilustrando como a célula pode regular a H1 para adaptarse a condicións celulares específicas.[16]
Notas[editar | editar a fonte]
- ↑ Ramakrishnan V, Finch JT, Graziano V, Lee PL, Sweet RM (1993). "Crystal structure of globular domain of histone H5 and its implications for nucleosome binding". Nature 362 (6417): 219–23. PMID 8384699. doi:10.1038/362219a0.
- ↑ Jeon, Kwang W.; Berezney, Ronald (1995). Structural and functional organization of the nuclear matrix. Boston: Academic Press. pp. 214–7. ISBN 0-12-364565-4.
- ↑ Finch JT, Klug A (1976). "Solenoidal model for superstructure in chromatin". Proc. Natl. Acad. Sci. U.S.A. 73 (6): 1897–901. PMC 430414. PMID 1064861. doi:10.1073/pnas.73.6.1897.
- ↑ Thoma F, Koller T (1977). "Influence of histone H1 on chromatin structure". Cell 12 (1): 101–7. PMID 561660. doi:10.1016/0092-8674(77)90188-X.
- ↑ Thoma F, Koller T, Klug A (1979). "Involvement of histone H1 in the organization of the nucleosome and of the salt-dependent superstructures of chromatin". J. Cell Biol. 83 (2 Pt 1): 403–27. PMC 2111545. PMID 387806. doi:10.1083/jcb.83.2.403.
- ↑ 6,0 6,1 van Holde K, Zlatanova J (1996). "What determines the folding of the chromatin fiber?". Proc. Natl. Acad. Sci. U.S.A. 93 (20): 10548–55. PMC 38190. PMID 8855215. doi:10.1073/pnas.93.20.10548.
- ↑ Varshavsky AJ, Bakayev VV, Georgiev GP (1976). "Heterogeneity of chromatin subunits in vitro and location of histone H1". Nucleic Acids Res. 3 (2): 477–92. PMC 342917. PMID 1257057. doi:10.1093/nar/3.2.477.
- ↑ Whitlock JP, Simpson RT (1976). "Removal of histone H1 exposes a fifty base pair DNA segment between nucleosomes". Biochemistry 15 (15): 3307–14. PMID 952859. doi:10.1021/bi00660a022.
- ↑ 9,0 9,1 Allan J, Hartman PG, Crane-Robinson C, Aviles FX (1980). "The structure of histone H1 and its location in chromatin". Nature 288 (5792): 675–9. PMID 7453800. doi:10.1038/288675a0.
- ↑ Staynov DZ, Crane-Robinson C (1988). "Footprinting of linker histones H5 and H1 on the nucleosome". EMBO J. 7 (12): 3685–91. PMC 454941. PMID 3208745.
- ↑ Bednar J, Horowitz RA, Grigoryev SA, Carruthers LM, Hansen JC, Koster AJ, Woodcock CL (1998). "Nucleosomes, linker DNA, and linker histone form a unique structural motif that directs the higher-order folding and compaction of chromatin". Proc. Natl. Acad. Sci. U.S.A. 95 (24): 14173–8. PMC 24346. PMID 9826673. doi:10.1073/pnas.95.24.14173.
- ↑ Dworkin-Rastl E, Kandolf H, Smith RC (1994). "The maternal histone H1 variant, H1M (B4 protein), is the predominant H1 histone in Xenopus pregastrula embryos". Dev. Biol. 161 (2): 425–39. PMID 8313993. doi:10.1006/dbio.1994.1042.
- ↑ Brown DT, Alexander BT, Sittman DB (1996). "Differential effect of H1 variant overexpression on cell cycle progression and gene expression". Nucleic Acids Res. 24 (3): 486–93. PMC 145659. PMID 8602362. doi:10.1093/nar/24.3.486.
- ↑ Gunjan A, Alexander BT, Sittman DB, Brown DT (1999). "Effects of H1 histone variant overexpression on chromatin structure". J. Biol. Chem. 274 (53): 37950–6. PMID 10608862. doi:10.1074/jbc.274.53.37950.
- ↑ Maresca TJ, Freedman BS, Heald R (2005). "Histone H1 is essential for mitotic chromosome architecture and segregation in Xenopus laevis egg extracts". J. Cell Biol. 169 (6): 859–69. PMC 2171634. PMID 15967810. doi:10.1083/jcb.200503031.
- ↑ 16,0 16,1 16,2 16,3 Freedman BS, Heald R (2010). "Functional Comparison of Linker Histones in Xenopus Reveals Isoform-Specific Regulation by Cdk1 and RanGTP". Curr. Biol. 20 (11): 1048–52. PMC 2902237. PMID 20471264. doi:10.1016/j.cub.2010.04.025.
- ↑ Shen X, Yu L, Weir JW, Gorovsky MA (1995). "Linker histones are not essential and affect chromatin condensation in vivo". Cell 82 (1): 47–56. PMID 7606784. doi:10.1016/0092-8674(95)90051-9.
- ↑ Jedrusik MA, Schulze E (2001). "A single histone H1 isoform (H1.1) is essential for chromatin silencing and germline development in Caenorhabditis elegans". Development 128 (7): 1069–80. PMID 11245572.
- ↑ Lu X, Wontakal SN, Emelyanov AV, Morcillo P, Konev AY, Fyodorov DV, Skoultchi AI (2009). "Linker histone H1 is essential for Drosophila development, the establishment of pericentric heterochromatin, and a normal polytene chromosome structure". Genes Dev. 23 (4): 452–65. PMC 2648648. PMID 19196654. doi:10.1101/gad.1749309.
- ↑ Misteli T, Gunjan A, Hock R, Bustin M, Brown DT (2000). "Dynamic binding of histone H1 to chromatin in living cells". Nature 408 (6814): 877–81. PMID 11130729. doi:10.1038/35048610.
- ↑ Chen D, Dundr M, Wang C, Leung A, Lamond A, Misteli T, Huang S (2005). "Condensed mitotic chromatin is accessible to transcription factors and chromatin structural proteins". J. Cell Biol. 168 (1): 41–54. PMC 2171683. PMID 15623580. doi:10.1083/jcb.200407182.
- ↑ Bustin M, Catez F, Lim JH (2005). "The dynamics of histone H1 function in chromatin". Mol. Cell 17 (5): 617–20. PMID 15749012. doi:10.1016/j.molcel.2005.02.019.
- ↑ Freedman BS, Miller KE, Heald R (2010). Cimini, Daniela, ed. "Xenopus Egg Extracts Increase Dynamics of Histone H1 on Sperm Chromatin". PLoS ONE 5 (9): e13111. PMC 2947519. PMID 20927327. doi:10.1371/journal.pone.0013111.
- ↑ Steinbach OC, Wolffe AP, Rupp RA (1997). "Somatic linker histones cause loss of mesodermal competence in Xenopus". Nature 389 (6649): 395–9. PMID 9311783. doi:10.1038/38755.
- ↑ De S, Brown DT, Lu ZH, Leno GH, Wellman SE, Sittman DB (2002). "Histone H1 variants differentially inhibit DNA replication through an affinity for chromatin mediated by their carboxyl-terminal domains". Gene 292 (1–2): 173–81. PMID 12119111. doi:10.1016/S0378-1119(02)00675-3.
- ↑ 26,0 26,1 Izzo A, Kamieniarz K, Schneider R (2008). "The histone H1 family: specific members, specific functions?". Biol. Chem. 389 (4): 333–43. PMID 18208346. doi:10.1515/BC.2008.037.
- ↑ Khochbin S (2001). "Histone H1 diversity: bridging regulatory signals to linker histone function". Gene 271 (1): 1–12. PMID 11410360. doi:10.1016/S0378-1119(01)00495-4.
- ↑ Godde JS, Ura K (2008). "Cracking the enigmatic linker histone code". J. Biochem. 143 (3): 287–93. PMID 18234717. doi:10.1093/jb/mvn013.
- ↑ Happel N, Doenecke D (2009). "Histone H1 and its isoforms: contribution to chromatin structure and function". Gene 431 (1–2): 1–12. PMID 19059319. doi:10.1016/j.gene.2008.11.003.
