Gancho AT
| AT_hook | |||||||||
|---|---|---|---|---|---|---|---|---|---|
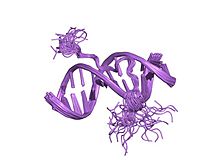 estrutura dun complexo do segundo dominio de unión ao ADN da HMG-I(y) humana unida a un ADN dodecámero que contén o sitio prdii do promotor do interferón-beta, nmr, 35 structuras | |||||||||
| Identificadores | |||||||||
| Símbolo | AT_hook | ||||||||
| Pfam | PF02178 | ||||||||
| InterPro | IPR017956 | ||||||||
| SMART | AT_hook | ||||||||
| SCOPe | 2eze / SUPFAM | ||||||||
| |||||||||

O gancho AT ou amocelo AT (AT-hook) é un motivo estrutural para a unión ao ADN presente en moitas proteínas, como por exemplo as proteínas HMG (grupo de alta mobilidade),[1] as proteínas de unión ao ADN de plantas [2] a proteína hBRG1, e a ATPase central do complexo proteico de remodelación SWI/SNF humano (switching/sucrose non-fermenting).[3]
Este motivo consiste nunha secuencia central palindrómica conservada formada polos aminoácidos prolina-arxinina-glicina-arxinina-prolina, aínda que algúns ganchos AT conteñen só unha soa prolina na secuencia central. Os ganchos AT tamén inclúen un número variable de residuos de lisina e arxinina cargados positivamente a cada lado da secuencia central.[4] O gancho AT únese ao suco menor dun ADN rico en adenina-timina (AT), de onde vén o nome AT. O nome de gancho ou amocelo procede do gancho (hook) de asparaxina/aspartato predito nos primeiros ganchos AT dos que se informou en 1990.[5] En 1997 os estudos estruturais usando resonancia magnética nuclear determinaron que un gancho AT unido ao ADN adoptaba unha forma de crecente ou gancho arredor do suco menor da fibra de ADN diana (como se ve na imaxe da dereita).[6] As proteínas HMGA conteñen tres ganchos AT, aínda que algunhas proteínas conteñen ata 30.[5] As secuencias de unión óptimas para as proteínas de gancho AT son repeticións de tipo (ATAA)n ou (TATT)n, aínda que as secuencias óptimas para a secuencia central do gancho AT son AAAT e AATT.[7]
Notas[editar | editar a fonte]
- ↑ Reeves R, Beckerbauer L (2001). "HMGI/Y proteins: flexible regulators of transcription and chromatin structure". Biochim. Biophys. Acta 1519 (1–2): 13–29. PMID 11406267. doi:10.1016/S0167-4781(01)00215-9.
- ↑ Meijer AH, van Dijk EL, Hoge JH (1996). "Novel members of a family of AT hook-containing DNA-binding proteins from rice are identified through their in vitro interaction with consensus target sites of plant and animal homeodomain proteins". Plant Mol. Biol. 31 (3): 607–18. PMID 8790293. doi:10.1007/BF00042233.
- ↑ Singh M, D'Silva L, Holak TA (2006). "DNA-binding properties of the recombinant high-mobility-group-like AT-hook-containing region from human BRG1 protein". Biol. Chem. 387 (10–11): 1469–78. PMID 17081121. doi:10.1515/BC.2006.184.
- ↑ Reeves R (2001). "Molecular biology of HMGA proteins: hubs of nuclear function". Gene 277 (1–2): 63–81. PMID 11602345. doi:10.1016/S0378-1119(01)00689-8.
- ↑ 5,0 5,1 Reeves R, Nissen MS (1990). "The A.T-DNA-binding domain of mammalian high mobility group I chromosomal proteins. A novel peptide motif for recognizing DNA structure". J. Biol. Chem. 265 (15): 8573–82. PMID 1692833. Arquivado dende o orixinal o 07 de xuño de 2020. Consultado o 18 de xullo de 2013.
- ↑ Huth JR, Bewley CA, Nissen MS; et al. (1997). "The solution structure of an HMG-I(Y)-DNA complex defines a new architectural minor groove binding motif". Nat. Struct. Biol. 4 (8): 657–65. PMID 9253416. doi:10.1038/nsb0897-657.
- ↑ Reeves R (2000). "Structure and function of the HMGI(Y) family of architectural transcription factors". Environ. Health Perspect. 108 (Suppl 5): 803–9. JSTOR 3454310. PMID 11035986. doi:10.2307/3454310. Arquivado dende o orixinal o 09 de xaneiro de 2009. Consultado o 18 de xullo de 2013.
