Citocromo c oxidase
| Citocromo c oxidase | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| |||||||||
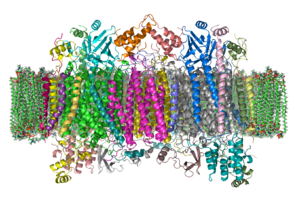
| Estrutura cristalina da citocromo c oxidase bovina nunha bicapa fosfolipídica. O espazo intermembrana está arriba na imaxe. Adaptado de PDB 1OCC. (É un homodímero nesta estrutura). | |||||||||
| Identificadores | |||||||||
| Número EC | 1.9.3.1 | ||||||||
| Número CAS | 9001-16-5 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| Gene Ontology | AmiGO / EGO | ||||||||
| |||||||||



A citocromo c oxidase ou Complexo IV respiratorio é un complexo encimático co número EC 1.9.3.1 que forma o último elemento da cadea de transporte de electróns respiratoria, polo que é unha grande complexo proteico transmembrana que se encontra na membrana mitocondrial interna dos eucariotas e na membrana plasmática das bacterias.
Recibe un electrón de cada unha das catro moléculas de citocromo c da cadea, e transfíreos a unha molécula de oxíxeno, convertendo o oxíxeno molecular en dúas moléculas de auga, para o cal se une a catro protóns procedentes da fase acuosa do interior da mitocondria. Ademais transloca catro protóns a través da membrana, axudando a establecer unha diferenza na cantidade de protóns a cada lado da membrana, creando un potencial electroquímico na membrana que se utilizará para activar a ATP sintase (fosforilación oxidativa) para que produza ATP, molécula na que se almacena enerxía.
Estrutura[editar | editar a fonte]
O complexo é un grande complexo proteico de membrana que consta de varios grupos prostéticos metálicos e 14 [1] subunidades proteicas en mamíferos. Nos mamíferos, 11 das subunidades orixínanse en xenes nucleares, e tres son sintetizadas na propia mitocondria. O complexo contén dous grupos hemo, que forman parte dun citocromo a e un citocromo a3, e dous centros con cobre chamados CuA e CuB.[2] O citocromo a3 e o CuB forman un centro binuclear que é o sitio onde se produce a redución do oxíxeno. O citocromo c, que é reducido polo compoñente precedente da cadea respiratoria (o complexo do citocromo bc1 ou complexo III), colócase preto do centro binuclear CuA e cédelle un electrón, o que fai que se oxide de novo a citocromo c Fe3+. O centro binulear CuA reducido cede agora un electrón ao citocromo a, que á súa vez pasa un electrón ao centro binuclear citocromo a3-CuB. Os dous ións metálicos deste centro binuclear están separados 4,5 Å e coordinan un ión hidróxido (OH-) no estado oxidado completo.
Os estudos de cristalografía de raios X da citocromo c oxidase indican que ten unha infrecuente modificación postraducional, que enlaza o C6 da Tyr(244) e o ε-N da His(240) (coa numeración do encima bovino). Xoga un papel vital para capacitar ao centro binuclear citocromo a3- CuB para aceptar catro electróns durante a redución do oxíxeno molecular a auga. Antes pensábase que o mecanismo de redución implicaba un intermediato peróxido, que se cría que orixinaba a produción de superóxido. Pero o mecanismo que se acepta actualmente consiste nunha rápida redución de catro electróns que implica a rotura inmediata dun enlace oxíxeno-oxíxeno, evitando calquera intermediato que puidese formar superóxido.[3]
Ensamblaxe[editar | editar a fonte]
O sitio onde ocorre a ensamblaxe do complexo crese que é preto dos complexos transportadores da membrana interna e externa da mitocondria ou TIM/TOM, onde os intermediatos do complexo están accesibles para unirse ás subunidades importadas do citosol. Os hemos e os cofactores están inseridos nas subunidades I e II. As subunidades I e IV son as que inician a ensamblaxe. Outras subunidades poden formar subcomplexos intermediarios que máis tarde se unen a outros para formar o complexo da citocromo c oxidase (COX). Durante as modificacións post-ensamblaxe, o encima é dimerizado, o cal é necesario para a acción activa/eficaz do encima. Os dímeros están conectados por unha molécula de cardiolipina.[4][5]
Bioquímica[editar | editar a fonte]
Resumo da reacción:
- 4 Fe2+-citocromo c + 8 H+entran + O2 → 4 Fe3+-citocromo c + 2 H2O + 4 H+saen
Pasan dous electróns desde dous citocromos c, a través dos sitios do CuA e citocromo a, ata o centro binuclear citocromo a3- CuB, reducindo os metais á forma Fe2+ e Cu+. O ligando hidróxido é protonado e pérdese en forma de auga, creando un baleiro entre os metais que encherá un O2. A molécula de oxíxeno é rapidamente reducida, con dous electróns procedentes do Fe2+ do citocromo a3, o cal se converte á forma ferril oxo (Fe4+=O). O átomo de oxíxeno que está preto do CuB recolle un electrón do Cu+, e tamén un segundo electrón e un protón procedente do hidroxilo da Tyr(244), que se converte nun radical tirosil. O segundo oxíxeno convértese nun ión hidróxido ao captar dous electróns e un protón. Un terceiro electrón procedente doutro citocromo c pasa a través dos dous primeiros transportadores de electróns ao centro binuclear citocromo a3- CuB, e este electrón e dous protóns converten o radical tirosil de novo en Tyr, e á vez o hidróxido unido ao CuB2+ convértese nunha molécula de auga. O cuarto electrón doutro citocromo c flúe a través do CuA e o citocromo a ata o centro binuclear citocromo a3- CuB, reducindo o Fe4+=O a Fe3+, e o átomo de oxíxeno capta un protón simultaneamente, rexenerando este oxíxeno como un ión hidróxido coordinado no medio do centro citocromo a3- CuB tal como estaba ao principio deste ciclo. O proceso neto é que se usan catro citocromos c reducidos, xunto con catro protóns, para reducir o O2 a dúas moléculas de auga.
Inhibición[editar | editar a fonte]
O cianuro, sulfuro, azida, e monóxido de carbono[6] únense á citocromo oxidase e inhíbena, o cal produce a asfixia química das células. O metanol convértese en ácido fórmico, que tamén inhibe o mesmo sistema da oxidase.
Localización subcelular e presenza en sitios extramitocondriais[editar | editar a fonte]
A citocromo c oxidase ten 3 subunidades que están codificadas no ADN mitocondrial. Destas 3 subunidades codificadas nas mitocondrias, dúas foron identificadas en localizacións extramitocondriais. No tecido pancreático acinar, estas subunidades foron atopadas en gránulos de cimóxeno. Ademais, na pituitaria anterior, atopáronse cantidades relativamente grandes destas subunidades en gránulos secretorios de hormona do crecemento.[7] A función extramitocondrial destas subunidades da citocromo c oxidase non foi aínda caracterizada. Ademais de nas subunidades da citocromo c oxidase, observouse tamén localizacións extramitocondriais en moitas outras proteínas mitocondriais.[8][9] Isto indica a posibilidade de que existan mecanismos específicos aínda non identificados para a translocación de proteínas desde as mitocondrias a outros destinos celulares.[7][9][10]
Trastornos e defectos xenéticos[editar | editar a fonte]
Os defectos que implican mutación`s xenéticas que alteran a funcionalidade da citocromo c oxidase (COX) ou a súa estrutura poden dar lugar a graves e ás veces mortais trastornos metabólicos. Tales trastornos maniféstanse xeralmente nos primeiros anos da infancia e afectan predominantemente a tecidos con alta demanda de enerxía (cerebro, corazón, músculo). Entre as moitas enfermidades mitocondriais clasificadas, as que implican unha ensamblaxe disfuncional da citocromo c oxidase crese que son as máis graves.[11]
A gran maioría dos trastornos da citocromo c oxidase están ligadas a mutacións nas proteínas codificadas no núcleo celular denominadas factores de ensamblaxe, ou proteínas de ensamblaxe. Estes factores de ensamblaxe contribúen á funcionalidade e estrutura da citocromo c oxidase, e actúan en varios procesos esenciais, como a transcrición e a tradución de subunidades codificadas nas mitocondrias, procesamento de preproteínas e a súa inserción en membranas, e biosíntese e incorporación de cofactores.[12]
Actualmente, identificáronse mutacións en seis factores de ensamblaxe da citocromo c oxidase, que son: SURF1, SCO1, SCO2, COX10, COX15, e LRPPRC. As mutacións nestas proteínas poden orixinar unha alteración da funcionalidade da ensamblaxe do subcomplexo, do transporte de cobre, ou da regulación traducional. Cada mutación nun xene está asociada coa etioloxía dunha doenza específica, e algunhas inflúen en múltiples trastornos. Entre os trastornos que implican unha ensamblaxe disfuncional da citocromo c oxidase por causa de mutacións xenéticas están: a síndrome de Leigh, cardiomiopatía, leucodistrofia, anemia, e xordeira neurosensorial.
Histoquímica[editar | editar a fonte]
A histoquímica da citocromo c oxidase (COX) utilízase para mapar o metablismo cerebral rexional en animais, xa que hai unha relación directa entre a actividade do encima e a actividade neuronal.[13] Este mapado cerebral realizouse en ratos con mutacións espontáneas con enfermidade cerebelar como os ratos mutantes "reeler"[14] e nun modelo transxénico da enfermidade de Alzheimer.[15] Esta técnica tamén se usou para mapar a actividade de aprendizaxe no cerebro animal.[16]
Notas[editar | editar a fonte]
- ↑ Balsa E, Marco R, Perales-Clemente E, Szklarczyk R, Calvo E, Landázuri MO, Enríquez JA (2012). "NDUFA4 is a subunit of complex IV of the mammalian electron transport chain". Cell Metab. 16 (3): 378–86. PMID 22902835. doi:10.1016/j.cmet.2012.07.015.
- ↑ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S (1995). "Structures of metal sites of oxidized bovine heart cytochrome c oxidase at 2.8 A". Science 269 (5227): 1069–74. PMID 7652554. doi:10.1126/science.7652554.
- ↑ Voet, Donald (2010). Biochemistry. New York: J. Wiley & Sons. pp. 865–866. ISBN 0-470-57095-4.
- ↑ Khalimonchuk O, Rödel G (2005). "Biogenesis of cytochrome c oxidase". Mitochondrion 5 (6): 363–88. PMID 16199211. doi:10.1016/j.mito.2005.08.002.
- ↑ Fontanesi F, Soto IC, Horn D, Barrientos A (2006). "Assembly of mitochondrial cytochrome c-oxidase, a complicated and highly regulated cellular process". Am. J. Physiol., Cell Physiol. 291 (6): C1129–47. PMID 16760263. doi:10.1152/ajpcell.00233.2006.
- ↑ Alonso JR, Cardellach F, López S, Casademont J, Miró O (2003). "Carbon monoxide specifically inhibits cytochrome c oxidase of human mitochondrial respiratory chain". Pharmacol. Toxicol. 93 (3): 142–6. PMID 12969439. doi:10.1034/j.1600-0773.2003.930306.x.
- ↑ 7,0 7,1 Sadacharan, S. K., Singh, B., Bowes, T. and Gupta, R. S. (2005). Localization of mitochondrial DNA encoded cytochrome c oxidase subunits I and II in rat pancreatic zymogen granules and pituitary growth hormone granules. Histochem Cell Biol. 124:409-421.
- ↑ Gupta, R. S., Ramachandra, N. B., Bowes, T. and Singh, B. (2008) Unusual cellular disposition of the mitochondrial molecular chaperones Hsp60, Hsp70 and Hsp10. Novartis Found Symp. 291: 59-68.
- ↑ 9,0 9,1 Soltys, B. J. and Gupta, R. S. (1999). Mitochondrial proteins at unexpected cellular locations: export of proteins from mitochondria from an evolutionary perspective. International Review of Cytology. 94:133-196.
- ↑ Soltys , B. J. and Gupta, R.S. (1999) Mitochondrial proteins at unexpected locations: Are they exported?" Trends Biochem. Sci 24: 174-177.
- ↑ Pecina P, Houstková H, Hansíková H, Zeman J, Houstek J (2004). "Genetic defects of cytochrome c oxidase assembly" (PDF). Physiol Res. 53 Suppl 1: S213–23. PMID 15119951.
- ↑ Zee JM, Glerum DM (2006). "Defects in cytochrome oxidase assembly in humans: lessons from yeast". Biochem. Cell Biol. 84 (6): 859–69. PMID 17215873. doi:10.1139/o06-201.
- ↑ Wong-Riley MT. (1989). "Cytochrome oxidase: an endogenous metabolic marker for neuronal activity.". Trends Neurosci. 12 (3): 94–111. PMID 2469224. doi:10.1016/0166-2236(89)90165-3.
- ↑ Strazielle C, Hayzoun K, Derer M, Mariani J, Lalonde R. (2006). "Regional brain variations of cytochrome oxidase activity in Relnrl-orl mutant mice.". J. Neurosci. Res. 83 (5): 821–31. PMID 16511878. doi:10.1002/jnr.20772.
- ↑ Strazielle C, Sturchler-Pierrat C, Staufenbiel M, Lalonde R. (2003). "Regional brain cytochrome oxidase activity in beta-amyloid precursor protein transgenic mice with the Swedish mutation.". Neuroscience 118 (4): 1151–63. PMID 12732258. doi:10.1016/S0306-4522(03)00037-X.
- ↑ Conejo NM, González-Pardo H, Gonzalez-Lima F, Arias JL. (2010). "Spatial learning of the water maze: progression of brain circuits mapped with cytochrome oxidase histochemistry.". Neurobiol. Learn. Mem. 93 (3): 362–71. PMID 19969098. doi:10.1016/j.nlm.2009.12.002.
Véxase tamén[editar | editar a fonte]
Outros artigos[editar | editar a fonte]
- Citocromo c oxidase subunidade I
- Citocromo c oxidase subunidade II
- Citocromo c oxidase subunidade III
- Hemo A
External links[editar | editar a fonte]
- The Cytochrome Oxidase home page na Rice University
- Interactive Molecular model of cytochrome c oxidase (Require MDL Chime)
- UMichOPM families superfamily 4
- Cytochrome-c Oxidase Medical Subject Headings (MeSH) na Biblioteca Nacional de Medicina dos EUA.
